登録情報 データベース : PDB / ID : 1i8jタイトル CRYSTAL STRUCTURE OF PORPHOBILINOGEN SYNTHASE COMPLEXED WITH THE INHIBITOR 4,7-DIOXOSEBACIC ACID PORPHOBILINOGEN SYNTHASE キーワード / / / / 機能・相同性 生物種 Escherichia coli (大腸菌)手法 / / 解像度 : 1.9 Å データ登録者 Kervinen, J. / Jaffe, E.K. / Stauffer, F. / Neier, R. / Wlodawer, A. / Zdanov, A. ジャーナル : Biochemistry / 年 : 2001タイトル : Mechanistic basis for suicide inactivation of porphobilinogen synthase by 4,7-dioxosebacic acid, an inhibitor that shows dramatic species selectivity.著者 : Kervinen, J. / Jaffe, E.K. / Stauffer, F. / Neier, R. / Wlodawer, A. / Zdanov, A. 履歴 登録 2001年3月14日 登録サイト / 処理サイト 改定 1.0 2001年6月20日 Provider / タイプ 改定 1.1 2008年4月27日 Group 改定 1.2 2011年7月13日 Group / Version format compliance改定 1.3 2017年10月4日 Group / カテゴリ / Item / _software.name改定 1.4 2023年8月9日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Refinement description カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_struct_conn_angle / struct_conn / struct_site Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id
すべて表示 表示を減らす Remark 600 HETEROGEN Atoms C4 and C7 of DSB form a Schiff base to NZ of Lys246 and NZ of Lys194. The oxygens ... HETEROGEN Atoms C4 and C7 of DSB form a Schiff base to NZ of Lys246 and NZ of Lys194. The oxygens on the C4 and C7 are eliminated during this reaction.
 データを開く
データを開く 基本情報
基本情報 要素
要素 Delta-aminolevulinic acid dehydratase
Delta-aminolevulinic acid dehydratase  キーワード
キーワード LYASE (リアーゼ) /
LYASE (リアーゼ) /  HEME BIOSYNTHESIS (ヘム) /
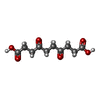
HEME BIOSYNTHESIS (ヘム) /  MAGNESIUM (マグネシウム) / 4 / 7-DIOXOSEBACIC ACID
MAGNESIUM (マグネシウム) / 4 / 7-DIOXOSEBACIC ACID 機能・相同性情報
機能・相同性情報 porphobilinogen synthase /
porphobilinogen synthase /  porphobilinogen synthase activity / protoporphyrinogen IX biosynthetic process / heme biosynthetic process / magnesium ion binding / zinc ion binding /
porphobilinogen synthase activity / protoporphyrinogen IX biosynthetic process / heme biosynthetic process / magnesium ion binding / zinc ion binding /  細胞質基質
細胞質基質
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌) X線回折 /
X線回折 /  分子置換 / 解像度: 1.9 Å
分子置換 / 解像度: 1.9 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Biochemistry / 年: 2001
ジャーナル: Biochemistry / 年: 2001 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1i8j.cif.gz
1i8j.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1i8j.ent.gz
pdb1i8j.ent.gz PDB形式
PDB形式 1i8j.json.gz
1i8j.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/i8/1i8j
https://data.pdbj.org/pub/pdb/validation_reports/i8/1i8j ftp://data.pdbj.org/pub/pdb/validation_reports/i8/1i8j
ftp://data.pdbj.org/pub/pdb/validation_reports/i8/1i8j
 リンク
リンク 集合体
集合体

 要素
要素 Delta-aminolevulinic acid dehydratase / DELTA-AMINOLEVULINIC ACID DEHYDRATASE / 5-AMINOLEVULINIC ACID DEHYDRATASE
Delta-aminolevulinic acid dehydratase / DELTA-AMINOLEVULINIC ACID DEHYDRATASE / 5-AMINOLEVULINIC ACID DEHYDRATASE
 Escherichia coli (大腸菌) / 発現宿主:
Escherichia coli (大腸菌) / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: P0ACB2,
Escherichia coli (大腸菌) / 参照: UniProt: P0ACB2,  porphobilinogen synthase
porphobilinogen synthase 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 回転陽極 / タイプ: RIGAKU RU200 / 波長: 1.5418 Å
回転陽極 / タイプ: RIGAKU RU200 / 波長: 1.5418 Å : 1.5418 Å / 相対比: 1
: 1.5418 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj