+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3312 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | HIV-1 cleaved wild type JR-FL EnvdCT trimer in complex with PGT151 and 10E8 Fabs at 8.8 A resolution | |||||||||
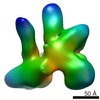
 マップデータ マップデータ | Reconstruction of EnvdCT in complex with 10E8 and PGT151 Fabs. Partial density of a third 10E8 Fab visible due to partial binding occupancy. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  HIV-1 / Env / PGT151 / 10E8 / HIV-1 / Env / PGT151 / 10E8 /  antibody (抗体) / MPER antibody (抗体) / MPER | |||||||||
| 生物種 |    Human Immunodeficiency Virus-1 (ヒト免疫不全ウイルス) / Human Immunodeficiency Virus-1 (ヒト免疫不全ウイルス) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 8.8 Å クライオ電子顕微鏡法 / 解像度: 8.8 Å | |||||||||
 データ登録者 データ登録者 | Lee JH / Ward AB | |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2016 ジャーナル: Science / 年: 2016タイトル: Cryo-EM structure of a native, fully glycosylated, cleaved HIV-1 envelope trimer. 著者: Jeong Hyun Lee / Gabriel Ozorowski / Andrew B Ward /  要旨: The envelope glycoprotein trimer (Env) on the surface of HIV-1 recognizes CD4(+) T cells and mediates viral entry. During this process, Env undergoes substantial conformational rearrangements, making ...The envelope glycoprotein trimer (Env) on the surface of HIV-1 recognizes CD4(+) T cells and mediates viral entry. During this process, Env undergoes substantial conformational rearrangements, making it difficult to study in its native state. Soluble stabilized trimers have provided valuable insights into the Env structure, but they lack the hydrophobic membrane proximal external region (MPER, an important target of broadly neutralizing antibodies), the transmembrane domain, and the cytoplasmic tail. Here we present (i) a cryogenic electron microscopy (cryo-EM) structure of a clade B virus Env, which lacks only the cytoplasmic tail and is stabilized by the broadly neutralizing antibody PGT151, at a resolution of 4.2 angstroms and (ii) a reconstruction of this form of Env in complex with PGT151 and MPER-targeting antibody 10E8 at a resolution of 8.8 angstroms. These structures provide new insights into the wild-type Env structure. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3312.map.gz emd_3312.map.gz | 58.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3312-v30.xml emd-3312-v30.xml emd-3312.xml emd-3312.xml | 15.5 KB 15.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_3312.png emd_3312.png | 565.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3312 http://ftp.pdbj.org/pub/emdb/structures/EMD-3312 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3312 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3312 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3312.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3312.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of EnvdCT in complex with 10E8 and PGT151 Fabs. Partial density of a third 10E8 Fab visible due to partial binding occupancy. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cleaved JR-FL EnvdCT in complex with PGT151 and 10E8 Fabs
| 全体 | 名称: Cleaved JR-FL EnvdCT in complex with PGT151 and 10E8 Fabs |
|---|---|
| 要素 |
|
-超分子 #1000: Cleaved JR-FL EnvdCT in complex with PGT151 and 10E8 Fabs
| 超分子 | 名称: Cleaved JR-FL EnvdCT in complex with PGT151 and 10E8 Fabs タイプ: sample / ID: 1000 集合状態: One trimer bound to one PGT151 and two 10E8 Fabs Number unique components: 3 |
|---|---|
| 分子量 | 理論値: 585 KDa |
-分子 #1: HIV-1 Envelope glycoprotein
| 分子 | 名称: HIV-1 Envelope glycoprotein / タイプ: protein_or_peptide / ID: 1 / Name.synonym: HIV-1 Env 詳細: wild-type JR-FL Env trimer with the cytoplasmic tail truncated コピー数: 1 / 集合状態: Trimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:    Human Immunodeficiency Virus-1 (ヒト免疫不全ウイルス) Human Immunodeficiency Virus-1 (ヒト免疫不全ウイルス)株: JR-FL / 別称: HIV-1 |
| 分子量 | 理論値: 435 KDa |
| 組換発現 | 生物種:  Mammalian (哺乳類) / 組換株: Human / 組換細胞: HEK293F / 組換プラスミド: phCMV3 Mammalian (哺乳類) / 組換株: Human / 組換細胞: HEK293F / 組換プラスミド: phCMV3 |
-分子 #2: Immunoglobulin G PGT151
| 分子 | 名称: Immunoglobulin G PGT151 / タイプ: protein_or_peptide / ID: 2 / Name.synonym: IgG PGT151 / 詳細: PGT151 cleaved into Fab / コピー数: 1 / 集合状態: Heterodimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 50 KDa |
| 組換発現 | 生物種:  Mammalian (哺乳類) / 組換株: Human / 組換細胞: HEK293F Mammalian (哺乳類) / 組換株: Human / 組換細胞: HEK293F |
-分子 #3: Immunoglobulin G 10E8
| 分子 | 名称: Immunoglobulin G 10E8 / タイプ: protein_or_peptide / ID: 3 / Name.synonym: IgG 10E8 / 詳細: 10E8 expressed as Fab / コピー数: 2 / 集合状態: Heterodimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 50 MDa |
| 組換発現 | 生物種:  Mammalian (哺乳類) / 組換株: Human / 組換細胞: HEK293F Mammalian (哺乳類) / 組換株: Human / 組換細胞: HEK293F |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 詳細: 50 mM Tris pH 7.4, 150 mM NaCl, 0.1% DDM, 0.03 mg/mL sodium deoxycholate |
| グリッド | 詳細: 400 mesh C-Flat, CF-2/2-4C, plasma cleaned for 5 seconds |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER 詳細: Samples were treated with biobeads prior to freezing. 手法: Grids were manually plunged at RT. |
- 電子顕微鏡法 #1
電子顕微鏡法 #1
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 22500 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.3 µm / 倍率(公称値): 22500 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.3 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| Microscopy ID | 1 |
| アライメント法 | Legacy - 非点収差: Objective astigmatism corrected at 22,500x magnification. |
| 日付 | 2014年12月16日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) デジタル化 - サンプリング間隔: 5.0 µm / 実像数: 3969 / 平均電子線量: 32.4 e/Å2 詳細: Each full dose image is an aligned stack of frames recorded each using a dose of ~10 e-/Angstrom^2/sec. However, in the Nov session, the microscope experienced FEG instability resulting in ...詳細: Each full dose image is an aligned stack of frames recorded each using a dose of ~10 e-/Angstrom^2/sec. However, in the Nov session, the microscope experienced FEG instability resulting in intensity drop over the course of data collection. |
| Tilt angle min | 0 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 電子顕微鏡法 #2
電子顕微鏡法 #2
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 22500 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.3 µm / 倍率(公称値): 22500 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.3 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| Microscopy ID | 2 |
| アライメント法 | Legacy - 非点収差: Objective astigmatism corrected at 22,500x magnification. |
| 詳細 | Unstable beam intensity over the course of data collection. |
| 日付 | 2014年11月17日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) デジタル化 - サンプリング間隔: 5.0 µm / 実像数: 3969 / 平均電子線量: 28.1 e/Å2 詳細: Each full dose image is an aligned stack of frames recorded each using a dose of ~10 e-/Angstrom^2/sec. However, in the Nov session, the microscope experienced FEG instability resulting in ...詳細: Each full dose image is an aligned stack of frames recorded each using a dose of ~10 e-/Angstrom^2/sec. However, in the Nov session, the microscope experienced FEG instability resulting in intensity drop over the course of data collection. |
| Tilt angle min | 0 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Each micrograph |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 8.8 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Relion / 使用した粒子像数: 15525 |
| 詳細 | The full data set was sorted into multiple 3D classes, which all had various Fab binding stoichiometries of PGT151 and 10E8. This reconstruction is the only sub-population that refined below 10A resolution. |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - #0 - Chain ID: L / Chain - #1 - Chain ID: H / Chain - #2 - Chain ID: P |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | The N-terminal helix of the gp41 peptide and Fab constant regions were removed prior to fitting. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
-原子モデル構築 2
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B / Chain - #2 - Chain ID: C / Chain - #3 - Chain ID: D / Chain - #4 - Chain ID: E / Chain - #5 - Chain ID: F |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー