+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9293 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | EM structure of Bacillus subtilis ribonucleotide reductase inhibited double-helical filament of NrdE alpha subunit with dATP | |||||||||
 マップデータ マップデータ | ASU of Bacillus subtilis ribonucleotide reductase inhibited double-helix filament composed of NrdE alpha subunits with dATP | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  ribonucleotide reductase (リボヌクレオシド二リン酸レダクターゼ) / ribonucleotide reductase (リボヌクレオシド二リン酸レダクターゼ) /  allostery (アロステリック効果) / allostery (アロステリック効果) /  nucleotide metabolism (ヌクレオチド) / nucleotide metabolism (ヌクレオチド) /  filament / filament /  dATP (デオキシアデノシン三リン酸) / dATP (デオキシアデノシン三リン酸) /  ATP (アデノシン三リン酸) / ATP (アデノシン三リン酸) /  OXIDOREDUCTASE (酸化還元酵素) / PROTEIN FIBRIL OXIDOREDUCTASE (酸化還元酵素) / PROTEIN FIBRIL | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 ribonucleoside-diphosphate reductase complex / ribonucleoside-diphosphate reductase complex /  リボヌクレオシド二リン酸レダクターゼ / ribonucleoside-diphosphate reductase activity, thioredoxin disulfide as acceptor / deoxyribonucleotide biosynthetic process / リボヌクレオシド二リン酸レダクターゼ / ribonucleoside-diphosphate reductase activity, thioredoxin disulfide as acceptor / deoxyribonucleotide biosynthetic process /  DNA複製 / DNA複製 /  ATP binding ATP binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Bacillus subtilis (枯草菌) Bacillus subtilis (枯草菌) | |||||||||
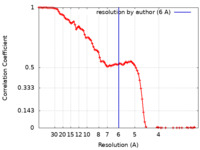
| 手法 | らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 6.0 Å クライオ電子顕微鏡法 / 解像度: 6.0 Å | |||||||||
 データ登録者 データ登録者 | Thomas WC / Bacik JP | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: Convergent allostery in ribonucleotide reductase. 著者: William C Thomas / F Phil Brooks / Audrey A Burnim / John-Paul Bacik / JoAnne Stubbe / Jason T Kaelber / James Z Chen / Nozomi Ando /  要旨: Ribonucleotide reductases (RNRs) use a conserved radical-based mechanism to catalyze the conversion of ribonucleotides to deoxyribonucleotides. Within the RNR family, class Ib RNRs are notable for ...Ribonucleotide reductases (RNRs) use a conserved radical-based mechanism to catalyze the conversion of ribonucleotides to deoxyribonucleotides. Within the RNR family, class Ib RNRs are notable for being largely restricted to bacteria, including many pathogens, and for lacking an evolutionarily mobile ATP-cone domain that allosterically controls overall activity. In this study, we report the emergence of a distinct and unexpected mechanism of activity regulation in the sole RNR of the model organism Bacillus subtilis. Using a hypothesis-driven structural approach that combines the strengths of small-angle X-ray scattering (SAXS), crystallography, and cryo-electron microscopy (cryo-EM), we describe the reversible interconversion of six unique structures, including a flexible active tetramer and two inhibited helical filaments. These structures reveal the conformational gymnastics necessary for RNR activity and the molecular basis for its control via an evolutionarily convergent form of allostery. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9293.map.gz emd_9293.map.gz | 14.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9293-v30.xml emd-9293-v30.xml emd-9293.xml emd-9293.xml | 20.1 KB 20.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9293_fsc.xml emd_9293_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9293.png emd_9293.png | 44 KB | ||
| Filedesc metadata |  emd-9293.cif.gz emd-9293.cif.gz | 7 KB | ||
| その他 |  emd_9293_half_map_1.map.gz emd_9293_half_map_1.map.gz emd_9293_half_map_2.map.gz emd_9293_half_map_2.map.gz | 59.5 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9293 http://ftp.pdbj.org/pub/emdb/structures/EMD-9293 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9293 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9293 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9293.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9293.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ASU of Bacillus subtilis ribonucleotide reductase inhibited double-helix filament composed of NrdE alpha subunits with dATP | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.505 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Half-set map #1
| ファイル | emd_9293_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-set map #1 | ||||||||||||
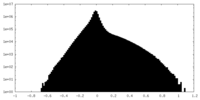
| 投影像・断面図 |
| ||||||||||||
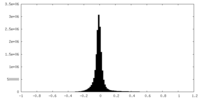
| 密度ヒストグラム |
-ハーフマップ: Half-set map #2
| ファイル | emd_9293_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-set map #2 | ||||||||||||
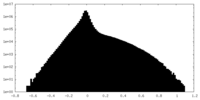
| 投影像・断面図 |
| ||||||||||||
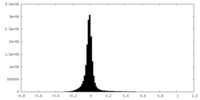
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Inhibited filament of ribonucleoside-diphosphate reductase compos...
| 全体 | 名称: Inhibited filament of ribonucleoside-diphosphate reductase composed of NrdE alpha subunit |
|---|---|
| 要素 |
|
-超分子 #1: Inhibited filament of ribonucleoside-diphosphate reductase compos...
| 超分子 | 名称: Inhibited filament of ribonucleoside-diphosphate reductase composed of NrdE alpha subunit タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: The filament is a double helix. Each helix is composed of NrdE subunits dimerizing at alternating canonical and non-canonical interfaces. |
|---|---|
| 由来(天然) | 生物種:   Bacillus subtilis (枯草菌) Bacillus subtilis (枯草菌) |
-分子 #1: Ribonucleoside-diphosphate reductase
| 分子 | 名称: Ribonucleoside-diphosphate reductase / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO EC番号:  リボヌクレオシド二リン酸レダクターゼ リボヌクレオシド二リン酸レダクターゼ |
|---|---|
| 由来(天然) | 生物種:   Bacillus subtilis (枯草菌) Bacillus subtilis (枯草菌) |
| 分子量 | 理論値: 80.791469 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MSQNQVPKWI QLNNEIMIQK DGKFQFDKDK EAVHSYFVDY INQNTVFFHN LKEKLDYLVE NQYYEEEFLS LYSFEDIKEV FKTAYAKKF RFPSFMSAFK FYNDYALKTN DKKKILERYE DRISIVALFF ANGDTEKAKE YVNLMINQEY QPSTPTFLNA G RKRRGELV ...文字列: MSQNQVPKWI QLNNEIMIQK DGKFQFDKDK EAVHSYFVDY INQNTVFFHN LKEKLDYLVE NQYYEEEFLS LYSFEDIKEV FKTAYAKKF RFPSFMSAFK FYNDYALKTN DKKKILERYE DRISIVALFF ANGDTEKAKE YVNLMINQEY QPSTPTFLNA G RKRRGELV SCFLLEVNDS LNDISRAIDI SMQLSKLGGG VSLNLSKLRA KGEAIKDVEN ATKGVVGVMK LLDNAFRYAD QM GQRQGSG AAYLNIFHRD INDFLDTKKI SADEDVRVKT LSIGVVIPDK FVELAREDKA AYVFYPHTIY KEYGQHMDEM DMN EMYDKF VDNPRVKKEK INPRKLLEKL AMLRSESGYP YIMFQDNVNK VHANNHISKV KFSNLCSEVL QASQVSSYTD YDEE DEIGL DISCNLGSLN ILNVMEHKSI EKTVKLATDS LTHVSETTDI RNAPAVRRAN KAMKSIGLGA MNLHGYLAQN GIAYE SPEA RDFANTFFMM VNFYSIQRSA EIAKEKGETF DQYEGSTYAT GEYFDKYVST DFSPKYEKIA NLFEGMHIPT TEDWKK LKA FVAEHGMYHS YRLCIAPTGS ISYVQSSTAS VMPIMERIEE RTYGNSKTYY PMPGLASNNW FFYKEAYDMD MFKVVDM IA TIQQHIDQGI SFTLFLKDTM TTRDLNRIDL YAHHRGIKTI YYARTKDTGQ DSCLSCVV UniProtKB:  リボヌクレオシド二リン酸レダクターゼ リボヌクレオシド二リン酸レダクターゼ |
-分子 #2: 2'-DEOXYADENOSINE 5'-TRIPHOSPHATE
| 分子 | 名称: 2'-DEOXYADENOSINE 5'-TRIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 8 / 式: DTP |
|---|---|
| 分子量 | 理論値: 491.182 Da |
| Chemical component information |  ChemComp-DTP: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 0.81 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.6 構成要素:
詳細: Glycerol in original buffer was diluted to < 0.25% w/v. | |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 300 K / 装置: FEI VITROBOT MARK IV / 詳細: 3.5 seconds blotting. | |||||||||||||||
| 詳細 | Samples of the dATP-induced NrdE filament were produced by incubating 40 uM holo-NrdE with 100 uM dATP and 1 mM CDP in assay buffer. The mixture was then diluted to 10 uM NrdE in the same nucleotide-containing buffer. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 1.2 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.2 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 特殊光学系 | 球面収差補正装置: None / 色収差補正装置: None |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4000 pixel / デジタル化 - サイズ - 縦: 4000 pixel / デジタル化 - 画像ごとのフレーム数: 2-90 / 撮影したグリッド数: 2 / 実像数: 500 / 平均露光時間: 20.0 sec. / 平均電子線量: 20.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER / 当てはまり具合の基準: Corellation coefficient | ||||||
| 得られたモデル |  PDB-6myx: |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X