+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7bgu | ||||||
|---|---|---|---|---|---|---|---|
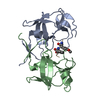
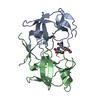
| タイトル | Mason-Pfizer Monkey Virus Protease mutant C7A/D26N/C106A in complex with peptidomimetic inhibitor | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | HYDROLASE / Mason-Pfizer Monkey Virus / M-PMV / retropepsin / active site inhibitor / active site mutant | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報dUTP diphosphatase / dUTP diphosphatase activity / ribonuclease H / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; アスパラギン酸プロテアーゼ / DNA integration / viral genome integration into host DNA / RNA-directed DNA polymerase / establishment of integrated proviral latency / RNA stem-loop binding / RNA-directed DNA polymerase activity ...dUTP diphosphatase / dUTP diphosphatase activity / ribonuclease H / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; アスパラギン酸プロテアーゼ / DNA integration / viral genome integration into host DNA / RNA-directed DNA polymerase / establishment of integrated proviral latency / RNA stem-loop binding / RNA-directed DNA polymerase activity / RNA-DNA hybrid ribonuclease activity / 転移酵素; リンを含む基を移すもの; 核酸を移すもの / viral nucleocapsid / DNA recombination / DNA-directed DNA polymerase / aspartic-type endopeptidase activity / structural constituent of virion / 加水分解酵素; エステル加水分解酵素 / DNA-directed DNA polymerase activity / viral translational frameshifting / symbiont entry into host cell / proteolysis / DNA binding / zinc ion binding 類似検索 - 分子機能 | ||||||
| 生物種 |  Mason-Pfizer monkey virus (ウイルス) Mason-Pfizer monkey virus (ウイルス)synthetic construct (人工物) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.433 Å 分子置換 / 解像度: 2.433 Å | ||||||
 データ登録者 データ登録者 | Wosicki, S. / Gilski, M. / Kazmierczyk, M. / Jaskolski, M. / Zabranska, H. / Pichova, I. | ||||||
| 資金援助 |  チェコ, 1件 チェコ, 1件
| ||||||
 引用 引用 |  ジャーナル: Protein Sci. / 年: 2021 ジャーナル: Protein Sci. / 年: 2021タイトル: Crystal structures of inhibitor complexes of M-PMV protease with visible flap loops. 著者: Wosicki, S. / Kazmierczyk, M. / Gilski, M. / Zabranska, H. / Pichova, I. / Jaskolski, M. #1:  ジャーナル: Acta Crystallogr D Struct Biol / 年: 2019 ジャーナル: Acta Crystallogr D Struct Biol / 年: 2019タイトル: Comparison of a retroviral protease in monomeric and dimeric states. 著者: Wosicki, S. / Gilski, M. / Zabranska, H. / Pichova, I. / Jaskolski, M. #2: ジャーナル: Acta Crystallogr. D Biol. Crystallogr. / 年: 2011 タイトル: High-resolution structure of a retroviral protease folded as a monomer. 著者: Gilski, M. / Kazmierczyk, M. / Krzywda, S. / Zabranska, H. / Cooper, S. / Popovic, Z. / Khatib, F. / DiMaio, F. / Thompson, J. / Baker, D. / Pichova, I. / Jaskolski, M. #3: ジャーナル: Nature / 年: 1989 タイトル: Crystal structure of a retroviral protease proves relationship to aspartic protease family. 著者: Miller, M. / Jaskolski, M. / Rao, J.K. / Leis, J. / Wlodawer, A. #4:  ジャーナル: Science / 年: 1989 ジャーナル: Science / 年: 1989タイトル: Conserved folding in retroviral proteases: crystal structure of a synthetic HIV-1 protease. 著者: Wlodawer, A. / Miller, M. / Jaskolski, M. / Sathyanarayana, B.K. / Baldwin, E. / Weber, I.T. / Selk, L.M. / Clawson, L. / Schneider, J. / Kent, S.B. #5:  ジャーナル: Nat. Struct. Mol. Biol. / 年: 2011 ジャーナル: Nat. Struct. Mol. Biol. / 年: 2011タイトル: Crystal structure of a monomeric retroviral protease solved by protein folding game players. 著者: Khatib, F. / DiMaio, F. / Cooper, S. / Kazmierczyk, M. / Gilski, M. / Krzywda, S. / Zabranska, H. / Pichova, I. / Thompson, J. / Popovic, Z. / Jaskolski, M. / Baker, D. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7bgu.cif.gz 7bgu.cif.gz | 198.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7bgu.ent.gz pdb7bgu.ent.gz | 158.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7bgu.json.gz 7bgu.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7bgu_validation.pdf.gz 7bgu_validation.pdf.gz | 475.8 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7bgu_full_validation.pdf.gz 7bgu_full_validation.pdf.gz | 480.8 KB | 表示 | |
| XML形式データ |  7bgu_validation.xml.gz 7bgu_validation.xml.gz | 20.5 KB | 表示 | |
| CIF形式データ |  7bgu_validation.cif.gz 7bgu_validation.cif.gz | 28 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bg/7bgu https://data.pdbj.org/pub/pdb/validation_reports/bg/7bgu ftp://data.pdbj.org/pub/pdb/validation_reports/bg/7bgu ftp://data.pdbj.org/pub/pdb/validation_reports/bg/7bgu | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7bgtC  6s1vS S: 精密化の開始モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 実験データセット #1 | データ参照:  10.18150/C9DYSH / データの種類: diffraction image data 10.18150/C9DYSH / データの種類: diffraction image data |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域:
|
 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj

