+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5t47 | ||||||
|---|---|---|---|---|---|---|---|
| Title | Crystal structure of the D. melanogaster eIF4E-eIF4G complex | ||||||
 Components Components |
| ||||||
 Keywords Keywords | TRANSLATION / GENE REGULATION / CAP BINDING PROTEIN / 4E-BINDING PROTEIN / TRANSLATION INITIATION / eIF4F | ||||||
| Function / homology |  Function and homology information Function and homology informationTOR signaling pathway / Activation of the mRNA upon binding of the cap-binding complex and eIFs, and subsequent binding to 43S / Transport of the SLBP independent Mature mRNA / Transport of the SLBP Dependant Mature mRNA / Transport of Mature mRNA Derived from an Intronless Transcript / : / L13a-mediated translational silencing of Ceruloplasmin expression / mTORC1-mediated signalling / Translation initiation complex formation / Ribosomal scanning and start codon recognition ...TOR signaling pathway / Activation of the mRNA upon binding of the cap-binding complex and eIFs, and subsequent binding to 43S / Transport of the SLBP independent Mature mRNA / Transport of the SLBP Dependant Mature mRNA / Transport of Mature mRNA Derived from an Intronless Transcript / : / L13a-mediated translational silencing of Ceruloplasmin expression / mTORC1-mediated signalling / Translation initiation complex formation / Ribosomal scanning and start codon recognition / muscle cell postsynaptic specialization / RNA metabolic process / neuronal ribonucleoprotein granule / eukaryotic initiation factor 4G binding / eukaryotic initiation factor 4E binding / RNA cap binding / eukaryotic translation initiation factor 4F complex / RNA 7-methylguanosine cap binding / translation initiation factor activity / neuromuscular junction / P-body / translational initiation / mitotic cell cycle / regulation of translation / nuclear body / translation / RNA binding / cytosol / cytoplasm Similarity search - Function | ||||||
| Biological species |  | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.2 Å MOLECULAR REPLACEMENT / Resolution: 2.2 Å | ||||||
 Authors Authors | Gruener, S. / Peter, D. / Weber, R. / Wohlbold, L. / Chung, M.-Y. / Weichenrieder, O. / Valkov, E. / Igreja, C. / Izaurralde, E. | ||||||
 Citation Citation |  Journal: Mol.Cell / Year: 2016 Journal: Mol.Cell / Year: 2016Title: The Structures of eIF4E-eIF4G Complexes Reveal an Extended Interface to Regulate Translation Initiation. Authors: Gruner, S. / Peter, D. / Weber, R. / Wohlbold, L. / Chung, M.Y. / Weichenrieder, O. / Valkov, E. / Igreja, C. / Izaurralde, E. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5t47.cif.gz 5t47.cif.gz | 273 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5t47.ent.gz pdb5t47.ent.gz | 227.6 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5t47.json.gz 5t47.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/t4/5t47 https://data.pdbj.org/pub/pdb/validation_reports/t4/5t47 ftp://data.pdbj.org/pub/pdb/validation_reports/t4/5t47 ftp://data.pdbj.org/pub/pdb/validation_reports/t4/5t47 | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5t46C  5t48C  4ue8S S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 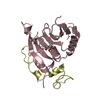
| ||||||||
| Unit cell |
|
- Components
Components
| #1: Protein | Mass: 21249.037 Da / Num. of mol.: 2 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   #2: Protein | Mass: 7410.389 Da / Num. of mol.: 2 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   #3: Chemical | #4: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 1.9 Å3/Da / Density % sol: 36 % |
|---|---|
| Crystal grow | Temperature: 291 K / Method: vapor diffusion, hanging drop / pH: 8.25 / Details: 0.1 M TrisHCl pH 8.25, 0.2 M MgCl2, 32% PEG4000 |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SLS SLS  / Beamline: X10SA / Wavelength: 0.99992 Å / Beamline: X10SA / Wavelength: 0.99992 Å |
| Detector | Type: DECTRIS PILATUS 6M / Detector: PIXEL / Date: Apr 19, 2015 / Details: DYNAMICALLY BENDABLE MIRRORS |
| Radiation | Monochromator: Si(111) / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.99992 Å / Relative weight: 1 |
| Reflection | Resolution: 2.2→41.42 Å / Num. obs: 20740 / % possible obs: 96.6 % / Observed criterion σ(I): -3 / Redundancy: 3 % / Biso Wilson estimate: 27.65 Å2 / CC1/2: 0.992 / Rsym value: 0.121 / Net I/σ(I): 6.2 |
| Reflection shell | Resolution: 2.2→2.27 Å / Redundancy: 2.5 % / Rmerge(I) obs: 0.609 / Mean I/σ(I) obs: 1.7 / CC1/2: 0.683 / Rsym value: 0.609 / % possible all: 91 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 4UE8 Resolution: 2.2→41.415 Å / SU ML: 0.28 / Cross valid method: FREE R-VALUE / σ(F): 1.07 / Phase error: 29 Details: TORSIONAL NCS RESTRAINTS WERE APPLIED RELATING CHAINS A, B TO CHAINS C, D. HYDROGENS WERE REFINED IN THE RIDING POSITIONS.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 42.45 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.2→41.415 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller













 PDBj
PDBj




