| ソフトウェア | | 名称 | バージョン | 分類 |
|---|
| PHENIX | (PHENIX.REFINE)| 精密化 | | ESRF | AUTOPROCESS| データ削減 | | Aimless | | データスケーリング | | PHASER | | 位相決定 | | |
|
|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換
開始モデル: PDB ENTRY 4B5R
解像度: 1.71→41.134 Å / SU ML: 0.27 / σ(F): 1.33 / 位相誤差: 28.03 / 立体化学のターゲット値: ML
| Rfactor | 反射数 | %反射 |
|---|
| Rfree | 0.2405 | 2822 | 4.9 % |
|---|
| Rwork | 0.2009 | - | - |
|---|
| obs | 0.2028 | 31391 | 99.48 % |
|---|
|
|---|
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL |
|---|
| 原子変位パラメータ | Biso mean: 41.4 Å2 |
|---|
| 精密化ステップ | サイクル: LAST / 解像度: 1.71→41.134 Å
| タンパク質 | 核酸 | リガンド | 溶媒 | 全体 |
|---|
| 原子数 | 0 | 2037 | 55 | 183 | 2275 |
|---|
|
|---|
| 拘束条件 | | Refine-ID | タイプ | Dev ideal | 数 |
|---|
| X-RAY DIFFRACTION | f_bond_d| 0.004 | 2390 | | X-RAY DIFFRACTION | f_angle_d| 1.122 | 3733 | | X-RAY DIFFRACTION | f_dihedral_angle_d| 15.628 | 1183 | | X-RAY DIFFRACTION | f_chiral_restr| 0.044 | 489 | | X-RAY DIFFRACTION | f_plane_restr| 0.008 | 99 | | | | | |
|
|---|
| LS精密化 シェル | | 解像度 (Å) | Rfactor Rfree | Num. reflection Rfree | Rfactor Rwork | Num. reflection Rwork | Refine-ID | % reflection obs (%) |
|---|
| 1.7101-1.7396 | 0.4039 | 133 | 0.3727 | 2536 | X-RAY DIFFRACTION | 92 | | 1.7396-1.7712 | 0.3453 | 138 | 0.3471 | 2817 | X-RAY DIFFRACTION | 100 | | 1.7712-1.8053 | 0.3098 | 134 | 0.3163 | 2763 | X-RAY DIFFRACTION | 100 | | 1.8053-1.8421 | 0.3247 | 140 | 0.3004 | 2770 | X-RAY DIFFRACTION | 100 | | 1.8421-1.8822 | 0.3127 | 128 | 0.2864 | 2808 | X-RAY DIFFRACTION | 100 | | 1.8822-1.926 | 0.3133 | 179 | 0.2779 | 2748 | X-RAY DIFFRACTION | 100 | | 1.926-1.9741 | 0.314 | 137 | 0.2865 | 2739 | X-RAY DIFFRACTION | 100 | | 1.9741-2.0275 | 0.34 | 133 | 0.2719 | 2818 | X-RAY DIFFRACTION | 100 | | 2.0275-2.0871 | 0.3119 | 127 | 0.2687 | 2803 | X-RAY DIFFRACTION | 100 | | 2.0871-2.1545 | 0.3158 | 142 | 0.2471 | 2768 | X-RAY DIFFRACTION | 100 | | 2.1545-2.2315 | 0.2812 | 114 | 0.2451 | 2787 | X-RAY DIFFRACTION | 100 | | 2.2315-2.3209 | 0.3197 | 149 | 0.2352 | 2806 | X-RAY DIFFRACTION | 100 | | 2.3209-2.4265 | 0.3102 | 142 | 0.2237 | 2757 | X-RAY DIFFRACTION | 100 | | 2.4265-2.5544 | 0.2645 | 173 | 0.2163 | 2743 | X-RAY DIFFRACTION | 100 | | 2.5544-2.7144 | 0.2443 | 154 | 0.2181 | 2758 | X-RAY DIFFRACTION | 100 | | 2.7144-2.9239 | 0.2557 | 145 | 0.2124 | 2784 | X-RAY DIFFRACTION | 100 | | 2.9239-3.2181 | 0.2327 | 155 | 0.1763 | 2763 | X-RAY DIFFRACTION | 100 | | 3.2181-3.6835 | 0.2024 | 165 | 0.1607 | 2748 | X-RAY DIFFRACTION | 100 | | 3.6835-4.6398 | 0.1647 | 112 | 0.1497 | 2827 | X-RAY DIFFRACTION | 100 | | 4.6398-41.1459 | 0.2144 | 122 | 0.1882 | 2749 | X-RAY DIFFRACTION | 99 |
|
|---|
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION | ID | L11 (°2) | L12 (°2) | L13 (°2) | L22 (°2) | L23 (°2) | L33 (°2) | S11 (Å °) | S12 (Å °) | S13 (Å °) | S21 (Å °) | S22 (Å °) | S23 (Å °) | S31 (Å °) | S32 (Å °) | S33 (Å °) | T11 (Å2) | T12 (Å2) | T13 (Å2) | T22 (Å2) | T23 (Å2) | T33 (Å2) | Origin x (Å) | Origin y (Å) | Origin z (Å) |
|---|
| 1 | 4.1535 | 1.3178 | -4.0389 | 6.5639 | -1.6545 | 4.0675 | -0.7278 | 1.7677 | 0.0445 | -0.7086 | 0.6281 | -0.3366 | 0.9399 | -0.0644 | 0.1166 | 0.4525 | -0.0717 | -0.0082 | 0.5866 | -0.024 | 0.4335 | -0.667 | -13.902 | 36.1422 | | 2 | 4.9859 | -1.0393 | 1.5971 | 1.4397 | 0.0761 | 3.8655 | 0.0681 | 0.0041 | -0.1204 | -0.009 | -0.1531 | 0.3812 | -0.1006 | 0.0416 | 0.0669 | 0.3252 | -0.0707 | 0.0474 | 0.3204 | -0.0438 | 0.4553 | -16.0282 | -12.5131 | 34.3435 | | 3 | 3.4892 | -4.415 | 3.0717 | 5.941 | -3.1094 | 7.2499 | 0.1357 | 0.6618 | -0.6128 | -0.6976 | 0.3342 | 0.456 | 0.9339 | -0.7107 | -0.5816 | 0.3066 | -0.0186 | -0.0083 | 0.5523 | -0.0303 | 0.474 | -23.5513 | -6.0362 | 20.114 | | 4 | 0.1361 | 0.8411 | 0.279 | 6.8518 | -0.9635 | 3.8033 | -0.1836 | 1.0417 | 1.3225 | -0.7684 | 0.5901 | 1.2738 | 0.5142 | -1.3166 | -0.4656 | 0.6246 | 0.133 | 0.0503 | 1.3169 | 0.3511 | 0.6133 | -15.2745 | 9.5783 | 9.7289 | | 5 | 3.2955 | -1.7124 | -1.3344 | 6.2972 | 4.0341 | 4.471 | 0.3972 | 0.2893 | 0.6535 | -0.5753 | 0.0669 | -0.4437 | -0.5982 | 0.2099 | -0.4744 | 0.3255 | -0.0518 | 0.1195 | 0.4264 | 0.0119 | 0.4327 | 0.029 | 7.3895 | 8.6937 | | 6 | 3.549 | 3.9473 | -1.6528 | 5.5126 | -0.4291 | 2.2157 | -0.1047 | 0.5574 | 0.2532 | 0.3482 | 0.2926 | 0.0673 | 0.2466 | 0.0244 | -0.1196 | 0.3734 | 0.0472 | -0.0315 | 0.4188 | 0.021 | 0.3009 | -2.943 | -8.9771 | 13.726 | | 7 | 5.5357 | 1.0016 | 3.209 | 8.2081 | -0.5444 | 5.7537 | 0.2651 | 0.9215 | -0.359 | -0.681 | 0.3527 | -1.3118 | 0.6338 | 1.1959 | -0.8382 | 0.3339 | 0.0562 | 0.073 | 0.4882 | -0.1034 | 0.4252 | -3.3192 | -20.36 | 13.3496 | | 8 | 2.9864 | 2.8044 | -1.8604 | 2.7743 | -2.184 | 2.0044 | 0.0672 | 0.285 | 0.0305 | -0.4974 | -0.1201 | -0.005 | 0.1732 | -0.258 | 0.1329 | 0.3815 | -0.0624 | 0.0074 | 0.4372 | 0.0303 | 0.2597 | -6.7871 | -6.7934 | 3.7561 | | 9 | 2.8745 | -1.8313 | -2.7665 | 6.019 | 0.0748 | 3.1653 | 0.0528 | 0.1769 | 0.0782 | -0.1352 | 0.0111 | 0.2384 | 0.1044 | -0.8158 | -0.0552 | 0.1809 | 0.0136 | 0.0204 | 0.4651 | 0.0492 | 0.291 | -5.4751 | 3.1805 | 8.2105 | | 10 | 6.4609 | 2.1816 | -4.366 | 1.5773 | -2.1572 | 3.5943 | 0.3717 | 0.4072 | 0.9788 | 0.6701 | 0.3096 | 0.6421 | -0.6409 | -0.6131 | -0.7213 | 0.3218 | 0.0951 | 0.1156 | 0.4408 | 0.0524 | 0.5395 | -16.363 | 8.3192 | 21.4615 | | 11 | 2.1981 | -3.606 | -0.957 | 9.6865 | -3.9402 | 8.3425 | -0.2892 | 0.1272 | 0.5163 | 0.8303 | 0.2923 | -0.0705 | -0.5806 | -0.9042 | -0.0015 | 0.328 | 0.0733 | 0.0875 | 0.3707 | -0.0429 | 0.3721 | -18.3133 | 1.568 | 36.3793 | | 12 | 3.835 | -2.2321 | -1.7007 | 3.8589 | 2.2729 | 7.3102 | -0.2933 | -0.0504 | 0.0256 | 1.275 | 0.3686 | 0.3657 | -0.1497 | -0.8587 | -0.0466 | 0.7431 | 0.1127 | 0.1715 | 0.4015 | 0.0073 | 0.4975 | -19.5149 | 11.2736 | 38.5249 | | 13 | 4.8396 | -1.4466 | -1.096 | 4.7496 | 0.9583 | 6.9468 | 0.3413 | 0.6298 | 0.5981 | -0.2945 | -0.021 | -0.5849 | 0.1158 | 0.4256 | -0.2402 | 0.2971 | 0.0562 | 0.0842 | 0.4235 | 0.0261 | 0.3758 | -12.485 | 1.1328 | 24.7284 | | 14 | 4.9935 | 0.4494 | -2.7008 | 6.5687 | -6.5246 | 9.3527 | 0.2664 | 0.2839 | -0.5398 | -0.2075 | 0.192 | 0.8754 | 0.1876 | -1.071 | -0.5128 | 0.3716 | -0.1519 | -0.0318 | 0.5071 | -0.0115 | 0.3543 | -16.2994 | -10.6797 | 15.2367 | | 15 | 0.8977 | 1.5341 | -0.8113 | 2.6115 | -1.8725 | 3.6672 | -0.0842 | -0.0372 | -0.2667 | -0.0757 | 0.0412 | -0.0275 | -0.0669 | 0.3689 | 0.0736 | 0.2732 | -0.0379 | 0.0407 | 0.4293 | -0.0354 | 0.3361 | -11.0565 | -22.4372 | 17.8727 | | 16 | 5.4741 | 5.742 | -0.0736 | 8.5811 | 2.618 | 5.1307 | 0.4133 | -0.6115 | -0.3042 | 0.0528 | -0.4216 | -0.128 | 0.2345 | -0.0363 | -0.1008 | 0.3214 | 0.0492 | -0.01 | 0.3633 | 0.0401 | 0.3034 | -19.1337 | -35.7933 | 23.1547 | | 17 | 7.9904 | -2.7017 | -5.2097 | 4.657 | 3.5895 | 8.7244 | -0.1326 | 0.0808 | -0.1746 | 0.4141 | 0.1951 | -0.1365 | 0.4842 | 0.7146 | 0.0151 | 0.3397 | 0.0481 | -0.0085 | 0.3892 | 0.0222 | 0.2882 | -19.7359 | -35.9165 | 12.3351 | | 18 | 8.8919 | 5.4533 | 2.6633 | 6.8393 | 3.6984 | 2.203 | 0.3079 | -0.4784 | 0.5572 | 0.3601 | -0.2121 | -0.0822 | -0.3726 | 0.4127 | -0.0729 | 0.3617 | -0.0711 | 0.0311 | 0.4074 | -0.0127 | 0.3567 | -16.8086 | -24.9474 | 26.9198 | | 19 | 0.9378 | -1.0175 | -0.1407 | 6.4459 | 3.0671 | 4.3997 | -0.0582 | -0.0483 | 0.2334 | 0.3762 | 0.0803 | -0.3122 | 0.1097 | 0.2072 | 0.0502 | 0.2422 | -0.0246 | -0.0221 | 0.3099 | -0.0254 | 0.298 | -8.8525 | -10.0059 | 30.6032 | | 20 | 5.9321 | -3.1159 | 1.8056 | 6.7948 | -1.0971 | 5.0678 | -0.2116 | -0.1434 | 1.0616 | -0.1003 | 0.1162 | -0.294 | -0.1789 | 0.4025 | 0.0998 | 0.3942 | -0.0909 | -0.017 | 0.3506 | 0.0031 | 0.4423 | 0.2667 | -7.6852 | 42.2619 |
|
|---|
| 精密化 TLSグループ | | ID | Refine-ID | Refine TLS-ID | Selection details |
|---|
| 1 | X-RAY DIFFRACTION | 1 | (CHAIN A AND RESID 1:4)| 2 | X-RAY DIFFRACTION | 2 | (CHAIN A AND RESID 5:8)| 3 | X-RAY DIFFRACTION | 3 | (CHAIN A AND RESID 9:12)| 4 | X-RAY DIFFRACTION | 4 | (CHAIN A AND RESID 13:16)| 5 | X-RAY DIFFRACTION | 5 | (CHAIN A AND RESID 17:20)| 6 | X-RAY DIFFRACTION | 6 | (CHAIN A AND RESID 21:24)| 7 | X-RAY DIFFRACTION | 7 | (CHAIN A AND RESID 25:28)| 8 | X-RAY DIFFRACTION | 8 | (CHAIN A AND RESID 29:33)| 9 | X-RAY DIFFRACTION | 9 | (CHAIN A AND RESID 34:39)| 10 | X-RAY DIFFRACTION | 10 | (CHAIN A AND RESID 40:45)| 11 | X-RAY DIFFRACTION | 11 | (CHAIN A AND RESID 46:49)| 12 | X-RAY DIFFRACTION | 12 | (CHAIN A AND RESID 50:56)| 13 | X-RAY DIFFRACTION | 13 | (CHAIN A AND RESID 57:60)| 14 | X-RAY DIFFRACTION | 14 | (CHAIN A AND RESID 61:64)| 15 | X-RAY DIFFRACTION | 15 | (CHAIN A AND RESID 65:69)| 16 | X-RAY DIFFRACTION | 16 | (CHAIN A AND RESID 70:73)| 17 | X-RAY DIFFRACTION | 17 | (CHAIN A AND RESID 74:80)| 18 | X-RAY DIFFRACTION | 18 | (CHAIN A AND | | | | | | | | | | | | | | | | | |
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 THERMOANAEROBACTER TENGCONGENSIS (バクテリア)
THERMOANAEROBACTER TENGCONGENSIS (バクテリア) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.71 Å
分子置換 / 解像度: 1.71 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nucleic Acids Res. / 年: 2016
ジャーナル: Nucleic Acids Res. / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5fjc.cif.gz
5fjc.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5fjc.ent.gz
pdb5fjc.ent.gz PDB形式
PDB形式 5fjc.json.gz
5fjc.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5fjc_validation.pdf.gz
5fjc_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5fjc_full_validation.pdf.gz
5fjc_full_validation.pdf.gz 5fjc_validation.xml.gz
5fjc_validation.xml.gz 5fjc_validation.cif.gz
5fjc_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/fj/5fjc
https://data.pdbj.org/pub/pdb/validation_reports/fj/5fjc ftp://data.pdbj.org/pub/pdb/validation_reports/fj/5fjc
ftp://data.pdbj.org/pub/pdb/validation_reports/fj/5fjc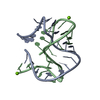













 リンク
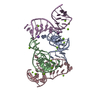
リンク 集合体
集合体
 要素
要素 THERMOANAEROBACTER TENGCONGENSIS (バクテリア)
THERMOANAEROBACTER TENGCONGENSIS (バクテリア)










 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID29 / 波長: 0.9792
/ ビームライン: ID29 / 波長: 0.9792  解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj


































