| 登録情報 | データベース: PDB / ID: 4r30
|
|---|
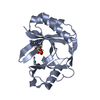
| タイトル | Structure of human laforin dual specificity phosphatase domain |
|---|
 要素 要素 | Laforin |
|---|
 キーワード キーワード | HYDROLASE / Dual specificity phosphatase / Glucan phosphatase / Malin / glycogen |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
negative regulation of phosphatase activity / carbohydrate phosphatase activity / negative regulation of dephosphorylation / glycogen binding / regulation of protein import into nucleus / carbohydrate phosphorylation / alpha-1,4-glucan glucosyltransferase (UDP-glucose donor) activity / L-glutamate transmembrane transport / starch binding / histone H2AXS139 phosphatase activity ...negative regulation of phosphatase activity / carbohydrate phosphatase activity / negative regulation of dephosphorylation / glycogen binding / regulation of protein import into nucleus / carbohydrate phosphorylation / alpha-1,4-glucan glucosyltransferase (UDP-glucose donor) activity / L-glutamate transmembrane transport / starch binding / histone H2AXS139 phosphatase activity / RNA polymerase II CTD heptapeptide repeat Y1 phosphatase activity / RNA polymerase II CTD heptapeptide repeat T4 phosphatase activity / RNA polymerase II CTD heptapeptide repeat S2 phosphatase activity / RNA polymerase II CTD heptapeptide repeat S7 phosphatase activity / MAP kinase serine/threonine phosphatase activity / myosin phosphatase activity / habituation / 加水分解酵素; エステル加水分解酵素; 1価のリン酸エステル加水分解酵素 / calmodulin-dependent protein phosphatase activity / RNA polymerase II CTD heptapeptide repeat S5 phosphatase activity / glycogen biosynthetic process / dephosphorylation / cytoplasmic side of rough endoplasmic reticulum membrane / glycogen metabolic process / protein-serine/threonine phosphatase / protein tyrosine phosphatase activity, metal-dependent / histone H2AXY142 phosphatase activity / protein serine/threonine phosphatase activity / phosphatase activity / negative regulation of cell cycle / peptidyl-tyrosine dephosphorylation / positive regulation of macroautophagy / autophagosome assembly / non-membrane spanning protein tyrosine phosphatase activity / regulation of protein ubiquitination / glial cell proliferation / regulation of protein localization to plasma membrane / protein dephosphorylation / regulation of proteasomal protein catabolic process / Myoclonic epilepsy of Lafora / Glycogen synthesis / protein-tyrosine-phosphatase / protein tyrosine phosphatase activity / mitochondrion organization / regulation of cell growth / Wnt signaling pathway / calcium ion transport / carbohydrate binding / proteasome-mediated ubiquitin-dependent protein catabolic process / perikaryon / protein dimerization activity / negative regulation of gene expression / dendrite / protein homodimerization activity / nucleoplasm / nucleus / plasma membrane / cytosol / cytoplasm類似検索 - 分子機能 Laforin, CBM20 domain / Laforin / Laforin-like, dual specificity phosphatase domain / Carbohydrate binding module family 20 / Starch binding domain / CBM20 (carbohydrate binding type-20) domain profile. / Starch binding domain / Dual specificity phosphatase, catalytic domain / Carbohydrate-binding-like fold / Dual specificity phosphatase, catalytic domain ...Laforin, CBM20 domain / Laforin / Laforin-like, dual specificity phosphatase domain / Carbohydrate binding module family 20 / Starch binding domain / CBM20 (carbohydrate binding type-20) domain profile. / Starch binding domain / Dual specificity phosphatase, catalytic domain / Carbohydrate-binding-like fold / Dual specificity phosphatase, catalytic domain / Dual specificity phosphatase, catalytic domain / Dual specificity protein phosphatase domain profile. / Dual specificity protein phosphatase domain / Protein tyrosine phosphatase superfamily / Protein-Tyrosine Phosphatase; Chain A / Tyrosine specific protein phosphatases active site. / Protein-tyrosine phosphatase, active site / Tyrosine-specific protein phosphatases domain / Tyrosine specific protein phosphatases domain profile. / Protein-tyrosine phosphatase-like / Immunoglobulin-like fold / Alpha-Beta Complex / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.3 Å 分子置換 / 解像度: 2.3 Å |
|---|
 データ登録者 データ登録者 | Sankhala, R.S. / Koksal, A.C. / Cingolani, G. |
|---|
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2015 ジャーナル: J.Biol.Chem. / 年: 2015
タイトル: Dimeric quaternary structure of human laforin.
著者: Sankhala, R.S. / Koksal, A.C. / Ho, L. / Nitschke, F. / Minassian, B.A. / Cingolani, G. |
|---|
| 履歴 | | 登録 | 2014年8月13日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2014年12月31日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2015年1月7日 | Group: Database references |
|---|
| 改定 1.2 | 2015年3月11日 | Group: Database references |
|---|
| 改定 1.3 | 2023年9月20日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_conn / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.3 Å
分子置換 / 解像度: 2.3 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Biol.Chem. / 年: 2015
ジャーナル: J.Biol.Chem. / 年: 2015 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4r30.cif.gz
4r30.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4r30.ent.gz
pdb4r30.ent.gz PDB形式
PDB形式 4r30.json.gz
4r30.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4r30_validation.pdf.gz
4r30_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4r30_full_validation.pdf.gz
4r30_full_validation.pdf.gz 4r30_validation.xml.gz
4r30_validation.xml.gz 4r30_validation.cif.gz
4r30_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/r3/4r30
https://data.pdbj.org/pub/pdb/validation_reports/r3/4r30 ftp://data.pdbj.org/pub/pdb/validation_reports/r3/4r30
ftp://data.pdbj.org/pub/pdb/validation_reports/r3/4r30
 リンク
リンク 集合体
集合体




 要素
要素 Homo sapiens (ヒト) / 遺伝子: EPM2A / プラスミド: pQE80L / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: EPM2A / プラスミド: pQE80L / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  NSLS
NSLS  / ビームライン: X29A / 波長: 1.07 Å
/ ビームライン: X29A / 波長: 1.07 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj











