+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4bul | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Novel hydroxyl tricyclics (e.g. GSK966587) as potent inhibitors of bacterial type IIA topoisomerases | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | ISOMERASE / TYPE IIA TOPOISOMERASES / NBTIS | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA topoisomerase type II (double strand cut, ATP-hydrolyzing) complex / DNA negative supercoiling activity / DNA topoisomerase (ATP-hydrolysing) / DNA topological change / DNA-templated DNA replication / chromosome / response to antibiotic / DNA binding / ATP binding / metal ion binding / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  SYNTHETIC CONSTRUCT (人工物) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.6 Å 分子置換 / 解像度: 2.6 Å | ||||||
 データ登録者 データ登録者 | Miles, T.J. / Hennessy, A.J. / Bax, B. / Brooks, G. / Brown, B.S. / Brown, P. / Cailleau, N. / Chen, D. / Dabbs, S. / Davies, D.T. ...Miles, T.J. / Hennessy, A.J. / Bax, B. / Brooks, G. / Brown, B.S. / Brown, P. / Cailleau, N. / Chen, D. / Dabbs, S. / Davies, D.T. / Esken, J.M. / Giordano, I. / Hoover, J.L. / Huang, J. / Jones, G.E. / Sukmar, S.K.K. / Spitzfaden, C. / Markwell, R.E. / Minthorn, E.A. / Rittenhouse, S. / Gwynn, M.N. / Pearson, N.D. | ||||||
 引用 引用 |  ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2013 ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2013タイトル: Novel Hydroxyl Tricyclics (E.G., Gsk966587) as Potent Inhibitors of Bacterial Type Iia Topoisomerases. 著者: Miles, T.J. / Hennessy, A.J. / Bax, B. / Brooks, G. / Brown, B.S. / Brown, P. / Cailleau, N. / Chen, D. / Dabbs, S. / Davies, D.T. / Esken, J.M. / Giordano, I. / Hoover, J.L. / Huang, J. / ...著者: Miles, T.J. / Hennessy, A.J. / Bax, B. / Brooks, G. / Brown, B.S. / Brown, P. / Cailleau, N. / Chen, D. / Dabbs, S. / Davies, D.T. / Esken, J.M. / Giordano, I. / Hoover, J.L. / Huang, J. / Jones, G.E. / Kusalakumari Sukmar, S.K. / Spitzfaden, C. / Markwell, R.E. / Minthorn, E.A. / Rittenhouse, S. / Gwynn, M.N. / Pearson, N.D. #1:  ジャーナル: Nature / 年: 2010 ジャーナル: Nature / 年: 2010タイトル: Type Iia Topoisomerase Inhibition by a New Class of Antibacterial Agents. 著者: Bax, B.D. / Chan, P.F. / Eggleston, D.S. / Fosberry, A. / Gentry, D.R. / Gorrec, F. / Giordano, I. / Hann, M.M. / Hennessy, A. / Hibbs, M. / Huang, J. / Jones, E. / Jones, J. / Brown, K.K. / ...著者: Bax, B.D. / Chan, P.F. / Eggleston, D.S. / Fosberry, A. / Gentry, D.R. / Gorrec, F. / Giordano, I. / Hann, M.M. / Hennessy, A. / Hibbs, M. / Huang, J. / Jones, E. / Jones, J. / Brown, K.K. / Lewis, C.J. / May, E.W. / Saunders, M.R. / Singh, O. / Spitzfaden, C.E. / Shen, C. / Shillings, A. / Theobald, A.J. / Wohlkonig, A. / Pearson, N.D. / Gwynn, M.N. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4bul.cif.gz 4bul.cif.gz | 627.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4bul.ent.gz pdb4bul.ent.gz | 511.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4bul.json.gz 4bul.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  4bul_validation.pdf.gz 4bul_validation.pdf.gz | 864.7 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  4bul_full_validation.pdf.gz 4bul_full_validation.pdf.gz | 878.1 KB | 表示 | |
| XML形式データ |  4bul_validation.xml.gz 4bul_validation.xml.gz | 52.4 KB | 表示 | |
| CIF形式データ |  4bul_validation.cif.gz 4bul_validation.cif.gz | 75.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bu/4bul https://data.pdbj.org/pub/pdb/validation_reports/bu/4bul ftp://data.pdbj.org/pub/pdb/validation_reports/bu/4bul ftp://data.pdbj.org/pub/pdb/validation_reports/bu/4bul | HTTPS FTP |
-関連構造データ
| 関連構造データ |  2xcsS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 2分子 AC
| #1: タンパク質 | 分子量: 78109.000 Da / 分子数: 2 断片: GYRA N-TERMINAL 56KDA DOMAIN, RESIDUES 2-491, GYRB C-TERMINAL 27KDA DOMAIN, RESIDUES 410-644 変異: YES / 由来タイプ: 組換発現 詳細: FUSION TRUNCATE WITH DELETION AND Y123F GYRB27A56GKDELY123F (BAX ET AL., 2010) 由来: (組換発現)  発現宿主:  参照: UniProt: P66937, UniProt: Q99XG5, EC: 5.99.1.3, EC: 5.99.1.3 |
|---|
-DNA鎖 , 2種, 4分子 EGFH
| #2: DNA鎖 | 分子量: 6156.963 Da / 分子数: 2 / 由来タイプ: 合成 / 由来: (合成) SYNTHETIC CONSTRUCT (人工物) #3: DNA鎖 | 分子量: 6112.970 Da / 分子数: 2 / 由来タイプ: 合成 / 由来: (合成) SYNTHETIC CONSTRUCT (人工物) |
|---|
-非ポリマー , 3種, 453分子 
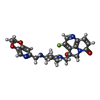



| #4: 化合物 | ChemComp-MN / #5: 化合物 | ChemComp-AE8 / ( | #6: 水 | ChemComp-HOH / | |
|---|
-詳細
| 非ポリマーの詳細 | GSK966587 (S)-4-((4-(((2,3-DIHYDRO-[1,4]DIOXINO[2, 3-C]PYRIDIN-7-YL)METHYL)AMINO)PIPERIDIN-1-YL) ...GSK966587 (S)-4-((4-(((2,3-DIHYDRO-[1,4]DIOXINO[2, 3-C]PYRIDIN-7-YL)METHYL)AMINO)PIPERIDIN-1-YL)METHYL)- 3-FLUORO-4-HYDROXY-4H-PYRROLO[3,2,1-DE][1,5] NAPHTHYRID |
|---|---|
| 配列の詳細 | C-TERMINAL DOMAIN (410-644) WITH GREEK KEY DOMAIN (544-579) DELETED AND REPLACED WITH TWO RESIDUES, ...C-TERMINAL DOMAIN (410-644) WITH GREEK KEY DOMAIN (544-579) DELETED AND REPLACED WITH TWO RESIDUES, TG. CATALYTIC TYROSINE MUTATED TO PHENYLALAN |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.15 Å3/Da / 溶媒含有率: 60.94 % / 解説: NONE |
|---|
-データ収集
| 回折 | 平均測定温度: 100 K | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: ID23-1 / 波長: 0.96865 / ビームライン: ID23-1 / 波長: 0.96865 | |||||||||||||||
| 検出器 | タイプ: ADSC QUANTUM 315 / 検出器: CCD | |||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||||||||
| 放射波長 | 波長: 0.96865 Å / 相対比: 1 | |||||||||||||||
| Reflection twin |
| |||||||||||||||
| 反射 | 解像度: 2.6→81.38 Å / Num. obs: 60721 / % possible obs: 95.8 % / Observed criterion σ(I): 0 / 冗長度: 2.4 % / Rmerge(I) obs: 0.09 / Net I/σ(I): 9.5 | |||||||||||||||
| 反射 シェル | 解像度: 2.6→2.74 Å / 冗長度: 2.4 % / Rmerge(I) obs: 0.38 / Mean I/σ(I) obs: 2.3 / % possible all: 96.7 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 2XCS 解像度: 2.6→24.79 Å / Cor.coef. Fo:Fc: 0.959 / Cor.coef. Fo:Fc free: 0.932 / SU B: 11.49 / SU ML: 0.136 / 交差検証法: THROUGHOUT / ESU R: 0.107 / ESU R Free: 0.048 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: U VALUES WITH TLS ADDED
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: BABINET MODEL WITH MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.6→24.79 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj







































