+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2x7r | ||||||
|---|---|---|---|---|---|---|---|
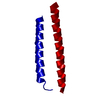
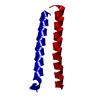
| タイトル | Crystal structure of a late fusion intermediate of HIV-1 gp41 | ||||||
 要素 要素 | (TRANSMEMBRANE PROTEIN GP41) x 2 | ||||||
 キーワード キーワード | VIRAL PROTEIN / ENVELOPE GLYCOPROTEIN / MEMBRANE ANCHORED FUSION PROTEIN | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Synthesis and processing of ENV and VPU / symbiont-mediated evasion of host immune response / positive regulation of establishment of T cell polarity / Alpha-defensins / Dectin-2 family / Binding and entry of HIV virion / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / actin filament organization / host cell endosome membrane ...Synthesis and processing of ENV and VPU / symbiont-mediated evasion of host immune response / positive regulation of establishment of T cell polarity / Alpha-defensins / Dectin-2 family / Binding and entry of HIV virion / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / actin filament organization / host cell endosome membrane / Assembly Of The HIV Virion / Budding and maturation of HIV virion / clathrin-dependent endocytosis of virus by host cell / viral protein processing / receptor ligand activity / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / symbiont entry into host cell / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  HUMAN IMMUNODEFICIENCY VIRUS TYPE 1 LW12.3 ISOLATE (ヒト免疫不全ウイルス) HUMAN IMMUNODEFICIENCY VIRUS TYPE 1 LW12.3 ISOLATE (ヒト免疫不全ウイルス) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2 Å 分子置換 / 解像度: 2 Å | ||||||
 データ登録者 データ登録者 | Natrajan, G. / Buzon, V. / Weissenhorn, W. | ||||||
 引用 引用 |  ジャーナル: Plos Pathog. / 年: 2010 ジャーナル: Plos Pathog. / 年: 2010タイトル: Crystal Structure of HIV-1 Gp41 Including Both Fusion Peptide and Membrane Proximal External Regions. 著者: Buzon, V. / Natrajan, G. / Schibli, D. / Campelo, F. / Kozlov, M.M. / Weissenhorn, W. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2x7r.cif.gz 2x7r.cif.gz | 74.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2x7r.ent.gz pdb2x7r.ent.gz | 56.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2x7r.json.gz 2x7r.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2x7r_validation.pdf.gz 2x7r_validation.pdf.gz | 467.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2x7r_full_validation.pdf.gz 2x7r_full_validation.pdf.gz | 479.1 KB | 表示 | |
| XML形式データ |  2x7r_validation.xml.gz 2x7r_validation.xml.gz | 14.4 KB | 表示 | |
| CIF形式データ |  2x7r_validation.cif.gz 2x7r_validation.cif.gz | 19 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/x7/2x7r https://data.pdbj.org/pub/pdb/validation_reports/x7/2x7r ftp://data.pdbj.org/pub/pdb/validation_reports/x7/2x7r ftp://data.pdbj.org/pub/pdb/validation_reports/x7/2x7r | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1aikS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||||
| 2 | 
| ||||||||||||||||||
| 3 | 
| ||||||||||||||||||
| 単位格子 |
| ||||||||||||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: タンパク質 | 分子量: 6837.859 Da / 分子数: 3 / 断片: EXTRA CELLULAR DOMAIN, RESIDUES 534-581 / 由来タイプ: 組換発現 由来: (組換発現)  HUMAN IMMUNODEFICIENCY VIRUS TYPE 1 LW12.3 ISOLATE (ヒト免疫不全ウイルス) HUMAN IMMUNODEFICIENCY VIRUS TYPE 1 LW12.3 ISOLATE (ヒト免疫不全ウイルス)発現宿主:  #2: タンパク質 | 分子量: 7825.539 Da / 分子数: 3 / 断片: EXTRA CELLULAR DOMAIN, RESIDUES 629-683 / 由来タイプ: 組換発現 由来: (組換発現)  HUMAN IMMUNODEFICIENCY VIRUS TYPE 1 LW12.3 ISOLATE (ヒト免疫不全ウイルス) HUMAN IMMUNODEFICIENCY VIRUS TYPE 1 LW12.3 ISOLATE (ヒト免疫不全ウイルス)発現宿主:  #3: 化合物 | #4: 化合物 | ChemComp-CL / | #5: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.05 Å3/Da / 溶媒含有率: 40.16 % / 解説: NONE |
|---|---|
| 結晶化 | pH: 6 / 詳細: 0.1M CITRIC ACID PH 6, 60% MPD |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: ID14-4 / 波長: 0.97 / ビームライン: ID14-4 / 波長: 0.97 |
| 検出器 | タイプ: ADSC CCD / 検出器: CCD / 日付: 2009年6月1日 / 詳細: TOROIDAL |
| 放射 | モノクロメーター: CHANNEL CUT ESRF / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.97 Å / 相対比: 1 |
| Reflection twin | Operator: h,-h-k,-l / Fraction: 0.505 |
| 反射 | 解像度: 2→91.38 Å / Num. obs: 22184 / % possible obs: 96.5 % / Observed criterion σ(I): 2.2 / 冗長度: 5.9 % / Biso Wilson estimate: 23.2 Å2 / Rmerge(I) obs: 0.06 / Net I/σ(I): 17.9 |
| 反射 シェル | 解像度: 2→2.11 Å / 冗長度: 2.4 % / Rmerge(I) obs: 0.33 / Mean I/σ(I) obs: 2.8 / % possible all: 84 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 1AIK 解像度: 2→49.729 Å / σ(F): 1.48 / 位相誤差: 21.27 / 立体化学のターゲット値: TWIN_LSQ_F
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL / Bsol: 87.855 Å2 / ksol: 0.367 e/Å3 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2→49.729 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー
























 PDBj
PDBj













