[English] 日本語
 Yorodumi
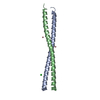
Yorodumi- PDB-5vpc: Transcription factor FosB/JunD bZIP domain in its oxidized form, ... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5vpc | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Transcription factor FosB/JunD bZIP domain in its oxidized form, type-II crystal | |||||||||
 Components Components |
| |||||||||
 Keywords Keywords | TRANSCRIPTION / activator protein-1 / basic leucine zipper / bZIP / fos / jun / transcription factor / DNA-binding protein / redox switch / coiled-coil | |||||||||
| Function / homology |  Function and homology information Function and homology informationtranscription factor AP-1 complex / positive regulation of peroxisome proliferator activated receptor signaling pathway / NGF-stimulated transcription / positive regulation of macrophage activation / cellular response to fatty acid / response to steroid hormone / osteoblast development / response to corticosterone / response to morphine / response to light stimulus ...transcription factor AP-1 complex / positive regulation of peroxisome proliferator activated receptor signaling pathway / NGF-stimulated transcription / positive regulation of macrophage activation / cellular response to fatty acid / response to steroid hormone / osteoblast development / response to corticosterone / response to morphine / response to light stimulus / response to cAMP / positive regulation of osteoblast differentiation / response to mechanical stimulus / cellular response to hormone stimulus / behavioral response to cocaine / transcription repressor complex / response to progesterone / cellular response to calcium ion / nuclear receptor binding / response to amphetamine / transcription coregulator binding / response to nicotine / female pregnancy / circadian rhythm / protein-DNA complex / response to peptide hormone / RNA polymerase II transcription regulator complex / sequence-specific double-stranded DNA binding / regulation of cell population proliferation / DNA-binding transcription activator activity, RNA polymerase II-specific / response to lipopolysaccharide / transcription regulator complex / cellular response to hypoxia / Estrogen-dependent gene expression / response to ethanol / transcription by RNA polymerase II / DNA-binding transcription factor activity, RNA polymerase II-specific / transcription coactivator activity / transcription cis-regulatory region binding / regulation of cell cycle / RNA polymerase II cis-regulatory region sequence-specific DNA binding / response to xenobiotic stimulus / intracellular membrane-bounded organelle / regulation of transcription by RNA polymerase II / chromatin / enzyme binding / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / DNA binding / nucleoplasm / nucleus / cytosol Similarity search - Function | |||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.498 Å MOLECULAR REPLACEMENT / Resolution: 2.498 Å | |||||||||
 Authors Authors | Yin, Z. / Machius, M.C. / Rudenko, G. | |||||||||
| Funding support |  United States, 2items United States, 2items
| |||||||||
 Citation Citation |  Journal: Nucleic Acids Res. / Year: 2017 Journal: Nucleic Acids Res. / Year: 2017Title: Activator Protein-1: redox switch controlling structure and DNA-binding. Authors: Yin, Z. / Machius, M. / Nestler, E.J. / Rudenko, G. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5vpc.cif.gz 5vpc.cif.gz | 153.1 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5vpc.ent.gz pdb5vpc.ent.gz | 124.6 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5vpc.json.gz 5vpc.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/vp/5vpc https://data.pdbj.org/pub/pdb/validation_reports/vp/5vpc ftp://data.pdbj.org/pub/pdb/validation_reports/vp/5vpc ftp://data.pdbj.org/pub/pdb/validation_reports/vp/5vpc | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5vpaSC  5vpbC  5vpdC  5vpeC  5vpfC S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Unit cell |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Noncrystallographic symmetry (NCS) | NCS domain:
NCS domain segments:
|
 Movie
Movie Controller
Controller











 PDBj
PDBj


