+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2co5 | ||||||
|---|---|---|---|---|---|---|---|
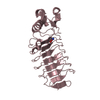
| タイトル | F93 FROM STIV, a winged-helix DNA-binding protein | ||||||
 要素 要素 | VIRAL PROTEIN F93 | ||||||
 キーワード キーワード | VIRAL PROTEIN/WINGED HELIX / VIRAL PROTEIN-WINGED HELIX COMPLEX / WINGED HELIX / DNA-BINDING / HTH / WHTH / F93 / DISULFIDE BOND / STIV / SULFOLOBUS TURRETED ICOSAHEDRAL VIRUS / VIRAL PROTEIN / VIRUS / ARCHAEA / CRENARCHAEA / ARCHAEAL VIRUS / CRENARCHAEAL VIRUS / THERMOPHILIC PROTEIN / THERMOPHILIC VIRUS / SULFOLOBUS / YELLOWSTONE | ||||||
| 機能・相同性 | Winged helix-like DNA-binding domain superfamily/Winged helix DNA-binding domain / Arc Repressor Mutant, subunit A / Winged helix DNA-binding domain superfamily / Winged helix-like DNA-binding domain superfamily / Orthogonal Bundle / Mainly Alpha / PadR family transcriptional regulator 機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |    SULFOLOBUS TURRETED ICOSAHEDRAL VIRUS (ウイルス) SULFOLOBUS TURRETED ICOSAHEDRAL VIRUS (ウイルス) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単波長異常分散 / 解像度: 2.2 Å 単波長異常分散 / 解像度: 2.2 Å | ||||||
 データ登録者 データ登録者 | Larson, E.T. / Reiter, D. / Lawrence, C.M. | ||||||
 引用 引用 |  ジャーナル: Virology / 年: 2007 ジャーナル: Virology / 年: 2007タイトル: A Winged-Helix Protein from Sulfolobus Turreted Icosahedral Virus Points Toward Stabilizing Disulfide Bonds in the Intracellular Proteins of a Hyperthermophilic Virus. 著者: Larson, E.T. / Eilers, B. / Menon, S. / Reiter, D. / Ortmann, A. / Young, M.J. / Lawrence, C.M. #1:  ジャーナル: Proc Natl Acad Sci U S A / 年: 2004 ジャーナル: Proc Natl Acad Sci U S A / 年: 2004タイトル: The structure of a thermophilic archaeal virus shows a double-stranded DNA viral capsid type that spans all domains of life. 著者: George Rice / Liang Tang / Kenneth Stedman / Francisco Roberto / Josh Spuhler / Eric Gillitzer / John E Johnson / Trevor Douglas / Mark Young /  要旨: Of the three domains of life (Eukarya, Bacteria, and Archaea), the least understood is Archaea and its associated viruses. Many Archaea are extremophiles, with species that are capable of growth at ...Of the three domains of life (Eukarya, Bacteria, and Archaea), the least understood is Archaea and its associated viruses. Many Archaea are extremophiles, with species that are capable of growth at some of the highest temperatures and extremes of pH of all known organisms. Phylogenetic rRNA-encoding DNA analysis places many of the hyperthermophilic Archaea (species with an optimum growth > or = 80 degrees C) at the base of the universal tree of life, suggesting that thermophiles were among the first forms of life on earth. Very few viruses have been identified from Archaea as compared to Bacteria and Eukarya. We report here the structure of a hyperthermophilic virus isolated from an archaeal host found in hot springs in Yellowstone National Park. The sequence of the circular double-stranded DNA viral genome shows that it shares little similarity to other known genes in viruses or other organisms. By comparing the tertiary and quaternary structures of the coat protein of this virus with those of a bacterial and an animal virus, we find conformational relationships among all three, suggesting that some viruses may have a common ancestor that precedes the division into three domains of life >3 billion years ago. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2co5.cif.gz 2co5.cif.gz | 53.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2co5.ent.gz pdb2co5.ent.gz | 41.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2co5.json.gz 2co5.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/co/2co5 https://data.pdbj.org/pub/pdb/validation_reports/co/2co5 ftp://data.pdbj.org/pub/pdb/validation_reports/co/2co5 ftp://data.pdbj.org/pub/pdb/validation_reports/co/2co5 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: タンパク質 | 分子量: 12000.183 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)    SULFOLOBUS TURRETED ICOSAHEDRAL VIRUS (ウイルス) SULFOLOBUS TURRETED ICOSAHEDRAL VIRUS (ウイルス)株: ISOLATE YNPRC179 解説: STIV WAS ISOLATED FROM SULFOLOBUS SPECIES IN ACIDIC HOT SPRINGS (PH 2.9-3.9, 72-92 DEGREES C)IN THE RABBIT CREEK THERMAL AREA WITHIN MIDWAY GEYSER BASIN IN YELLOWSTONE NATIONAL PARK, USA. プラスミド: PEXP14-STIVF93 / 発現宿主:  #2: 水 | ChemComp-HOH / | Has protein modification | Y | 配列の詳細 | C-TERMINAL 6XHIS TAG WAS ADDED DURING CLONING TO FACILITATE | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.1 Å3/Da / 溶媒含有率: 41 % / 解説: NONE |
|---|---|
| 結晶化 | 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 4.75 詳細: HANGING DROP VAPOR DIFFUSION, 9.0-10.5 MG/ML F93 IN 10 MM TRIS-HCL, PH 8.0, 50 MM NACL, 0.5 MM TCEP MIXED WITH BUFFER CONTAINING 0.1 M SODIUM ACETATE, PH 4.75-5.0, 0.1 M MAGNESIUM NITRATE ...詳細: HANGING DROP VAPOR DIFFUSION, 9.0-10.5 MG/ML F93 IN 10 MM TRIS-HCL, PH 8.0, 50 MM NACL, 0.5 MM TCEP MIXED WITH BUFFER CONTAINING 0.1 M SODIUM ACETATE, PH 4.75-5.0, 0.1 M MAGNESIUM NITRATE HEXAHYDRATE, 16-20% PEG 20,000. |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SSRL SSRL  / ビームライン: BL9-1 / 波長: 0.98789 / ビームライン: BL9-1 / 波長: 0.98789 |
| 検出器 | タイプ: ADSC QUANTUM 315 / 検出器: CCD / 日付: 2006年3月17日 詳細: VERTICAL FOCUSING MIRROR, SINGLE CRYSTAL SI(311) BENT MONOCHROMATOR (HORIZONTAL FOCUSING) |
| 放射 | モノクロメーター: SIDE-SCATTERING CUBEROOT I- BEAM BENT SINGLE CRYSTAL, ASYMETRIC CUT 12.2 DEGS. プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.98789 Å / 相対比: 1 |
| 反射 | 解像度: 2.2→50 Å / Num. obs: 10466 / % possible obs: 99.8 % / Observed criterion σ(I): 3 / 冗長度: 7.1 % / Rmerge(I) obs: 0.06 / Net I/σ(I): 33 |
| 反射 シェル | 解像度: 2.2→2.28 Å / 冗長度: 7.3 % / Rmerge(I) obs: 0.34 / Mean I/σ(I) obs: 4.2 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  単波長異常分散 単波長異常分散開始モデル: NONE 解像度: 2.2→46.37 Å / Cor.coef. Fo:Fc: 0.957 / Cor.coef. Fo:Fc free: 0.941 / SU B: 13.322 / SU ML: 0.17 / TLS residual ADP flag: LIKELY RESIDUAL / 交差検証法: THROUGHOUT / ESU R: 0.304 / ESU R Free: 0.213 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS. TLS MOTION DETERMINATION SERVER (J PAINTER & E A MERRITT (2006) J. APPL. CRYST. 39, 109-111) WAS USED FOR SELECTION OF OPTIMAL TLS GROUPS USED IN REFINEMENT.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: BABINET MODEL WITH MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 42.8 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.2→46.37 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj