+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1uwp | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Initial Events in the Photocycle of Photoactive Yellow Protein | ||||||
 要素 要素 | PHOTOACTIVE YELLOW PROTEIN | ||||||
 キーワード キーワード | SIGNALING PROTEIN / PAS / LOV / GAF / DOMAINS / FOLD PHOTOCYCLE / PHOTOACTIVE / PHOTORECEPTOR / YELLOW | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報photoreceptor activity / phototransduction / regulation of DNA-templated transcription / identical protein binding 類似検索 - 分子機能 | ||||||
| 生物種 |  HALORHODOSPIRA HALOPHILA (紅色硫黄細菌) HALORHODOSPIRA HALOPHILA (紅色硫黄細菌) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多波長異常分散 / 解像度: 1.2 Å 多波長異常分散 / 解像度: 1.2 Å | ||||||
 データ登録者 データ登録者 | Kort, R. / Hellingwerf, K.J. / Ravelli, R.B.G. | ||||||
 引用 引用 |  ジャーナル: J. Biol. Chem. / 年: 2004 ジャーナル: J. Biol. Chem. / 年: 2004タイトル: Initial events in the photocycle of photoactive yellow protein. 著者: Kort, R. / Hellingwerf, K.J. / Ravelli, R.B. #1: ジャーナル: Photochem.Photobiol. / 年: 2003 タイトル: Characterization of Photocycle Intermediates in Crystalline Photoactive Yellow Protein 著者: Kort, R. / Ravelli, R.B.G. / Schotte, F. / Bourgeois, D. / Crielaard, W. / Hellingwerf, K.J. / Wulff, M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1uwp.cif.gz 1uwp.cif.gz | 76.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1uwp.ent.gz pdb1uwp.ent.gz | 58.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1uwp.json.gz 1uwp.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1uwp_validation.pdf.gz 1uwp_validation.pdf.gz | 388.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1uwp_full_validation.pdf.gz 1uwp_full_validation.pdf.gz | 390.9 KB | 表示 | |
| XML形式データ |  1uwp_validation.xml.gz 1uwp_validation.xml.gz | 4.8 KB | 表示 | |
| CIF形式データ |  1uwp_validation.cif.gz 1uwp_validation.cif.gz | 7.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/uw/1uwp https://data.pdbj.org/pub/pdb/validation_reports/uw/1uwp ftp://data.pdbj.org/pub/pdb/validation_reports/uw/1uwp ftp://data.pdbj.org/pub/pdb/validation_reports/uw/1uwp | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||
| 単位格子 |
| ||||||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: タンパク質 | 分子量: 14123.052 Da / 分子数: 1 / 由来タイプ: 組換発現 / 詳細: LIGHT STATE 由来: (組換発現)  HALORHODOSPIRA HALOPHILA (紅色硫黄細菌) HALORHODOSPIRA HALOPHILA (紅色硫黄細菌)株: BN9626 解説: HALORHODOSPIRA HALOPHILA PREVIOUSLY KNOWN AS ECTOTHIORHODOSPIRA HALOPHILA プラスミド: PQE30 / 発現宿主:  | ||||||
|---|---|---|---|---|---|---|---|
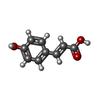
| #2: 化合物 | ChemComp-HC4 / | ||||||
| #3: 化合物 | | #4: 水 | ChemComp-HOH / | Has protein modification | Y | 配列の詳細 | THE MET-RESIDUES WERE SUBSTITUTE | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.72 Å3/Da / 溶媒含有率: 20 % / 解説: STATISTICS ARE GIVEN FOR REMOTE DATA SET (2 SWEEPS) |
|---|---|
| 結晶化 | 温度: 298 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7 詳細: 2.25 M NH4SO4, 20 MM SODIUM PHOSPHATE PH 7, VAPOR DIFFUSION, HANGING DROP, TEMP 298K |
-データ収集
| 回折 | 平均測定温度: 85 K | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: ID14-4 / 波長: 0.9393,0.9796 / ビームライン: ID14-4 / 波長: 0.9393,0.9796 | |||||||||
| 検出器 | タイプ: ADSC CCD / 検出器: CCD / 日付: 2002年3月30日 / 詳細: BENT MIRROR | |||||||||
| 放射 | モノクロメーター: SI(111) / プロトコル: MAD / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||
| 放射波長 |
| |||||||||
| 反射 | 解像度: 1.2→30 Å / Num. obs: 146753 / % possible obs: 98.3 % / Observed criterion σ(I): -3 / 冗長度: 4.7 % / Rmerge(I) obs: 0.035 / Net I/σ(I): 14.6 | |||||||||
| 反射 シェル | 解像度: 1.2→1.23 Å / 冗長度: 3.2 % / Rmerge(I) obs: 0.199 / Mean I/σ(I) obs: 4.5 / % possible all: 94.4 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  多波長異常分散 / 解像度: 1.2→40 Å / Cor.coef. Fo:Fc: 0.978 / Cor.coef. Fo:Fc free: 0.972 / 交差検証法: THROUGHOUT / ESU R: 0.046 / ESU R Free: 0.041 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD 多波長異常分散 / 解像度: 1.2→40 Å / Cor.coef. Fo:Fc: 0.978 / Cor.coef. Fo:Fc free: 0.972 / 交差検証法: THROUGHOUT / ESU R: 0.046 / ESU R Free: 0.041 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS. AN EARLY INTERMEDIATE OF THE PHOTOCYCLE OF PYP WAS DETERMINED BY THE DATA COLLECTION OF 2 MAD DATA SETS ON ONE CRYSTAL OF PYP. THE FIRST MAD ...詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS. AN EARLY INTERMEDIATE OF THE PHOTOCYCLE OF PYP WAS DETERMINED BY THE DATA COLLECTION OF 2 MAD DATA SETS ON ONE CRYSTAL OF PYP. THE FIRST MAD DATA SET WAS OBTAINED IN THE DARK, WHEREAS THE CRYSTAL WAS ILLUMINATED FOR THE SECOND DATA SET. DATA WERE CORRECTED FOR MINOR RADIATION DAMAGE USING 2 OTHER MAD DATA SETS, COLLECTED ON AN ISOMORPHOUS CRYSTAL THAT WAS KEPT IN THE DARK. THE SAME CRYSTAL WAS USED FOR PDB- ID 1UWN.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.4 Å / 溶媒モデル: BABINET MODEL PLUS MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 10.61 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.2→40 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー

























 PDBj
PDBj