+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1mex | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Antibody Catalysis of a Bimolecular Cycloaddition Reaction | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | IMMUNE SYSTEM / immunoglobulin fold | ||||||
| 機能・相同性 | Immunoglobulins / Immunoglobulin-like / Sandwich / Mainly Beta / Chem-RAC 機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.25 Å 分子置換 / 解像度: 1.25 Å | ||||||
 データ登録者 データ登録者 | Heine, A. / Wilson, I.A. | ||||||
 引用 引用 |  ジャーナル: To be Published ジャーナル: To be Publishedタイトル: Antibody Catalysis of a Bimolecular Cycloaddition Reaction 著者: Heine, A. / Wilson, I.A. | ||||||
| 履歴 |
| ||||||
| Remark 999 | SEQRES Author states the sequence should correspond to other antibody FAB sequences listed in the ...SEQRES Author states the sequence should correspond to other antibody FAB sequences listed in the Kabat and Wu "Sequence of proteins of immunological interest". In addition, author states residues 133 and 134 of chain H were refined as ALA, since there was no conclusive side chain density. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1mex.cif.gz 1mex.cif.gz | 197.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1mex.ent.gz pdb1mex.ent.gz | 163.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1mex.json.gz 1mex.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/me/1mex https://data.pdbj.org/pub/pdb/validation_reports/me/1mex ftp://data.pdbj.org/pub/pdb/validation_reports/me/1mex ftp://data.pdbj.org/pub/pdb/validation_reports/me/1mex | HTTPS FTP |
|---|
-関連構造データ
| 類似構造データ |
|---|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: 抗体 | 分子量: 23625.949 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
|---|---|
| #2: 抗体 | 分子量: 22790.404 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
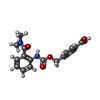
| #3: 化合物 | ChemComp-RAC / |
| #4: 水 | ChemComp-HOH / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.89 Å3/Da / 溶媒含有率: 35 % |
|---|---|
| 結晶化 | 温度: 295.5 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7 詳細: MPEG2000, pH 7.0, VAPOR DIFFUSION, SITTING DROP, temperature 295.5K |
-データ収集
| 回折 | 平均測定温度: 84 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SSRL SSRL  / ビームライン: BL11-1 / 波長: 0.96482 Å / ビームライン: BL11-1 / 波長: 0.96482 Å |
| 検出器 | タイプ: ADSC QUANTUM 4 / 検出器: CCD / 日付: 2001年5月6日 / 詳細: single crystal bent |
| 放射 | モノクロメーター: Si / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.96482 Å / 相対比: 1 |
| 反射 | 解像度: 1.25→20 Å / Num. all: 112412 / Num. obs: 112412 / % possible obs: 99.5 % / Observed criterion σ(I): -3 / 冗長度: 3.9 % / Rsym value: 0.043 / Net I/σ(I): 27.4 |
| 反射 シェル | 解像度: 1.25→1.28 Å / Mean I/σ(I) obs: 1.9 / Num. unique all: 7422 / Rsym value: 0.638 / % possible all: 99.5 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 / 解像度: 1.25→10 Å / Num. parameters: 34859 / Num. restraintsaints: 41965 / 交差検証法: FREE R / σ(F): 0 / 立体化学のターゲット値: ENGH AND HUBER 分子置換 / 解像度: 1.25→10 Å / Num. parameters: 34859 / Num. restraintsaints: 41965 / 交差検証法: FREE R / σ(F): 0 / 立体化学のターゲット値: ENGH AND HUBER詳細: ANISOTROPIC SCALING APPLIED BY THE METHOD OF PARKIN, MOEZZI & HOPE, J.APPL.CRYST.28(1995)53-56 ANISOTROPIC REFINEMENT REDUCED FREE R
| |||||||||||||||||||||||||||||||||
| Refine analyze | Num. disordered residues: 12 / Occupancy sum hydrogen: 3128 / Occupancy sum non hydrogen: 3842 | |||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.25→10 Å
| |||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー























 PDBj
PDBj