+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3744 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Pronase-treated straight filament in Alzheimer's disease brain | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  Human (ヒト) Human (ヒト) | |||||||||
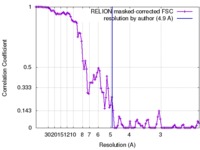
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 4.9 Å | |||||||||
 データ登録者 データ登録者 | Fitzpatrick AWP / Falcon B / He S / Murzin AG / Murshudov G / Garringer HG / Crowther RA / Ghetti B / Goedert M / Scheres SHW | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2017 ジャーナル: Nature / 年: 2017タイトル: Cryo-EM structures of tau filaments from Alzheimer's disease. 著者: Anthony W P Fitzpatrick / Benjamin Falcon / Shaoda He / Alexey G Murzin / Garib Murshudov / Holly J Garringer / R Anthony Crowther / Bernardino Ghetti / Michel Goedert / Sjors H W Scheres /   要旨: Alzheimer's disease is the most common neurodegenerative disease, and there are no mechanism-based therapies. The disease is defined by the presence of abundant neurofibrillary lesions and neuritic ...Alzheimer's disease is the most common neurodegenerative disease, and there are no mechanism-based therapies. The disease is defined by the presence of abundant neurofibrillary lesions and neuritic plaques in the cerebral cortex. Neurofibrillary lesions comprise paired helical and straight tau filaments, whereas tau filaments with different morphologies characterize other neurodegenerative diseases. No high-resolution structures of tau filaments are available. Here we present cryo-electron microscopy (cryo-EM) maps at 3.4-3.5 Å resolution and corresponding atomic models of paired helical and straight filaments from the brain of an individual with Alzheimer's disease. Filament cores are made of two identical protofilaments comprising residues 306-378 of tau protein, which adopt a combined cross-β/β-helix structure and define the seed for tau aggregation. Paired helical and straight filaments differ in their inter-protofilament packing, showing that they are ultrastructural polymorphs. These findings demonstrate that cryo-EM allows atomic characterization of amyloid filaments from patient-derived material, and pave the way for investigation of a range of neurodegenerative diseases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3744.map.gz emd_3744.map.gz | 4.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3744-v30.xml emd-3744-v30.xml emd-3744.xml emd-3744.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3744_fsc.xml emd_3744_fsc.xml | 9.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3744.png emd_3744.png | 35.9 KB | ||
| その他 |  emd_3744_half_map_1.map.gz emd_3744_half_map_1.map.gz emd_3744_half_map_2.map.gz emd_3744_half_map_2.map.gz | 58.3 MB 66.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3744 http://ftp.pdbj.org/pub/emdb/structures/EMD-3744 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3744 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3744 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3744_validation.pdf.gz emd_3744_validation.pdf.gz | 352.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3744_full_validation.pdf.gz emd_3744_full_validation.pdf.gz | 351.5 KB | 表示 | |
| XML形式データ |  emd_3744_validation.xml.gz emd_3744_validation.xml.gz | 15.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3744 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3744 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3744 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3744 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3744.map.gz / 形式: CCP4 / 大きさ: 75.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3744.map.gz / 形式: CCP4 / 大きさ: 75.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
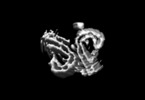
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.15 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
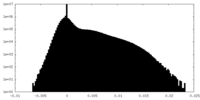
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: #1
| ファイル | emd_3744_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
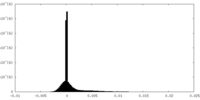
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_3744_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
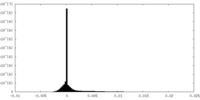
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Tau
| 全体 | 名称: Tau |
|---|---|
| 要素 |
|
-超分子 #1: Tau
| 超分子 | 名称: Tau / タイプ: tissue / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Tau
| 分子 | 名称: Tau / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human (ヒト) Human (ヒト) |
| 配列 | 文字列: VQIVYKPVDL SKVTSKCGSL GNIHHKPGGG QVEVKSEKLD FKDRVQSKIG SLDNITHVPG GGNKKIETHK LTF |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | tissue |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 20 mM Tris-HCl pH 7.4 containing 100 MM NaCl |
| グリッド | モデル: Quanitifoil Au R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | Sarkosyl-insoluble material was extracted from grey matter of frontal and temporal cortex from the patients brain and treated with pronase, as described in the Methods section of the paper. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum エネルギーフィルター - エネルギー下限: -10 eV エネルギーフィルター - エネルギー上限: 10 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 2-20 / 撮影したグリッド数: 1 / 実像数: 523 / 平均露光時間: 0.8 sec. / 平均電子線量: 2.5 e/Å2 詳細: images were collected in movie-mode at 1.2 frames per second |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 0.9 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.9 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Residue range: 226-242 |
|---|---|
| 詳細 | Fourier-space refinement of the complete atomic model against the paired helical filament and straight filament maps was performed in REFMAC. A stack of three consecutive monomers from each of the protofilaments was refined to preserve nearest-neighbour interactions for the middle chain. |
| 精密化 | 空間: RECIPROCAL / プロトコル: AB INITIO MODEL / 温度因子: 79 / 当てはまり具合の基準: Fourier shell correlation |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)