+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22349 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of human light chain apoferritin calculated from EER movies (intra-fraction motion correction experiment, no supersampling) | |||||||||
 マップデータ マップデータ | Sharpened map | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
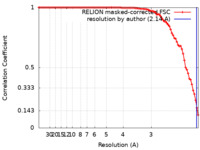
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.14 Å | |||||||||
 データ登録者 データ登録者 | Guo H / Rubinstein J | |||||||||
| 資金援助 |  カナダ, 2件 カナダ, 2件
| |||||||||
 引用 引用 |  ジャーナル: IUCrJ / 年: 2020 ジャーナル: IUCrJ / 年: 2020タイトル: Electron-event representation data enable efficient cryoEM file storage with full preservation of spatial and temporal resolution. 著者: Hui Guo / Erik Franken / Yuchen Deng / Samir Benlekbir / Garbi Singla Lezcano / Bart Janssen / Lingbo Yu / Zev A Ripstein / Yong Zi Tan / John L Rubinstein /   要旨: Direct detector device (DDD) cameras have revolutionized electron cryomicroscopy (cryoEM) with their high detective quantum efficiency (DQE) and output of movie data. A high ratio of camera frame ...Direct detector device (DDD) cameras have revolutionized electron cryomicroscopy (cryoEM) with their high detective quantum efficiency (DQE) and output of movie data. A high ratio of camera frame rate (frames per second) to camera exposure rate (electrons per pixel per second) allows electron counting, which further improves the DQE and enables the recording of super-resolution information. Movie output also allows the correction of specimen movement and compensation for radiation damage. However, these movies come at the cost of producing large volumes of data. It is common practice to sum groups of successive camera frames to reduce the final frame rate, and therefore the file size, to one suitable for storage and image processing. This reduction in the temporal resolution of the camera requires decisions to be made during data acquisition that may result in the loss of information that could have been advantageous during image analysis. Here, experimental analysis of a new electron-event representation (EER) data format for electron-counting DDD movies is presented, which is enabled by new hardware developed by Thermo Fisher Scientific for their Falcon DDD cameras. This format enables the recording of DDD movies at the raw camera frame rate without sacrificing either spatial or temporal resolution. Experimental data demonstrate that the method retains super-resolution information and allows the correction of specimen movement at the physical frame rate of the camera while maintaining manageable file sizes. The EER format will enable the development of new methods that can utilize the full spatial and temporal resolution of DDD cameras. #1:  ジャーナル: Biorxiv / 年: 2020 ジャーナル: Biorxiv / 年: 2020タイトル: Electron Event Representation (EER) data enables efficient cryoEM file storage with full preservation of spatial and temporal resolution 著者: Guo H / Franken E / Deng Y / Benlekbir S / Lezcano GS / Janssen B / Yu L / Ripstein ZA / Tan YZ / Rubinstein JL | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22349.map.gz emd_22349.map.gz | 59.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22349-v30.xml emd-22349-v30.xml emd-22349.xml emd-22349.xml | 18.8 KB 18.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22349_fsc.xml emd_22349_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22349.png emd_22349.png | 148.9 KB | ||
| マスクデータ |  emd_22349_msk_1.map emd_22349_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_22349_half_map_1.map.gz emd_22349_half_map_1.map.gz emd_22349_half_map_2.map.gz emd_22349_half_map_2.map.gz | 45.6 MB 45.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22349 http://ftp.pdbj.org/pub/emdb/structures/EMD-22349 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22349 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22349 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22349_validation.pdf.gz emd_22349_validation.pdf.gz | 78.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22349_full_validation.pdf.gz emd_22349_full_validation.pdf.gz | 77.6 KB | 表示 | |
| XML形式データ |  emd_22349_validation.xml.gz emd_22349_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22349 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22349 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22349 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22349 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10474 (タイトル: EER movies of human light chain apoferritin / Data size: 642.1 EMPIAR-10474 (タイトル: EER movies of human light chain apoferritin / Data size: 642.1 Data #1: Falcon3 EER movies of human light chain apoferritin at 75000 nominal magnification used for intra-fraction motion correction experiment [micrographs - multiframe] Data #2: Falcon4 EER movies of human light chain apoferritin at 47000 nominal magnification used for super-resolution experiment [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22349.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22349.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_22349_msk_1.map emd_22349_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_22349_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_22349_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human light chain apoferritin
| 全体 | 名称: Human light chain apoferritin |
|---|---|
| 要素 |
|
-超分子 #1: Human light chain apoferritin
| 超分子 | 名称: Human light chain apoferritin / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293F Homo sapiens (ヒト) / 組換細胞: HEK293F |
| 分子量 | 理論値: 480 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 10 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Homemade / 材質: COPPER/RHODIUM / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 30.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 295 K / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 325 / 平均露光時間: 60.0 sec. / 平均電子線量: 41.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 1.6 µm / 最小 デフォーカス(補正後): 0.4 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)