+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1586 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human Adenovirus type 2 ts1 mutant | |||||||||
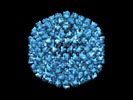
 マップデータ マップデータ | Human adenovirus type 5 at 8.9 Angstrom resolution, showing the structure of the mature, infective virion | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Human adenovirus 2 (ヒトアデノウイルス) Human adenovirus 2 (ヒトアデノウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 8.7 Å | |||||||||
 データ登録者 データ登録者 | Perez-Berna AJ / Marabini R / Scheres SHW / Menendez-Conejero R / Dmitriev IP / Curiel DT / Mangel WF / Flint SJ / San Martin C | |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2009 ジャーナル: J Mol Biol / 年: 2009タイトル: Structure and uncoating of immature adenovirus. 著者: Ana J Pérez-Berná / Roberto Marabini / Sjors H W Scheres / Rosa Menéndez-Conejero / Igor P Dmitriev / David T Curiel / Walter F Mangel / S Jane Flint / Carmen San Martín /  要旨: Maturation via proteolytic processing is a common trait in the viral world and is often accompanied by large conformational changes and rearrangements in the capsid. The adenovirus protease has been ...Maturation via proteolytic processing is a common trait in the viral world and is often accompanied by large conformational changes and rearrangements in the capsid. The adenovirus protease has been shown to play a dual role in the viral infectious cycle: (a) in maturation, as viral assembly starts with precursors to several of the structural proteins but ends with proteolytically processed versions in the mature virion, and (b) in entry, because protease-impaired viruses have difficulties in endosome escape and uncoating. Indeed, viruses that have not undergone proteolytic processing are not infectious. We studied the three-dimensional structure of immature adenovirus particles as represented by the adenovirus type 2 thermosensitive mutant ts1 grown under non-permissive conditions and compared it with the mature capsid. Our three-dimensional electron microscopy maps at subnanometer resolution indicate that adenovirus maturation does not involve large-scale conformational changes in the capsid. Difference maps reveal the locations of unprocessed peptides pIIIa and pVI and help define their role in capsid assembly and maturation. An intriguing difference appears in the core, indicating a more compact organization and increased stability of the immature cores. We have further investigated these properties by in vitro disassembly assays. Fluorescence and electron microscopy experiments reveal differences in the stability and uncoating of immature viruses, both at the capsid and core levels, as well as disassembly intermediates not previously imaged. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1586.map.gz emd_1586.map.gz | 199.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1586-v30.xml emd-1586-v30.xml emd-1586.xml emd-1586.xml | 9.1 KB 9.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1586.png 1586.png | 163.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1586 http://ftp.pdbj.org/pub/emdb/structures/EMD-1586 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1586 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1586 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1586_validation.pdf.gz emd_1586_validation.pdf.gz | 327.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1586_full_validation.pdf.gz emd_1586_full_validation.pdf.gz | 326.5 KB | 表示 | |
| XML形式データ |  emd_1586_validation.xml.gz emd_1586_validation.xml.gz | 7.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1586 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1586 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1586 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1586 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1586.map.gz / 形式: CCP4 / 大きさ: 253 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1586.map.gz / 形式: CCP4 / 大きさ: 253 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human adenovirus type 5 at 8.9 Angstrom resolution, showing the structure of the mature, infective virion | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.76 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
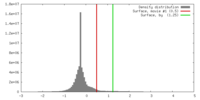
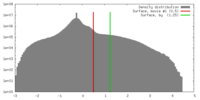
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human adenovirus type 2, ts1 mutant produced at 39 degrees
| 全体 | 名称: Human adenovirus type 2, ts1 mutant produced at 39 degrees |
|---|---|
| 要素 |
|
-超分子 #1000: Human adenovirus type 2, ts1 mutant produced at 39 degrees
| 超分子 | 名称: Human adenovirus type 2, ts1 mutant produced at 39 degrees タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|
-超分子 #1: Human adenovirus 2
| 超分子 | 名称: Human adenovirus 2 / タイプ: virus / ID: 1 / Name.synonym: Adenovirus / NCBI-ID: 10515 / 生物種: Human adenovirus 2 / ウイルスタイプ: VIRION / ウイルス・単離状態: SEROTYPE / ウイルス・エンベロープ: No / ウイルス・中空状態: No / Syn species name: Adenovirus |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) / 別称: VERTEBRATES Homo sapiens (ヒト) / 別称: VERTEBRATES |
| ウイルス殻 | Shell ID: 1 / 直径: 950 Å / T番号(三角分割数): 25 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 詳細: PBS (8 mM Na2HPO4, 2 mM KH2PO4, 137 mM NaCl, 2.7 mM KCl) |
|---|---|
| グリッド | 詳細: Quantifoil R2/4 |
| 凍結 | 凍結剤: ETHANE / 装置: LEICA EM CPC / 詳細: Vitrification instrument: Leica CPC |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 297 / 平均電子線量: 10 e/Å2 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.26 mm / 最大 デフォーカス(公称値): 5.1 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Phase flip in micrograph |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 8.7 Å / 解像度の算出法: FSC 0.33 CUT-OFF / ソフトウェア - 名称: xmipp, ctffind / 使用した粒子像数: 9188 |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称: URO |
| 詳細 | PDBEntryID_givenInChain. Protocol: Rigid Body |
| 精密化 | 空間: RECIPROCAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)