+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
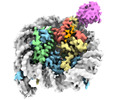
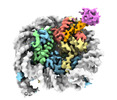
| タイトル | Cryo-EM structure of the human nucleosome core particle in complex with RNF168 and UbcH5c~Ub (UbcH5c chemically conjugated to histone H2A) (class 5) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Nucleosome core particle / chromatin / RNF168 / RING domain / UbcH5c / DNA repair / DNA double-strand break / Homologous recombination / 53BP1 / ubiquitin / STRUCTURAL PROTEIN-DNA-TRANSFERASE complex / TRANSFERASE / TRANSFERASE-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報histone H2AK15 ubiquitin ligase activity / histone ubiquitin ligase activity / (E3-independent) E2 ubiquitin-conjugating enzyme / Signaling by BMP / protein K6-linked ubiquitination / double-strand break repair via classical nonhomologous end joining / isotype switching / protein K11-linked ubiquitination / hypothalamus gonadotrophin-releasing hormone neuron development / female meiosis I ...histone H2AK15 ubiquitin ligase activity / histone ubiquitin ligase activity / (E3-independent) E2 ubiquitin-conjugating enzyme / Signaling by BMP / protein K6-linked ubiquitination / double-strand break repair via classical nonhomologous end joining / isotype switching / protein K11-linked ubiquitination / hypothalamus gonadotrophin-releasing hormone neuron development / female meiosis I / positive regulation of protein monoubiquitination / DNA repair-dependent chromatin remodeling / mitochondrion transport along microtubule / positive regulation of protein targeting to mitochondrion / fat pad development / K63-linked polyubiquitin modification-dependent protein binding / E2 ubiquitin-conjugating enzyme / response to ionizing radiation / female gonad development / seminiferous tubule development / male meiosis I / negative regulation of transcription elongation by RNA polymerase II / protein monoubiquitination / positive regulation of intrinsic apoptotic signaling pathway by p53 class mediator / ubiquitin conjugating enzyme activity / protein K63-linked ubiquitination / negative regulation of BMP signaling pathway / negative regulation of tumor necrosis factor-mediated signaling pathway / negative regulation of megakaryocyte differentiation / protein localization to CENP-A containing chromatin / protein autoubiquitination / protein K48-linked ubiquitination / Chromatin modifying enzymes / Replacement of protamines by nucleosomes in the male pronucleus / CENP-A containing nucleosome / interstrand cross-link repair / SUMOylation of DNA damage response and repair proteins / energy homeostasis / regulation of neuron apoptotic process / heterochromatin organization / epigenetic regulation of gene expression / Packaging Of Telomere Ends / regulation of proteasomal protein catabolic process / ubiquitin ligase complex / Recognition and association of DNA glycosylase with site containing an affected purine / Cleavage of the damaged purine / nucleosome binding / Maturation of protein E / Deposition of new CENPA-containing nucleosomes at the centromere / Maturation of protein E / nucleosomal DNA binding / ER Quality Control Compartment (ERQC) / Myoclonic epilepsy of Lafora / FLT3 signaling by CBL mutants / Prevention of phagosomal-lysosomal fusion / IRAK2 mediated activation of TAK1 complex / Alpha-protein kinase 1 signaling pathway / Glycogen synthesis / Recognition and association of DNA glycosylase with site containing an affected pyrimidine / Cleavage of the damaged pyrimidine / IRAK1 recruits IKK complex / IRAK1 recruits IKK complex upon TLR7/8 or 9 stimulation / Membrane binding and targetting of GAG proteins / Endosomal Sorting Complex Required For Transport (ESCRT) / IRAK2 mediated activation of TAK1 complex upon TLR7/8 or 9 stimulation / PTK6 Regulates RTKs and Their Effectors AKT1 and DOK1 / Negative regulation of FLT3 / Constitutive Signaling by NOTCH1 HD Domain Mutants / Regulation of FZD by ubiquitination / TICAM1,TRAF6-dependent induction of TAK1 complex / NOTCH2 Activation and Transmission of Signal to the Nucleus / TICAM1-dependent activation of IRF3/IRF7 / Inhibition of DNA recombination at telomere / Meiotic synapsis / APC/C:Cdc20 mediated degradation of Cyclin B / p75NTR recruits signalling complexes / positive regulation of DNA repair / Downregulation of ERBB4 signaling / telomere organization / APC-Cdc20 mediated degradation of Nek2A / PINK1-PRKN Mediated Mitophagy / TRAF6-mediated induction of TAK1 complex within TLR4 complex / TRAF6 mediated IRF7 activation in TLR7/8 or 9 signaling / Pexophagy / Regulation of innate immune responses to cytosolic DNA / InlA-mediated entry of Listeria monocytogenes into host cells / VLDLR internalisation and degradation / RNA Polymerase I Promoter Opening / Interleukin-7 signaling / Downregulation of ERBB2:ERBB3 signaling / NF-kB is activated and signals survival / NRIF signals cell death from the nucleus / Regulation of PTEN localization / Assembly of the ORC complex at the origin of replication / Activated NOTCH1 Transmits Signal to the Nucleus / SUMOylation of chromatin organization proteins / Regulation of BACH1 activity / Translesion synthesis by REV1 / Synthesis of active ubiquitin: roles of E1 and E2 enzymes / Translesion synthesis by POLK 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Hu Q / Botuyan MV / Zhao D / Cui G / Mer G | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2024 ジャーナル: Mol Cell / 年: 2024タイトル: Mechanisms of RNF168 nucleosome recognition and ubiquitylation. 著者: Qi Hu / Debiao Zhao / Gaofeng Cui / Janarjan Bhandari / James R Thompson / Maria Victoria Botuyan / Georges Mer /  要旨: RNF168 plays a central role in the DNA damage response (DDR) by ubiquitylating histone H2A at K13 and K15. These modifications direct BRCA1-BARD1 and 53BP1 foci formation in chromatin, essential for ...RNF168 plays a central role in the DNA damage response (DDR) by ubiquitylating histone H2A at K13 and K15. These modifications direct BRCA1-BARD1 and 53BP1 foci formation in chromatin, essential for cell-cycle-dependent DNA double-strand break (DSB) repair pathway selection. The mechanism by which RNF168 catalyzes the targeted accumulation of H2A ubiquitin conjugates to form repair foci around DSBs remains unclear. Here, using cryoelectron microscopy (cryo-EM), nuclear magnetic resonance (NMR) spectroscopy, and functional assays, we provide a molecular description of the reaction cycle and dynamics of RNF168 as it modifies the nucleosome and recognizes its ubiquitylation products. We demonstrate an interaction of a canonical ubiquitin-binding domain within full-length RNF168, which not only engages ubiquitin but also the nucleosome surface, clarifying how such site-specific ubiquitin recognition propels a signal amplification loop. Beyond offering mechanistic insights into a key DDR protein, our study aids in understanding site specificity in both generating and interpreting chromatin ubiquitylation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40615.map.gz emd_40615.map.gz | 38.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40615-v30.xml emd-40615-v30.xml emd-40615.xml emd-40615.xml | 27.9 KB 27.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_40615.png emd_40615.png | 108.5 KB | ||
| マスクデータ |  emd_40615_msk_1.map emd_40615_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-40615.cif.gz emd-40615.cif.gz | 7.7 KB | ||
| その他 |  emd_40615_half_map_1.map.gz emd_40615_half_map_1.map.gz emd_40615_half_map_2.map.gz emd_40615_half_map_2.map.gz | 50.1 MB 50.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40615 http://ftp.pdbj.org/pub/emdb/structures/EMD-40615 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40615 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40615 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40615_validation.pdf.gz emd_40615_validation.pdf.gz | 910.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40615_full_validation.pdf.gz emd_40615_full_validation.pdf.gz | 910.5 KB | 表示 | |
| XML形式データ |  emd_40615_validation.xml.gz emd_40615_validation.xml.gz | 12 KB | 表示 | |
| CIF形式データ |  emd_40615_validation.cif.gz emd_40615_validation.cif.gz | 14 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40615 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40615 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40615 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40615 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8sn7MC  8smwC  8smxC  8smyC  8smzC  8sn0C  8sn1C  8sn2C  8sn3C  8sn4C  8sn5C  8sn6C  8sn8C  8sn9C  8snaC  8txvC  8txwC  8txxC  8u13C  8u14C  8upfC  8uq8C  8uq9C  8uqaC  8uqbC  8uqcC  8uqdC  8uqeC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40615.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40615.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.328 Å | ||||||||||||||||||||
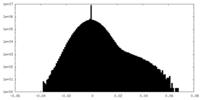
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_40615_msk_1.map emd_40615_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
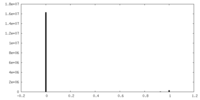
| 投影像・断面図 |
| ||||||||||||
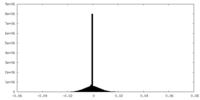
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_40615_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
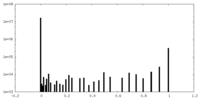
| 投影像・断面図 |
| ||||||||||||
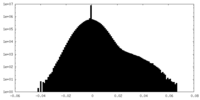
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_40615_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
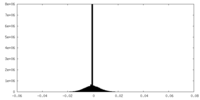
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Human nucleosome core particle in complex with RNF168 and UbcH5c~Ub
+超分子 #1: Human nucleosome core particle in complex with RNF168 and UbcH5c~Ub
+分子 #1: Histone H3.1
+分子 #2: Histone H4
+分子 #3: Histone H2A type 1-B/E
+分子 #4: Histone H2B type 1-J
+分子 #7: E3 ubiquitin-protein ligase RNF168
+分子 #8: Ubiquitin-conjugating enzyme E2 D3
+分子 #9: Polyubiquitin-B
+分子 #5: DNA (147-MER)
+分子 #6: DNA (147-MER)
+分子 #10: ZINC ION
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | 10 mM HEPES, 100 mM NaCl, 1 mM DTT, pH 7.5 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 14179 / 平均電子線量: 60.0 e/Å2 詳細: 14179 images were recorded in movie-mode of which 14113 were retained for particle picking. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT | ||||||||||
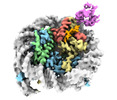
| 得られたモデル |  PDB-8sn7: |
 ムービー
ムービー コントローラー
コントローラー














































 Z
Z Y
Y X
X