+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4633 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of SH1 full particle. | |||||||||||||||
 マップデータ マップデータ | Block-based reconstruction of SH1 capsid, filtered at 3.8 Angstrom and sharpened with a B-factor=-90 | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | euryarcheal virus / SH1 / VIRUS | |||||||||||||||
| 機能・相同性 | ORF 31 / ORF 25 / ORF 24 機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |  Haloarcula hispanica virus SH1 (ウイルス) Haloarcula hispanica virus SH1 (ウイルス) | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||||||||
 データ登録者 データ登録者 | De Colibus L / Roine E / Walter TS / Ilca SL | |||||||||||||||
| 資金援助 |  英国, 英国,  フィンランド, 4件 フィンランド, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: Assembly of complex viruses exemplified by a halophilic euryarchaeal virus. 著者: Luigi De Colibus / Elina Roine / Thomas S Walter / Serban L Ilca / Xiangxi Wang / Nan Wang / Alan M Roseman / Dennis Bamford / Juha T Huiskonen / David I Stuart /    要旨: Many of the largest known viruses belong to the PRD1-adeno structural lineage characterised by conserved pseudo-hexameric capsomers composed of three copies of a single major capsid protein (MCP). ...Many of the largest known viruses belong to the PRD1-adeno structural lineage characterised by conserved pseudo-hexameric capsomers composed of three copies of a single major capsid protein (MCP). Here, by high-resolution cryo-EM analysis, we show that a class of archaeal viruses possess hetero-hexameric MCPs which mimic the PRD1-adeno lineage trimer. These hetero-hexamers are built from heterodimers and utilise a jigsaw-puzzle system of pegs and holes, and underlying minor capsid proteins, to assemble the capsid laterally from the 5-fold vertices. At these vertices proteins engage inwards with the internal membrane vesicle whilst 2-fold symmetric horn-like structures protrude outwards. The horns are assembled from repeated globular domains attached to a central spine, presumably facilitating multimeric attachment to the cell receptor. Such viruses may represent precursors of the main PRD1-adeno lineage, similarly engaging cell-receptors via 5-fold spikes and using minor proteins to define particle size. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4633.map.gz emd_4633.map.gz | 530.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4633-v30.xml emd-4633-v30.xml emd-4633.xml emd-4633.xml | 29.7 KB 29.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_4633.png emd_4633.png | 242.8 KB | ||
| Filedesc metadata |  emd-4633.cif.gz emd-4633.cif.gz | 6.9 KB | ||
| その他 |  emd_4633_additional_1.map.gz emd_4633_additional_1.map.gz emd_4633_additional_2.map.gz emd_4633_additional_2.map.gz emd_4633_half_map_1.map.gz emd_4633_half_map_1.map.gz emd_4633_half_map_2.map.gz emd_4633_half_map_2.map.gz | 8.5 MB 7.9 MB 530.5 MB 530.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4633 http://ftp.pdbj.org/pub/emdb/structures/EMD-4633 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4633 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4633 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4633.map.gz / 形式: CCP4 / 大きさ: 1.9 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4633.map.gz / 形式: CCP4 / 大きさ: 1.9 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Block-based reconstruction of SH1 capsid, filtered at 3.8 Angstrom and sharpened with a B-factor=-90 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Sharpened and non-icosahedrally averaged map of VP7 protein
| ファイル | emd_4633_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened and non-icosahedrally averaged map of VP7 protein | ||||||||||||
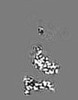
| 投影像・断面図 |
| ||||||||||||
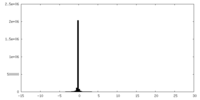
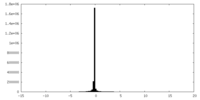
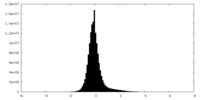
| 密度ヒストグラム |
-追加マップ: Sharpened and non-icosahedrally averaged map of VP4 protein
| ファイル | emd_4633_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened and non-icosahedrally averaged map of VP4 protein | ||||||||||||
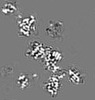
| 投影像・断面図 |
| ||||||||||||
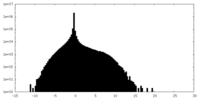
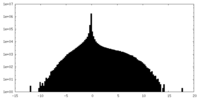
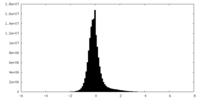
| 密度ヒストグラム |
-ハーフマップ: Block-based reconstruction of SH1 capsid, halfmap 2
| ファイル | emd_4633_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Block-based reconstruction of SH1 capsid, halfmap 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
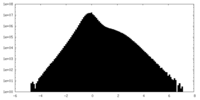
| 密度ヒストグラム |
-ハーフマップ: Block-based reconstruction of SH1 capsid. halfmap 1
| ファイル | emd_4633_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Block-based reconstruction of SH1 capsid. halfmap 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Haloarcula hispanica virus SH1
| 全体 | 名称:  Haloarcula hispanica virus SH1 (ウイルス) Haloarcula hispanica virus SH1 (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Haloarcula hispanica virus SH1
| 超分子 | 名称: Haloarcula hispanica virus SH1 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 326574 / 生物種: Haloarcula hispanica virus SH1 / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Haloarcula hispanica virus SH1 (ウイルス) Haloarcula hispanica virus SH1 (ウイルス) |
| ウイルス殻 | Shell ID: 1 / 名称: Capsid / 直径: 1000.0 Å / T番号(三角分割数): 28 |
-分子 #1: ORF 25
| 分子 | 名称: ORF 25 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haloarcula hispanica virus SH1 (ウイルス) Haloarcula hispanica virus SH1 (ウイルス) |
| 分子量 | 理論値: 25.042207 KDa |
| 配列 | 文字列: EYTISHTGGT LGSSKVTTAA NQTSPQRETA IIGFECPRKF AEIEYVGQRD STRFIPRTTE SITGTAGDDT VVSLTANIQP VAGETAIED QDYPVAVAYN VTQGVQVDID AVDYAADEVT LADNPADGDT VKVWPIMGDG DVQFRLVNQF GQEEGRVYPW A TPLYRWHD ...文字列: EYTISHTGGT LGSSKVTTAA NQTSPQRETA IIGFECPRKF AEIEYVGQRD STRFIPRTTE SITGTAGDDT VVSLTANIQP VAGETAIED QDYPVAVAYN VTQGVQVDID AVDYAADEVT LADNPADGDT VKVWPIMGDG DVQFRLVNQF GQEEGRVYPW A TPLYRWHD FPQLKRGREI NLHGSVTWEE NETVEVLLDA PQAITWEDSD YPEGQYVSTF EQDVEITL UniProtKB: ORF 25 |
-分子 #2: ORF 25
| 分子 | 名称: ORF 25 / タイプ: protein_or_peptide / ID: 2 / コピー数: 10 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haloarcula hispanica virus SH1 (ウイルス) Haloarcula hispanica virus SH1 (ウイルス) |
| 分子量 | 理論値: 24.913094 KDa |
| 配列 | 文字列: YTISHTGGTL GSSKVTTAAN QTSPQRETAI IGFECPRKFA EIEYVGQRDS TRFIPRTTES ITGTAGDDTV VSLTANIQPV AGETAIEDQ DYPVAVAYNV TQGVQVDIDA VDYAADEVTL ADNPADGDTV KVWPIMGDGD VQFRLVNQFG QEEGRVYPWA T PLYRWHDF ...文字列: YTISHTGGTL GSSKVTTAAN QTSPQRETAI IGFECPRKFA EIEYVGQRDS TRFIPRTTES ITGTAGDDTV VSLTANIQPV AGETAIEDQ DYPVAVAYNV TQGVQVDIDA VDYAADEVTL ADNPADGDTV KVWPIMGDGD VQFRLVNQFG QEEGRVYPWA T PLYRWHDF PQLKRGREIN LHGSVTWEEN ETVEVLLDAP QAITWEDSDY PEGQYVSTFE QDVEITL UniProtKB: ORF 25 |
-分子 #3: ORF 24
| 分子 | 名称: ORF 24 / タイプ: protein_or_peptide / ID: 3 / コピー数: 11 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haloarcula hispanica virus SH1 (ウイルス) Haloarcula hispanica virus SH1 (ウイルス) |
| 分子量 | 理論値: 18.840627 KDa |
| 配列 | 文字列: GNIGNLSAEK QISVYDGQPF VDEQDVPADD PNTPALTIEG PDGYVIAVDA GTPIAPEFRD SNGNKLDPST RVIVQKCDRQ GNPLGDGIV FNDTLGRFDY EQMRTDPDFM RKTAKSLMID EREIVKVFVD IPAGANGYDA DKSRLTLGDD TSDFGKAVEI V DHDELSDA ETRAV UniProtKB: ORF 24 |
-分子 #4: ORF 24
| 分子 | 名称: ORF 24 / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haloarcula hispanica virus SH1 (ウイルス) Haloarcula hispanica virus SH1 (ウイルス) |
| 分子量 | 理論値: 18.272 KDa |
| 配列 | 文字列: SAEKQISVYD GQPFVDEQDV PADDPNTPAL TIEGPDGYVI AVDAGTPIAP EFRDSNGNKL DPSTRVIVQK CDRQGNPLGD GIVFNDTLG RFDYEQMRTD PDFMRKTAKS LMIDEREIVK VFVDIPAGAN GYDADKSRLT LGDDTSDFGK AVEIVDHDEL S DAETRAV UniProtKB: ORF 24 |
-分子 #5: ORF 24
| 分子 | 名称: ORF 24 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haloarcula hispanica virus SH1 (ウイルス) Haloarcula hispanica virus SH1 (ウイルス) |
| 分子量 | 理論値: 19.040887 KDa |
| 配列 | 文字列: GNIGNLSAEK QISVYDGQPF VDEQDVPADD PNTPALTIEG PDGYVIAVDA GTPIAPEFRD SNGNKLDPST RVIVQKCDRQ GNPLGDGIV FNDTLGRFDY EQMRTDPDFM RKTAKSLMID EREIVKVFVD IPAGANGYDA DKSRLTLGDD TSDFGKAVEI V DHDELSDA ETRAVKA UniProtKB: ORF 24 |
-分子 #6: ORF 31
| 分子 | 名称: ORF 31 / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haloarcula hispanica virus SH1 (ウイルス) Haloarcula hispanica virus SH1 (ウイルス) |
| 分子量 | 理論値: 14.982346 KDa |
| 配列 | 文字列: ERLGRLVDVL ETKEFGDTTV ERSVTQNIDR TRTDSPNNEN QPIYFSTGPE AIAVENTEEW ERLDFGIVAE TVNIRTTDDI DIAFADPNK NGPVIRVREG ESPFTIGGDA GIESAFIWLR QAETASNTPG IQIIAF UniProtKB: ORF 31 |
-分子 #7: VP12
| 分子 | 名称: VP12 / タイプ: protein_or_peptide / ID: 7 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haloarcula hispanica virus SH1 (ウイルス) Haloarcula hispanica virus SH1 (ウイルス) |
| 分子量 | 理論値: 2.060531 KDa |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) |
-分子 #8: VP13
| 分子 | 名称: VP13 / タイプ: protein_or_peptide / ID: 8 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haloarcula hispanica virus SH1 (ウイルス) Haloarcula hispanica virus SH1 (ウイルス) |
| 分子量 | 理論値: 6.826406 KDa |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) ...文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 15 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 / 詳細: 20mM Tris-HCl pH 7.2, 1M NaCl, 10 mM MnCl2 |
| グリッド | モデル: C-flat-2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 支持フィルム - Film thickness: 20 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.027 kPa |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 4 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均露光時間: 4.4 sec. / 平均電子線量: 1.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 37037 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 倍率(公称値): 160000 |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: BACKBONE TRACE / 温度因子: 144.281 当てはまり具合の基準: Cross-correlation coefficient |
|---|---|
| 得られたモデル |  PDB-6qt9: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)