+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1cfv | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | MONOCLONAL ANTIBODY FRAGMENT FV4155 FROM E. COLI | ||||||
 要素 要素 | (MONOCLONAL ANTIBODY FV4155) x 2 | ||||||
 キーワード キーワード | IMMUNOGLOBULIN / FV FRAGMENT / STEROID HORMONE / FINE SPECIFICITY | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of B cell activation / phagocytosis, recognition / immunoglobulin complex / phagocytosis, engulfment / immunoglobulin complex, circulating / immunoglobulin receptor binding / complement activation, classical pathway / antigen binding / B cell receptor signaling pathway / adaptive immune response ...positive regulation of B cell activation / phagocytosis, recognition / immunoglobulin complex / phagocytosis, engulfment / immunoglobulin complex, circulating / immunoglobulin receptor binding / complement activation, classical pathway / antigen binding / B cell receptor signaling pathway / adaptive immune response / defense response to bacterium / immune response / external side of plasma membrane / innate immune response / extracellular space 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.1 Å 分子置換 / 解像度: 2.1 Å | ||||||
 データ登録者 データ登録者 | Trinh, C.H. / Phillips, S.E.V. | ||||||
 引用 引用 |  ジャーナル: Structure / 年: 1997 ジャーナル: Structure / 年: 1997タイトル: Antibody fragment Fv4155 bound to two closely related steroid hormones: the structural basis of fine specificity. 著者: Trinh, C.H. / Hemmington, S.D. / Verhoeyen, M.E. / Phillips, S.E. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1cfv.cif.gz 1cfv.cif.gz | 61.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1cfv.ent.gz pdb1cfv.ent.gz | 46.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1cfv.json.gz 1cfv.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1cfv_validation.pdf.gz 1cfv_validation.pdf.gz | 748.8 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1cfv_full_validation.pdf.gz 1cfv_full_validation.pdf.gz | 757.2 KB | 表示 | |
| XML形式データ |  1cfv_validation.xml.gz 1cfv_validation.xml.gz | 14.2 KB | 表示 | |
| CIF形式データ |  1cfv_validation.cif.gz 1cfv_validation.cif.gz | 19.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/cf/1cfv https://data.pdbj.org/pub/pdb/validation_reports/cf/1cfv ftp://data.pdbj.org/pub/pdb/validation_reports/cf/1cfv ftp://data.pdbj.org/pub/pdb/validation_reports/cf/1cfv | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||
| 単位格子 |
| ||||||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: 抗体 | 分子量: 12437.998 Da / 分子数: 1 / 断片: FV FRAGMENT / 由来タイプ: 組換発現 / 由来: (組換発現)   | ||||
|---|---|---|---|---|---|
| #2: 抗体 | 分子量: 13191.760 Da / 分子数: 1 / 断片: FV FRAGMENT / 由来タイプ: 組換発現 / 由来: (組換発現)   | ||||
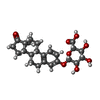
| #3: 化合物 | | #4: 化合物 | ChemComp-E3G / | #5: 水 | ChemComp-HOH / | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.36 Å3/Da / 溶媒含有率: 48 % | ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | pH: 7 詳細: PROTEIN WAS CRYSTALLIZED FROM 18% (W/V) PEG 8000, 200MM ZN ACETATE AND 100MM NA CACODYLATE, PH 7.0 | ||||||||||||||||||||||||||||||||||||||||
| 結晶 | *PLUS 溶媒含有率: 48 % | ||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 手法: 蒸気拡散法, ハンギングドロップ法 | ||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU RUH2R / 波長: 1.5418 回転陽極 / タイプ: RIGAKU RUH2R / 波長: 1.5418 |
| 検出器 | タイプ: RIGAKU RAXIS IIC / 検出器: IMAGE PLATE / 日付: 1996年1月1日 / 詳細: MSC/YALE MIRRORS |
| 放射 | モノクロメーター: NI FILTER / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 2.1→20 Å / Num. obs: 14295 / % possible obs: 97.1 % / Observed criterion σ(I): 3 / 冗長度: 7.4 % / Rsym value: 0.064 / Net I/σ(I): 9.5 |
| 反射 シェル | 解像度: 2.1→2.17 Å / 冗長度: 7.2 % / Mean I/σ(I) obs: 2.8 / Rsym value: 0.268 / % possible all: 98.9 |
| 反射 | *PLUS Num. measured all: 105121 / Rmerge(I) obs: 0.077 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 1NBV 解像度: 2.1→10 Å / 交差検証法: THROUGHOUT / σ(F): 0 詳細: OTHER REFINEMENT REMARKS: CONTACTS INVOLVING ZINC ATOMS CAN BE AT DISTANCES LESS THAN THE SPECIFIED DISTANCE CUTOFF OF 2.2 ANGSTROMS FOR CONTACTS NOT NOT INVOLVING HYDROGEN ATOMS. THE ...詳細: OTHER REFINEMENT REMARKS: CONTACTS INVOLVING ZINC ATOMS CAN BE AT DISTANCES LESS THAN THE SPECIFIED DISTANCE CUTOFF OF 2.2 ANGSTROMS FOR CONTACTS NOT NOT INVOLVING HYDROGEN ATOMS. THE FOLLOWING ATOMS ARE SITUATED ACROSS A TWO-FOLD AXIS AND HAVE AN OCCUPANCY OF 0.5. IT IS THEREFORE POSSIBLE THAT THESE ATOMS ARE IN CLOSE CONTACT WITH OTHER ATOMS AT DISTANCES LESS THAN THOSE NORMALLY OBSERVED. ATM1 RES C SSEQI ZN ZN L 201 O HOH L 300 O HOH L 303 O HOH L 389 RESIDUE VAL L 56 HAS DIHEDRAL ANGLES WHICH LIE OUTSIDE THEIR EXPECTED RANGE IN THE GAMMA QUADRANT OF THE RAMACHANDRAN PLOT. SOLVENT MOLECULES HOH L 300, HOH L 301 AND HOH L 394 ARE SITUATED ON SPECIAL POSITIONS AND HAVE OCCUPANCIES OF 0.50. RESIDUE VAL L 56 HAS DIHEDRAL ANGLES WHICH LIE OUTSIDE THEIR EXPECTED RANGE IN THE GAMMA QUADRANT OF THE RAMACHANDRAN PLOT.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 19.76 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.1→10 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: PROLSQ / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS Rfactor all: 0.178 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS |
 ムービー
ムービー コントローラー
コントローラー
























 PDBj
PDBj