+検索条件
-Structure paper
| タイトル | Mechanisms of RNF168 nucleosome recognition and ubiquitylation. |
|---|---|
| ジャーナル・号・ページ | Mol Cell, Vol. 84, Issue 5, Page 839-853.e12, Year 2024 |
| 掲載日 | 2024年3月7日 |
 著者 著者 | Qi Hu / Debiao Zhao / Gaofeng Cui / Janarjan Bhandari / James R Thompson / Maria Victoria Botuyan / Georges Mer /  |
| PubMed 要旨 | RNF168 plays a central role in the DNA damage response (DDR) by ubiquitylating histone H2A at K13 and K15. These modifications direct BRCA1-BARD1 and 53BP1 foci formation in chromatin, essential for ...RNF168 plays a central role in the DNA damage response (DDR) by ubiquitylating histone H2A at K13 and K15. These modifications direct BRCA1-BARD1 and 53BP1 foci formation in chromatin, essential for cell-cycle-dependent DNA double-strand break (DSB) repair pathway selection. The mechanism by which RNF168 catalyzes the targeted accumulation of H2A ubiquitin conjugates to form repair foci around DSBs remains unclear. Here, using cryoelectron microscopy (cryo-EM), nuclear magnetic resonance (NMR) spectroscopy, and functional assays, we provide a molecular description of the reaction cycle and dynamics of RNF168 as it modifies the nucleosome and recognizes its ubiquitylation products. We demonstrate an interaction of a canonical ubiquitin-binding domain within full-length RNF168, which not only engages ubiquitin but also the nucleosome surface, clarifying how such site-specific ubiquitin recognition propels a signal amplification loop. Beyond offering mechanistic insights into a key DDR protein, our study aids in understanding site specificity in both generating and interpreting chromatin ubiquitylation. |
 リンク リンク |  Mol Cell / Mol Cell /  PubMed:38242129 / PubMed:38242129 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 2.049 - 4.0 Å |
| 構造データ | EMDB-40604, PDB-8smw: EMDB-40605, PDB-8smx: EMDB-40606, PDB-8smy: EMDB-40607, PDB-8smz: EMDB-40608, PDB-8sn0: EMDB-40609, PDB-8sn1: EMDB-40610, PDB-8sn2: EMDB-40611, PDB-8sn3: EMDB-40612, PDB-8sn4: EMDB-40613, PDB-8sn5: EMDB-40614, PDB-8sn6: EMDB-40615, PDB-8sn7: EMDB-40616, PDB-8sn8: EMDB-40617, PDB-8sn9: EMDB-40618, PDB-8sna: EMDB-41706, PDB-8txv: EMDB-41707, PDB-8txw: EMDB-41708, PDB-8txx: EMDB-41800, PDB-8u13: EMDB-41801, PDB-8u14: EMDB-42446, PDB-8upf:  PDB-8uq8:  PDB-8uq9:  PDB-8uqa:  PDB-8uqb:  PDB-8uqc:  PDB-8uqd:  PDB-8uqe: |
| 化合物 |  ChemComp-ZN:  ChemComp-CL:  ChemComp-GOL:  ChemComp-NA:  ChemComp-HOH: |
| 由来 |
|
 キーワード キーワード | STRUCTURAL PROTEIN/DNA/TRANSFERASE / Nucleosome core particle / chromatin / RNF168 / RING domain / UbcH5c / DNA repair / DNA double-strand break / Homologous recombination / 53BP1 / ubiquitin / STRUCTURAL PROTEIN-DNA-TRANSFERASE complex / TRANSFERASE/DNA / TRANSFERASE / TRANSFERASE-DNA complex / MIU2-LRM domains / BRCA1-BARD1 / Histone H2A / Histone H2B / Ubiquitin ligase / Ubiquitin-conjugating enzyme / DNA damage response / DNA double-strand break repair / PROTEIN BINDING / PROTEIN BINDING-Transferase complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について

































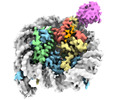

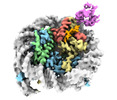

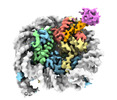







 homo sapiens (ヒト)
homo sapiens (ヒト)