-検索条件
-検索結果
検索 (著者・登録者: shao & zh)の結果906件中、1から50件目までを表示しています

EMDB-35871: 
Cryo-EM map of Gi-scFv16 complex

EMDB-35870: 
Cryo-EM map of human GPR34

EMDB-35906: 
cryo-EM structure of human EMC
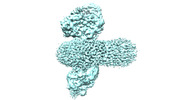
EMDB-35907: 
cryo-EM structure of human EMC and VDAC

PDB-8j0n: 
cryo-EM structure of human EMC

PDB-8j0o: 
cryo-EM structure of human EMC and VDAC

EMDB-38795: 
Cryo-EM structure of the [Pyr1]-apelin-13-bound human APLNR-Gi complex

PDB-8xzg: 
Cryo-EM structure of the [Pyr1]-apelin-13-bound human APLNR-Gi complex

EMDB-38794: 
Cryo-EM structure of the WN561-bound human APLNR-Gi complex

EMDB-38796: 
Cryo-EM structure of the MM07-bound human APLNR-Gi complex

EMDB-38797: 
Cryo-EM structure of the CMF-019-bound human APLNR-Gi complex

EMDB-38798: 
Cryo-EM structure of the WN353-bound human APLNR-Gi complex

PDB-8xzf: 
Cryo-EM structure of the WN561-bound human APLNR-Gi complex

PDB-8xzh: 
Cryo-EM structure of the MM07-bound human APLNR-Gi complex

PDB-8xzi: 
Cryo-EM structure of the CMF-019-bound human APLNR-Gi complex

PDB-8xzj: 
Cryo-EM structure of the WN353-bound human APLNR-Gi complex

EMDB-35832: 
Cryo-EM structure of the GPR34 receptor in complex with the antagonist YL-365

PDB-8iyx: 
Cryo-EM structure of the GPR34 receptor in complex with the antagonist YL-365

EMDB-35492: 
The cryo-EM structure of human sphingomyelin synthase-related protein in complex with ceramide

EMDB-35493: 
The cryo-EM structure of human sphingomyelin synthase-related protein in complex with diacylglycerol/phosphoethanolamine

EMDB-37383: 
The cryo-EM structure of human sphingomyelin synthase-related protein in complex with ceramide/phosphoethanolamine

EMDB-37385: 
The cryo-EM structure of human sphingomyelin synthase-related protein

PDB-8ijq: 
The cryo-EM structure of human sphingomyelin synthase-related protein in complex with ceramide

PDB-8ijr: 
The cryo-EM structure of human sphingomyelin synthase-related protein in complex with diacylglycerol/phosphoethanolamine

PDB-8w9w: 
The cryo-EM structure of human sphingomyelin synthase-related protein in complex with ceramide/phosphoethanolamine

PDB-8w9y: 
The cryo-EM structure of human sphingomyelin synthase-related protein

EMDB-35514: 
Cryo-EM structure of the inactive CD97

EMDB-38938: 
Cryo-EM structure of the inactive CD97

EMDB-38939: 
Cryo-EM structure of the inactive CD97

EMDB-38980: 
Cryo-EM structure of the inactive CD97

PDB-8ikj: 
Cryo-EM structure of the inactive CD97

EMDB-41725: 
Structural and biochemical rationale for Beta variant protein booster vaccine broad cross-neutralization of SARS-CoV-2

EMDB-41727: 
Structural and biochemical rationale for Beta variant protein booster vaccine broad cross-neutralization of SARS-CoV-2

PDB-8tyl: 
Structural and biochemical rationale for Beta variant protein booster vaccine broad cross-neutralization of SARS-CoV-2

PDB-8tyo: 
Structural and biochemical rationale for Beta variant protein booster vaccine broad cross-neutralization of SARS-CoV-2

EMDB-33990: 
Cryo-EM structure of EBV gHgL-gp42 in complex with mAbs 3E8 and 5E3 (localized refinement)
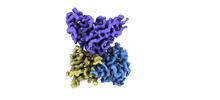
EMDB-33992: 
Cryo-EM structure of EBV gHgL-gp42 in complex with mAb 10E4 (localized refinement)
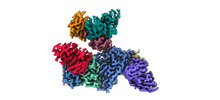
EMDB-33993: 
Cryo-EM density map of EBV gHgL-gp42 in complex with four mAbs 5E3, 3E8, 6H2 and 10E4
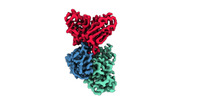
EMDB-33994: 
Cryo-EM structure of EBV gHgL-gp42 in complex with mAb 6H2 (localized refinement)

PDB-7yoy: 
Cryo-EM structure of EBV gHgL-gp42 in complex with mAbs 3E8 and 5E3 (localized refinement)

PDB-7yp1: 
Cryo-EM structure of EBV gHgL-gp42 in complex with mAb 10E4 (localized refinement)

PDB-7yp2: 
Cryo-EM structure of EBV gHgL-gp42 in complex with mAb 6H2 (localized refinement)

EMDB-35516: 
Cryo-EM structure of the CD97-G13 complex

PDB-8ikl: 
Cryo-EM structure of the CD97-G13 complex

EMDB-35940: 
Cryo-EM structure of FFAR3 bound with valeric acid and AR420626

EMDB-35941: 
Cryo-EM structure of FFAR3 complex bound with butyrate acid

EMDB-35942: 
Cryo-EM structure of FFAR2 complex bound with TUG-1375

EMDB-35944: 
Cryo-EM structure of FFAR2 complex bound with acetic acid

PDB-8j20: 
Cryo-EM structure of FFAR3 bound with valeric acid and AR420626

PDB-8j21: 
Cryo-EM structure of FFAR3 complex bound with butyrate acid
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
