-検索条件
-検索結果
検索 (著者・登録者: chen & yh)の結果97件中、1から50件目までを表示しています

EMDB-37604: 
Structural basis of translation inhibition by a valine tRNA-derived fragment

EMDB-37733: 
Structural basis of translation inhibition by a valine tRNA-derived fragment

EMDB-37734: 
Structural basis of translation inhibition by a valine tRNA-derived fragment
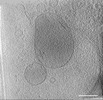
EMDB-39012: 
Representative tomogram of primary glioblastoma stem cell with circular inter-mitochondrial junctions.

EMDB-39015: 
Representative tomogram of microglia cell with nanotunnel-like structures resembling mitochondrial fission.
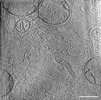
EMDB-39019: 
Representative tomogram of glioblastoma cell with nanotunnel-like structure and inter-mitochondrial junction.

EMDB-39021: 
Representative tomogram of normal human astrocyte with nanotunnel-like structure which is an extension of the mitochondrial outer membrane.

EMDB-39023: 
Representative tomogram of primary glioblastoma differentiated cell with parallel inter-mitochondrial junction.

EMDB-39024: 
Representative tomogram of primary glioblastoma stem cell with clustered mitochondria bearing various long-short axis ratios.
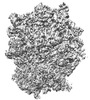
EMDB-34866: 
Cryo-EM Structures and Translocation Mechanism of Crenarchaeota Ribosome

EMDB-34869: 
Cryo-EM Structures and Translocation Mechanism of Crenarchaeota Ribosome

EMDB-35775: 
The rice Na+/H+ antiporter SOS1 in an auto-inhibited state

EMDB-35950: 
The truncated rice Na+/H+ antiporter SOS1 (1-976) in a constitutively active state
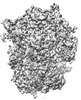
EMDB-34870: 
Cryo-EM Structures and Translocation Mechanism of Crenarchaeota Ribosome
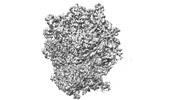
EMDB-34867: 
Cryo-EM Structures and Translocation Mechanism of Crenarchaeota Ribosome

EMDB-34868: 
Cryo-EM Structures and Translocation Mechanism of Crenarchaeota Ribosome

EMDB-34863: 
Cryo-EM Structures and Translocation Mechanism of Crenarchaeota Ribosome

EMDB-34864: 
Cryo-EM Structures and Translocation Mechanism of Crenarchaeota Ribosome

EMDB-34860: 
Cryo-EM Structures and Translocation Mechanism of Crenarchaeota Ribosome

EMDB-34861: 
Cryo-EM Structures and Translocation Mechanism of Crenarchaeota Ribosome

EMDB-34862: 
Cryo-EM Structures and Translocation Mechanism of Crenarchaeota Ribosome

EMDB-34710: 
Cryo-EM structure of WeiTsing

EMDB-33145: 
Cryo-EM structures of human mitochondrial NAD(P)+-dependent malic enzyme in apo form

EMDB-33146: 
Cryo-EM structures of human mitochondrial NAD(P)+-dependent malic enzyme in a ternary complex with NAD+ and allosteric inhibitor EA

EMDB-33147: 
Cryo-EM structures of human mitochondrial NAD(P)+-dependent malic enzyme in a ternary complex with NAD+ and allosteric inhibitor MDSA

EMDB-35522: 
Cryo-EM structure of the TUG891 bound GPR120-Giq complex(mask on receptor)

EMDB-35523: 
Cryo-EM structure of the TUG891 bound GPR120-Giq complex(mask on Giq-scFV16 complex)

EMDB-35524: 
Cryo-EM structure of the eicosapentaenoic acid bound GPR120-Gi1 complex(mask on receptor)

EMDB-35525: 
Cryo-EM structure of the eicosapentaenoic acid bound GPR120-Gi1 complex(mask on Gil-scFV16 complex)

EMDB-35529: 
Cryo-EM structure of the TUG891 bound GPR120-Giq complex (consensus map)

EMDB-35533: 
Cryo-EM structure of the eicosapentaenoic acid bound GPR120-Gi complex(consensus map)

EMDB-35356: 
Cryo-EM structure of the 9-hydroxystearic acid bound GPR120-Gi complex

EMDB-35357: 
Cryo-EM structure of the linoleic acid bound GPR120-Gi complex

EMDB-35358: 
Cryo-EM structure of the oleic acid bound GPR120-Gi complex

EMDB-35359: 
Cryo-EM structure of the TUG891 bound GPR120-Gi complex

EMDB-35360: 
Cryo-EM structure of the eicosapentaenoic acid bound GPR120-Gi complex

EMDB-29736: 
Cryo-EM structure of the TUG891 bound GPR120-Giq complex

EMDB-33923: 
Cryo-EM structure of SARS-CoV-2 Omicron spike glycoprotein in complex with three neutralizing nanobody 3-2A2-4
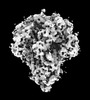
EMDB-32329: 
Cryo-EM map of PEDV (Pintung 52) S protein with all three protomers in the D0-down conformation determined in situ on intact viral particles.

EMDB-32332: 
Subtomogram averaging of PEDV (Pintung 52) S protein with all three protomers in the D0-down conformation determined in situ on intact viral particles.

EMDB-32333: 
Subtomogram averaging of PEDV (Pintung 52) S protein with one protomer in the D0-up conformation and two protomers in the D0-down conformation, determined in situ on intact viral particles

EMDB-32337: 
Subtomogram averaging of PEDV (Pintung 52) S protein with two protomers in the D0-up conformation and one protomer in the D0-down conformation, determined in situ on intact viral particles.

EMDB-32338: 
Cryo-EM map of PEDV S protein with one protomer in the D0-up conformation while the other two in the D0-down conformation
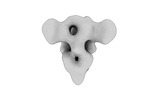
EMDB-32339: 
Subtomogram averaging of PEDV (Pintung 52) S protein with all three protomers in the D0-up conformation determined in situ on intact viral particles.

EMDB-32340: 
Subtomogram averaging of PEDV (Pintung 52) S protein in the postfusion form determined in situ on intact viral particles.

EMDB-33646: 
Cryo-EM map of IPEC-J2 cell-derived PEDV PT52 S protein with three D0-up

EMDB-33647: 
Cryo-EM map of IPEC-J2 cell-derived PEDV PT52 S protein one D0-down and two D0-up

EMDB-33648: 
Symmetry-expanded and locally refined protomer structure of IPEC-J2 cell-derived PEDV PT52 S with a CTD-close conformation
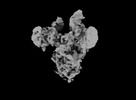
EMDB-33649: 
Symmetry-expanded and locally refined protomer structure of IPEC-J2 cell-derived PEDV PT52 S with a CTD-open conformation

EMDB-33700: 
Cryo-EM map of HEK293F cell-derived PEDV PT52 S protein with three D0-down
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
