-検索条件
-検索結果
検索 (著者・登録者: zhang & hq)の結果60件中、1から50件目までを表示しています

EMDB-35416: 
Cryo-EM structure of the Clr6S (Clr6-HDAC) complex from S. pombe

EMDB-35417: 
Clr6 HDAC (Clr6S)-nucleosome complex

EMDB-36278: 
Rpd3S-nucleosome (H3K36me3 modified)

EMDB-27205: 
Closed state of SARS-CoV-2 BA.2 variant spike protein

EMDB-27206: 
One RBD-up state of SARS-CoV-2 BA.2 variant spike protein

EMDB-27207: 
Middle state of SARS-CoV-2 BA.2 variant spike protein

EMDB-29016: 
Cryo-EM structure of SARS-CoV-2 postfusion spike in membrane
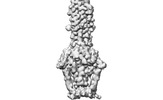
EMDB-29017: 
Cryo-EM structure of SARS-CoV-2 postfusion spike in membrane

EMDB-29018: 
Cryo-EM structure of SARS-CoV-2 postfusion spike in membrane

EMDB-33796: 
The NuA4 histone acetyltransferase complex from S. cerevisiae

EMDB-40094: 
17b10 fab in complex with full-length SARS-CoV-2 Spike G614 trimer

EMDB-40095: 
17B10 fab in complex with up-RBD of SARS-CoV-2 Spike G614 trimer

EMDB-35522: 
Cryo-EM structure of the TUG891 bound GPR120-Giq complex(mask on receptor)

EMDB-35523: 
Cryo-EM structure of the TUG891 bound GPR120-Giq complex(mask on Giq-scFV16 complex)

EMDB-35524: 
Cryo-EM structure of the eicosapentaenoic acid bound GPR120-Gi1 complex(mask on receptor)

EMDB-35525: 
Cryo-EM structure of the eicosapentaenoic acid bound GPR120-Gi1 complex(mask on Gil-scFV16 complex)

EMDB-35529: 
Cryo-EM structure of the TUG891 bound GPR120-Giq complex (consensus map)

EMDB-35533: 
Cryo-EM structure of the eicosapentaenoic acid bound GPR120-Gi complex(consensus map)

EMDB-35356: 
Cryo-EM structure of the 9-hydroxystearic acid bound GPR120-Gi complex

EMDB-35357: 
Cryo-EM structure of the linoleic acid bound GPR120-Gi complex

EMDB-35358: 
Cryo-EM structure of the oleic acid bound GPR120-Gi complex

EMDB-35359: 
Cryo-EM structure of the TUG891 bound GPR120-Gi complex

EMDB-35360: 
Cryo-EM structure of the eicosapentaenoic acid bound GPR120-Gi complex

EMDB-29736: 
Cryo-EM structure of the TUG891 bound GPR120-Giq complex

EMDB-33794: 
Core module of the NuA4 complex in S. cerevisiae

EMDB-32356: 
Computational design of a potent Epstein-Barr virus fusion protein gB nanoparticle vaccine

EMDB-32088: 
Computational design of a potent Epstein-Barr virus fusion protein gB nanoparticle vaccine

EMDB-26676: 
Three RBD-down state of SARS-CoV-2 D614G spike in complex with the SP1-77 neutralizing antibody Fab fragment

EMDB-26677: 
Three RBD-down state of SARS-CoV-2 D614G spike in complex with the SP1-77 neutralizing antibody Fab fragment (local refinement of the RBD and Fab variable domains)

EMDB-26678: 
An antibody from single human VH-rearranging mouse neutralizes all SARS-CoV-2 variants through BA.5 by inhibiting membrane fusion

EMDB-27043: 
Negative stain electron microscopy single particle reconstruction of monoclonal antibody SP1-77 Fab in complex with SARS-CoV2 2P spike

EMDB-27044: 
Negative stain electron microscopy single particle reconstruction of monoclonal antibody VHH7-7-53 Fab in complex with SARS-CoV2 2P spike

EMDB-27046: 
Negative stain electron microscopy single particle reconstruction of monoclonal antibody VHH7-5-82 Fab in complex with SARS-CoV2 2P spike

EMDB-26021: 
Structural and functional impact by SARS-CoV-2 Omicron spike mutations

EMDB-26029: 
Structural and functional impact by SARS-CoV-2 Omicron spike mutations

EMDB-24982: 
One RBD-up 1 of pre-fusion SARS-CoV-2 Delta variant spike protein

EMDB-24988: 
One RBD-up 2 of pre-fusion SARS-CoV-2 Gamma variant spike protein

EMDB-24981: 
Closed state of pre-fusion SARS-CoV-2 Delta variant spike protein

EMDB-24983: 
One RBD-up 2 of pre-fusion SARS-CoV-2 Delta variant spike protein

EMDB-24984: 
Closed state of pre-fusion SARS-CoV-2 Kappa variant spike protein

EMDB-24985: 
One RBD-up 1 of pre-fusion SARS-CoV-2 Kappa variant spike protein

EMDB-24986: 
One RBD-up 2 of pre-fusion SARS-CoV-2 Kappa variant spike protein

EMDB-24987: 
One RBD-up 1 of pre-fusion SARS-CoV-2 Gamma variant spike protein

EMDB-24121: 
Structural basis for enhanced infectivity and immune evasion of SARS-CoV-2 variants

EMDB-24122: 
Structural basis for enhanced infectivity and immune evasion of SARS-CoV-2 variants

EMDB-24123: 
Structural basis for enhanced infectivity and immune evasion of SARS-CoV-2 variants

EMDB-24124: 
Structural basis for enhanced infectivity and immune evasion of SARS-CoV-2 variants

EMDB-24125: 
Structural basis for enhanced infectivity and immune evasion of SARS-CoV-2 variants

EMDB-24126: 
Structural basis for enhanced infectivity and immune evasion of SARS-CoV-2 variants

EMDB-24127: 
Structural basis for enhanced infectivity and immune evasion of SARS-CoV-2 variants
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
