-検索条件
-検索結果
検索 (著者・登録者: tung & c)の結果126件中、1から50件目までを表示しています

EMDB-38216: 
Cryo-EM structure of SARS-CoV-2 S-BQ.1 in complex with antibody O5C2

PDB-8xbf: 
Cryo-EM structure of SARS-CoV-2 S-BQ.1 in complex with antibody O5C2

EMDB-18170: 
YPEL5-bound WDR26-CTLH E3 ligase - assembly I

EMDB-18171: 
YPEL5-bound WDR26-CTLH E3 ligase - assembly II

EMDB-18172: 
NMNAT1 core-bound RANBP9-TWA1-WDR26 module of WDR26-CTLH E3 ligase

EMDB-18173: 
NMNAT1 loop-bound RANBP9-TWA1-WDR26 module of WDR26-CTLH E3 ligase

EMDB-18174: 
NMNAT1-bound WDR26-CTLH E3 ligase assembly I - class 1

EMDB-18175: 
NMNAT1-bound WDR26-CTLH E3 ligase assembly I - class 2

EMDB-18176: 
NMNAT1-bound WDR26-CTLH E3 ligase assembly II - class 1

EMDB-18177: 
NMNAT1-bound WDR26-CTLH E3 ligase assembly II - class 2

EMDB-18178: 
NMNAT1-bound WDR26-CTLH E3 ligase assembly II - class 3

EMDB-18316: 
Structure of the non-canonical CTLH E3 substrate receptor WDR26 bound to YPEL5

EMDB-18345: 
Structure of the non-canonical CTLH E3 substrate receptor WDR26 bound to NMNAT1 substrate

PDB-8qbn: 
Structure of the non-canonical CTLH E3 substrate receptor WDR26 bound to YPEL5

PDB-8qe8: 
Structure of the non-canonical CTLH E3 substrate receptor WDR26 bound to NMNAT1 substrate

EMDB-18214: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex - hexameric assembly
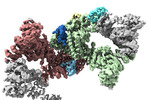
EMDB-18216: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused cullin dimer
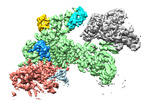
EMDB-18217: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused on E2-like density

EMDB-18218: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused dimeric core
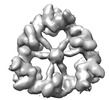
EMDB-18220: 
Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 CPH domain
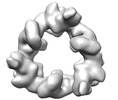
EMDB-18221: 
Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 DOC domain
EMDB-18222: 
Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 ARM9 domain
EMDB-18223: 
Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 ARIH-RBR element

EMDB-19179: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated conformation - symmetry expanded unneddylated dimer

PDB-8q7e: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex - hexameric assembly

PDB-8q7h: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused cullin dimer

PDB-8rhz: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated conformation - symmetry expanded unneddylated dimer

EMDB-37631: 
Hepatitis B virus capsid (HBV core protein)

EMDB-37634: 
SR protein kinase 2 bound at 2-fold vertex of Hepatitis B virus capsid

EMDB-38062: 
Hepatitis B virus capsid in complex with SR protein kinase 2

EMDB-17705: 
Structure of Chelator-GIDSR4 - Fbp1 - phospho-Ubc8~ubiquitin - class I

EMDB-17706: 
Structure of Chelator-GIDSR4 - Fbp1 - phospho-Ubc8~ubiquitin - class II

EMDB-17707: 
Chelator-GIDSR4 - Fbp1 - phospho-Ubc8~ubiquitin - class III

EMDB-17709: 
Structure of Chelator-GIDSR4 - Fbp1 - phospho-Ubc8~ubiquitin - class V

EMDB-17710: 
Structure of Chelator-GIDSR4 - Fbp1 - phospho-Ubc8~ubiquitin - class IV

EMDB-17713: 
Catalytic module of human CTLH E3 ligase bound to multiphosphorylated UBE2H~ubiquitin

EMDB-17715: 
SRS and Cat modules of human CTLHSR4 bound to multiphosphorylated UBE2H~ubiquitin

EMDB-17716: 
Structure of CTLHSR4 - phospho-UBE2H~ubiquitin bound to engineered VH
EMDB-17717: 
SRS and Cat modules of yeast Chelator-GIDSR4 bound to multiphosphorylated Ubc8~ubiquitin
EMDB-17764: 
Catalytic module of yeast GID E3 ligase bound to multiphosphorylated Ubc8~ubiquitin

PDB-8pjn: 
Catalytic module of human CTLH E3 ligase bound to multiphosphorylated UBE2H~ubiquitin

PDB-8pmq: 
Catalytic module of yeast GID E3 ligase bound to multiphosphorylated Ubc8~ubiquitin

EMDB-16355: 
Structure of Dimeric HECT E3 Ubiquitin Ligase UBR5

EMDB-16356: 
Structure of HECT E3 UBR5 forming K48 linked Ubiquitin chains

EMDB-16865: 
Tetrameric HECT E3 Ubiquitin Ligase UBR5

EMDB-16866: 
Cryo-EM map of ubiquitin-VME bound HECT E3 ligase UBR5

EMDB-16867: 
Ubiquitin transfer from E2 to E3: UBE2D2-ubiquitin linked to HECT E3 ligase UBR5

EMDB-17466: 
Cryo-EM map of HECT E3 ligase UBR5 forming K48 linked ubiquitin chains

PDB-8c06: 
Structure of Dimeric HECT E3 Ubiquitin Ligase UBR5

PDB-8c07: 
Structure of HECT E3 UBR5 forming K48 linked Ubiquitin chains
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
