-検索条件
-検索結果
検索 (著者・登録者: sham & a)の結果226件中、1から50件目までを表示しています

EMDB-50409: 
Subtomogram average of 80S ribosomes in native S. cerevisiae

EMDB-50415: 
Subtomogram average of 80S ribosomes in S. cerevisiae under acute glucose starvation

EMDB-50672: 
A 3.3A sub-tomogram average of HIV-1 CA-SP1 from 5 tomograms in EMPIAR-10164 obtained using RELION 5

EMDB-38453: 
Structure of the SARS-CoV-2 EG.5.1 spike glycoprotein in complex with ACE2 (1-up state)

EMDB-38454: 
Structure of the SARS-CoV-2 EG.5.1 spike RBD in complex with ACE2

PDB-8xlm: 
Structure of the SARS-CoV-2 EG.5.1 spike glycoprotein in complex with ACE2 (1-up state)

PDB-8xln: 
Structure of the SARS-CoV-2 EG.5.1 spike RBD in complex with ACE2

EMDB-37648: 
SARS-CoV-2 EG.5.1 spike glycoprotein (1-up state)

EMDB-37650: 
SARS-CoV-2 EG.5.1 spike glycoprotein (closed-2 state)

EMDB-37651: 
SARS-CoV-2 EG.5.1 spike glycoprotein (closed-1 state)

PDB-8wmd: 
Structure of the SARS-CoV-2 EG.5.1 spike glycoprotein (closed-2 state)

PDB-8wmf: 
Structure of the SARS-CoV-2 EG.5.1 spike glycoprotein (closed-1 state)

EMDB-18119: 
Sulfolobus acidocaldarius AAP filament.

PDB-8q30: 
Sulfolobus acidocaldarius AAP filament.

EMDB-19064: 
CryoEM reconstruction of SARS-CoV-2 spike in complex with nanobody tri-TMH (partially open conformation)

EMDB-19068: 
CryoEM reconstruction of SARS-CoV-2 Spike in complex with nanobody tri-TMH (closed conformation)

EMDB-29423: 
Structure of Escherichia coli CedA in complex with transcription initiation complex

PDB-8ftd: 
Structure of Escherichia coli CedA in complex with transcription initiation complex

EMDB-16334: 
Cryo-EM structure of a Staphylococus aureus 30S-RbfA complex

PDB-8byv: 
Cryo-EM structure of a Staphylococus aureus 30S-RbfA complex

EMDB-34715: 
Cryo-EM structure of ComA bound to its mature substrate CSP peptide
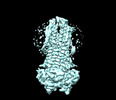
EMDB-36882: 
Cryo-EM structure of nucleotide-bound ComA with ZinC ion

EMDB-36936: 
Cryo-EM structure of nucleotide-bound ComA E647Q mutant with Mg2+

PDB-8hf7: 
Cryo-EM structure of ComA bound to its mature substrate CSP peptide

PDB-8k4b: 
Cryo-EM structure of nucleotide-bound ComA with ZinC ion

PDB-8k7a: 
Cryo-EM structure of nucleotide-bound ComA E647Q mutant with Mg2+

EMDB-35362: 
Cryo-EM structure of Mycobacterium tuberculosis FtsEX complex in peptidisc

EMDB-35363: 
Cryo-EM structure of Mycobacterium tuberculosis FtsEX/RipC complex in peptidisc
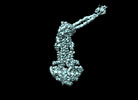
EMDB-35364: 
Cryo-EM structure of Mycobacterium tuberculosis ATP bound FtsEX/RipC complex in peptidisc

EMDB-35437: 
Cryo-EM structure of Mycobacterium tuberculosis ADP bound FtsEX/RipC complex in peptidisc

EMDB-36304: 
Cryo-EM structure of Mycobacterium tuberculosis ATP bound FtsE(E165Q)X/RipC complex in peptidisc

PDB-8idb: 
Cryo-EM structure of Mycobacterium tuberculosis FtsEX complex in peptidisc

PDB-8idc: 
Cryo-EM structure of Mycobacterium tuberculosis FtsEX/RipC complex in peptidisc

PDB-8idd: 
Cryo-EM structure of Mycobacterium tuberculosis ATP bound FtsEX/RipC complex in peptidisc

PDB-8igq: 
Cryo-EM structure of Mycobacterium tuberculosis ADP bound FtsEX/RipC complex in peptidisc

PDB-8jia: 
Cryo-EM structure of Mycobacterium tuberculosis ATP bound FtsE(E165Q)X/RipC complex in peptidisc

EMDB-34712: 
Cryo-EM structure of nucleotide-bound ComA at outward-facing state with EC gate closed conformation

EMDB-34713: 
Cryo-EM structure of nucleotide-bound ComA at outward-facing state with EC gate open conformation

EMDB-34714: 
Cryo-EM structure of nucleotide-bound ComA E647Q mutant

EMDB-34716: 
Cryo-EM structure of ComC bound ComA C17A at inward-facing state

PDB-8hf4: 
Cryo-EM structure of nucleotide-bound ComA at outward-facing state with EC gate closed conformation

PDB-8hf5: 
Cryo-EM structure of nucleotide-bound ComA at outward-facing state with EC gate open conformation

PDB-8hf6: 
Cryo-EM structure of nucleotide-bound ComA E647Q mutant

EMDB-28036: 
Cryo-EM structure of the full-length human NF1 dimer

PDB-8edm: 
Cryo-EM structure of the full-length human NF1 dimer

EMDB-28035: 
Cryo-EM structure of the full-length human NF1 dimer

EMDB-28037: 
Cryo-EM structure of the full-length human NF1 dimer

EMDB-28038: 
Cryo-EM structure of the full-length human NF1 dimer

PDB-8edl: 
Cryo-EM structure of the full-length human NF1 dimer

PDB-8edn: 
Cryo-EM structure of the full-length human NF1 dimer
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
