-検索条件
-検索結果
検索 (著者・登録者: schall & k)の結果58件中、1から50件目までを表示しています

EMDB-19163: 
Trimeric HSV-1F gB ectodomain in postfusion conformation with three bound HDIT101 Fab molecules.

EMDB-19164: 
Trimeric HSV-1F gB ectodomain in postfusion conformation with three bound HDIT102 Fab molecules.

EMDB-19165: 
Trimeric HSV-2F gB ectodomain in postfusion conformation with three bound HDIT101 Fab molecules.

EMDB-19166: 
Trimeric HSV-2G gB ectodomain in postfusion conformation with three bound HDIT102 Fab molecules.

PDB-8rgz: 
Trimeric HSV-1F gB ectodomain in postfusion conformation with three bound HDIT101 Fab molecules.

PDB-8rh0: 
Trimeric HSV-1F gB ectodomain in postfusion conformation with three bound HDIT102 Fab molecules.

PDB-8rh1: 
Trimeric HSV-2F gB ectodomain in postfusion conformation with three bound HDIT101 Fab molecules.

PDB-8rh2: 
Trimeric HSV-2G gB ectodomain in postfusion conformation with three bound HDIT102 Fab molecules.

EMDB-16024: 
SARS-CoV-2 S protein in complex with pT1644 Fab

EMDB-16026: 
SARS-CoV-2 S protein in complex with pT1696 Fab

EMDB-15165: 
Structure of Arp4-Ies4-N-actin-Arp8-Ino80HSA subcomplex (A-module) of Chaetomium thermophilum INO80

PDB-8a5d: 
Structure of Arp4-Ies4-N-actin-Arp8-Ino80HSA subcomplex (A-module) of Chaetomium thermophilum INO80

EMDB-15163: 
Cryo-EM reconstruction of Arp4-Ies4-N-actin-Arp8-Ino80HSA subcomplex (A-module) of INO80

EMDB-15164: 
Human Ino80 A-module + YY1

EMDB-15177: 
Cryo-EM reconstruction of Arp4-Ies4-N-actin-Arp8-Ino80HSA subcomplex (A-module) of S. cerevisiae INO80

EMDB-15179: 
Cryo-EM reconstruction of Arp4-Ies4-N-actin-Arp8-Ino80HSA subcomplex (A-module) of S. cerevisiae INO80

EMDB-15180: 
Structure of Arp4-Ies4-N-actin-Arp8-Ino80HSA subcomplex (A-module) of Chaetomium thermophilum INO80 on curved DNA

EMDB-15184: 
Structure of Arp4-Ies4-N-actin-Arp8-Ino80HSA subcomplex (A-module) of Chaetomium thermophilum INO80 on straight DNA

EMDB-15186: 
Cryo-EM reconstruction of DNA bound Arp4-Ies4-N-actin-Arp8-Ino80HSA subcomplex (A-module) of S. cerevisiae INO80

EMDB-15187: 
A-module of Chaetomium thermophilum INO80 bound to curved DNA

EMDB-15188: 
Nucleosome bound Chaetomium thermophilum INO80 (ADP-BeF3 state) core complex with Arp5 grappler in parallel conformation

EMDB-15211: 
Ino80 core complex with A-module on nucleosome

EMDB-15647: 
Nucleosome-bound Ino80 ATPase

EMDB-15688: 
CryoEM structure of INO80 core nucleosome complex in closed grappler conformation (ADP-BeFx state)

PDB-8a5a: 
Structure of Arp4-Ies4-N-actin-Arp8-Ino80HSA subcomplex (A-module) of INO80

PDB-8a5o: 
Structure of Arp4-Ies4-N-actin-Arp8-Ino80HSA subcomplex (A-module) of S. cerevisiae INO80

PDB-8a5p: 
Structure of Arp4-Ies4-N-actin-Arp8-Ino80HSA subcomplex (A-module) of Chaetomium thermophilum INO80 on curved DNA

PDB-8a5q: 
Structure of Arp4-Ies4-N-actin-Arp8-Ino80HSA subcomplex (A-module) of Chaetomium thermophilum INO80 on straight DNA

PDB-8atf: 
Nucleosome-bound Ino80 ATPase

PDB-8av6: 
CryoEM structure of INO80 core nucleosome complex in closed grappler conformation
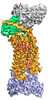
EMDB-25171: 
Cryo-EM structure of ACKR3 in complex with CXCL12, an intracellular Fab, and an extracellular Fab

EMDB-25172: 
Cryo-EM structure of ACKR3 in complex with chemokine N-terminal mutant CXCL12_LRHQ, an intracellular Fab, and an extracellular Fab
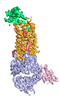
EMDB-25173: 
Cryo-EM structure of ACKR3 in complex with CXCL12 and an intracellular Fab
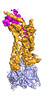
EMDB-25174: 
Cryo-EM structure of human ACKR3 in complex with chemokine N-terminal mutant CXCL12_LRHQ and an intracellular Fab
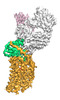
EMDB-25175: 
Cryo-EM structure of human ACKR3 in complex with CXCL12, a small molecule partial agonist CCX662, and an extracellular Fab
EMDB-25176: 
Cryo-EM structure of human ACKR3 in complex with CXCL12, a small molecule partial agonist CCX662, an extracellular Fab, and an intracellular Fab
EMDB-25177: 
Cryo-EM structure of human ACKR3 in complex with a small molecule partial agonist CCX662, and an intracellular Fab

PDB-7sk3: 
Cryo-EM structure of ACKR3 in complex with CXCL12, an intracellular Fab, and an extracellular Fab

PDB-7sk4: 
Cryo-EM structure of ACKR3 in complex with chemokine N-terminal mutant CXCL12_LRHQ, an intracellular Fab, and an extracellular Fab

PDB-7sk5: 
Cryo-EM structure of ACKR3 in complex with CXCL12 and an intracellular Fab

PDB-7sk6: 
Cryo-EM structure of human ACKR3 in complex with chemokine N-terminal mutant CXCL12_LRHQ and an intracellular Fab

PDB-7sk7: 
Cryo-EM structure of human ACKR3 in complex with CXCL12, a small molecule partial agonist CCX662, and an extracellular Fab

PDB-7sk8: 
Cryo-EM structure of human ACKR3 in complex with CXCL12, a small molecule partial agonist CCX662, an extracellular Fab, and an intracellular Fab

PDB-7sk9: 
Cryo-EM structure of human ACKR3 in complex with a small molecule partial agonist CCX662, and an intracellular Fab

EMDB-21872: 
Structure of the 50S subunit of the ribosome from Methicillin Resistant Staphylococcus aureus in complex with the antibiotic, contezolid

EMDB-21873: 
Structure of the 50S subunit of the ribosome from Methicillin Resistant Staphylococcus aureus in complex with the antibiotic, radezolid

EMDB-21887: 
Structure of the 50S subunit of the ribosome from Methicillin Resistant Staphylococcus aureus in complex with the antibiotic, tedizolid

EMDB-21888: 
Structure of the 50S subunit of the ribosome from Methicillin Resistant Staphylococcus aureus in complex with an isomer of the tedizolid

PDB-6wqn: 
Structure of the 50S subunit of the ribosome from Methicillin Resistant Staphylococcus aureus in complex with the antibiotic, contezolid

PDB-6wqq: 
Structure of the 50S subunit of the ribosome from Methicillin Resistant Staphylococcus aureus in complex with the antibiotic, radezolid
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
