-検索条件
-検索結果
検索 (著者・登録者: saga & y)の結果103件中、1から50件目までを表示しています

EMDB-45235: 
Subtomogram average (C1) of fatty acid synthase from S.cerevisiae prepared using cryo-plasmaFIB milling

EMDB-40815: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies C3V5, V1V3, N611 and base from participant 017

EMDB-40816: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies gp41-N611/FP and base from participant 03

EMDB-40817: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies gp41-N611/FP and base from participant 07

EMDB-40818: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies gp120-GH and base from participant 09

EMDB-40819: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies C3V5, V1V3, gp41-GH/FP and base from participant 11
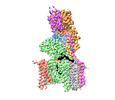
EMDB-17779: 
Structure of human oligosaccharyltransferase OST-A complex bound to NGI-1

PDB-8pn9: 
Structure of human oligosaccharyltransferase OST-A complex bound to NGI-1

EMDB-36905: 
SARS-CoV-2 BA.1 RBD with UT28-RD

EMDB-36906: 
SARS-CoV-2 BA.1 spike with UT28-RD

PDB-8k5g: 
Structure of the SARS-CoV-2 BA.1 RBD with UT28-RD

PDB-8k5h: 
Structure of the SARS-CoV-2 BA.1 spike with UT28-RD

EMDB-18383: 
Cryo-EM structure of SidH from Legionella pneumophila

EMDB-18407: 
Cryo-EM structure of SidH from Legionella pneumophila in complex with LubX

PDB-8qfs: 
Cryo-EM structure of SidH from Legionella pneumophila

PDB-8qhc: 
Cryo-EM structure of SidH from Legionella pneumophila in complex with LubX

EMDB-18231: 
CryoDRGN Reconstruction of 80S Ribosome in Rotated State from S.cerevisiae Prepared using Cryo-plasmaFIB Milling

EMDB-18232: 
CryoDRGN Reconstruction of 80S Ribosome in Unrotated State from S.cerevisiae Prepared using Cryo-plasmaFIB Milling

EMDB-27025: 
CRYO-EM STRUCTURE OF HUMAN 15-PGDH IN COMPLEX WITH SMALL MOLECULE SW222746

EMDB-29005: 
human 15-PGDH with NADH bound

PDB-8cwl: 
Cryo-EM structure of Human 15-PGDH in complex with small molecule SW222746

PDB-8fd8: 
human 15-PGDH with NADH bound

EMDB-27010: 
CRYO-EM STRUCTURE OF HUMAN 15-PGDH IN COMPLEX WITH SMALL MOLECULE SW209415

PDB-8cvn: 
Cryo-EM Structure of Human 15-PGDH in Complex with Small Molecule SW209415

EMDB-33045: 
SARS-CoV-2 spike glycoprotein in complex with neutralizing antibody UT28K
EMDB-14170: 
Low resolution negative stain map of SslE

EMDB-14467: 
2.1A T20S Proteosome from 200kV Glacios with Selectris and Falcon 4

EMDB-14173: 
1.6A Apoferritin from 200kV Glacios with Selectris X and Falcon 4

EMDB-22266: 
Structure of the Lactococcus lactis Csm CTR_4:3 CRISPR-Cas Complex

EMDB-22267: 
Structure of the Lactococcus lactis Csm CTR_3:2 CRISPR-Cas Complex

EMDB-22268: 
Structure of the Lactococcus lactis Csm Apo- CRISPR-Cas Complex

EMDB-22269: 
Structure of the Lactococcus lactis Csm NTR CRISPR-Cas Complex

PDB-6xn3: 
Structure of the Lactococcus lactis Csm CTR_4:3 CRISPR-Cas Complex

PDB-6xn4: 
Structure of the Lactococcus lactis Csm CTR_3:2 CRISPR-Cas Complex

PDB-6xn5: 
Structure of the Lactococcus lactis Csm Apo- CRISPR-Cas Complex

PDB-6xn7: 
Structure of the Lactococcus lactis Csm NTR CRISPR-Cas Complex

EMDB-13583: 
Structure of SidJ/CaM bound to SdeA in pre-glutamylation state

PDB-7ppo: 
Structure of SidJ/CaM bound to SdeA in pre-glutamylation state

EMDB-13591: 
Structure of SidJ/CaM bound to SdeA in post-catalysis state

PDB-7pqe: 
Structure of SidJ/CaM bound to SdeA in post-catalysis state

EMDB-24518: 
Cryo-EM structure of human p97-R155H mutant bound to ADP.

EMDB-24519: 
Cryo-EM structure of human p97-R155H mutant bound to ATPgS.

EMDB-24522: 
Cryo-EM structure of human p97-R191Q mutant bound to ADP.

EMDB-24523: 
Cryo-EM structure of human p97-R191Q mutant bound to ATPgS.

EMDB-24524: 
Cryo-EM structure of human p97-A232E mutant bound to ADP

EMDB-24525: 
Cryo-EM structure of human p97-A232E mutant bound to ATPgS.

EMDB-24526: 
Cryo-EM structure of human p97-E470D mutant bound to ADP.

EMDB-24528: 
Cryo-EM structure of human p97-E470D mutant bound to ATPgS.

EMDB-24529: 
Cryo-EM structure of human p97-D592N mutant bound to ADP.

EMDB-24530: 
Cryo-EM structure of human p97-D592N mutant bound to ATPgS.
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
