-検索条件
-検索結果
検索 (著者・登録者: rao & b)の結果725件中、1から50件目までを表示しています
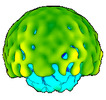
EMDB-50358: 
In vitro-induced genome-releasing intermediate of Rhodobacter microvirus Ebor computed with C5 symmetry

EMDB-50229: 
Cryo-tomogram of FIB-milled vegetatively growing yeast cell with mitochondria

EMDB-50230: 
Cryo-tomogram of FIB-milled pre-meiotic yeast cell with mitochondria
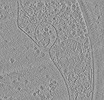
EMDB-50231: 
Cryo-tomogram of FIB-milled meiotic yeast cell containing mitochondria with filaments
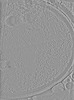
EMDB-50232: 
Cryo-tomogram of FIB-milled yeast spore with mitochondria

EMDB-50233: 
Cryo-tomogram of FIB-milled meiotic yeast cell containing mitochondria with filament arrays

EMDB-50234: 
Cryo-tomogram of purified meiotic yeast mitochondria with Ald4 filaments

EMDB-42543: 
KIF1A[1-393] AMP-PNP bound two-heads-bound state in complex with a microtubule (class T23L1)

EMDB-42544: 
KIF1A[1-393] AMP-PNP bound two-heads-bound state in complex with a microtubule - class T2L1

EMDB-42545: 
KIF1A[1-393] - AMP-PNP two-heads-bound state in complex with a microtubule - class T3L1

EMDB-42546: 
KIF1A[1-393] AMP-PNP bound one-head-bound state in complex with a microtubule - class T1L02*

EMDB-42547: 
KIF1A[1-393] ADP bound in complex with a microtubule

EMDB-42548: 
KIF1A[1-393] APO in complex with a microtubule

EMDB-42549: 
KIF1A[1-393] P305L mutant AMP-PNP bound two-heads-bound state in complex with a microtubule

EMDB-42550: 
KIF1A[1-393] AMP-PNP bound one-head-bound state in complex with a microtubule - class T1L02*

EMDB-42551: 
KIF1A[1-393] P305L mutant ADP bound in complex with a microtubule

EMDB-42552: 
KIF1A[1-393] P305L mutant APO in complex with a microtubule

EMDB-42553: 
KIF1A[1-393] P364L mutant AMP-PNP bound two-heads-bound state in complex with a microtubule

PDB-8utn: 
KIF1A[1-393] AMP-PNP bound two-heads-bound state in complex with a microtubule (class T23L1)

PDB-8uto: 
KIF1A[1-393] AMP-PNP bound two-heads-bound state in complex with a microtubule - class T2L1

PDB-8utp: 
KIF1A[1-393] - AMP-PNP two-heads-bound state in complex with a microtubule - class T3L1

PDB-8utq: 
KIF1A[1-393] AMP-PNP bound one-head-bound state in complex with a microtubule - class T1L02*

PDB-8utr: 
KIF1A[1-393] ADP bound in complex with a microtubule

PDB-8uts: 
KIF1A[1-393] APO in complex with a microtubule

PDB-8utt: 
KIF1A[1-393] P305L mutant AMP-PNP bound two-heads-bound state in complex with a microtubule

PDB-8utu: 
KIF1A[1-393] P305L mutant AMP-PNP bound one and two heads bound states merged, in complex with a microtubule

PDB-8utv: 
KIF1A[1-393] P305L mutant ADP bound in complex with a microtubule

PDB-8utw: 
KIF1A[1-393] P305L mutant APO in complex with a microtubule

PDB-8uty: 
KIF1A[1-393] P364L mutant AMP-PNP bound two-heads-bound state in complex with a microtubule

EMDB-50356: 
Empty capsid of Rhodobacter microvirus Ebor computed with I4 symmetry

EMDB-50357: 
Native capsid of Rhodobacter microvirus Ebor computed with I4 symmetry
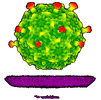
EMDB-50359: 
Rhodobacter microvirus Ebor attached to B10 host cell reconstructed by single particle analysis with applied C5 symmetry
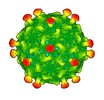
EMDB-50360: 
Rhodobacter microvirus Ebor attached to the outer membrane vesicle

EMDB-50361: 
Rhodobacter microvirus Ebor attached to the host cell reconstructed by subtomogram averaging

PDB-9ffg: 
Empty capsid of Rhodobacter microvirus Ebor computed with I4 symmetry

PDB-9ffh: 
Native capsid of Rhodobacter microvirus Ebor computed with I4 symmetry

EMDB-42845: 
Selenocysteine synthase- SelA

PDB-8uzw: 
Selenocysteine synthase- SelA
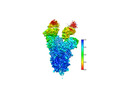
EMDB-37240: 
SARS-CoV-2 Omicron spike in complex with 5817 Fab

EMDB-37241: 
The interface structure of Omicron RBD binding to 5817 Fab

PDB-8khc: 
SARS-CoV-2 Omicron spike in complex with 5817 Fab

PDB-8khd: 
The interface structure of Omicron RBD binding to 5817 Fab

EMDB-35990: 
Cryo-EM structure of Mycobacterium tuberculosis OppABCD in the pre-translocation state

EMDB-35991: 
Cryo-EM structure of Mycobacterium tuberculosis OppABCD in the resting state

EMDB-35992: 
Cryo-EM structure of Mycobacterium tuberculosis OppABCD in the pre-catalytic intermediate state

EMDB-35993: 
Cryo-EM structure of Mycobacterium tuberculosis OppABCD in the catalytic intermediate state

PDB-8j5q: 
Cryo-EM structure of Mycobacterium tuberculosis OppABCD in the pre-translocation state

PDB-8j5r: 
Cryo-EM structure of Mycobacterium tuberculosis OppABCD in the resting state

PDB-8j5s: 
Cryo-EM structure of Mycobacterium tuberculosis OppABCD in the pre-catalytic intermediate state

PDB-8j5t: 
Cryo-EM structure of Mycobacterium tuberculosis OppABCD in the catalytic intermediate state
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
