-検索条件
-検索結果
検索 (著者・登録者: mishra & ak)の結果全50件を表示しています

EMDB-35817: 
Structure of Niacin-GPR109A-G protein complex

EMDB-35822: 
Structure of MK6892-GPR109A-G-protein complex

EMDB-35831: 
Structure of GSK256073-GPR109A-G-protein complex

EMDB-36193: 
Structure of Acipimox-GPR109A-G protein complex

EMDB-36280: 
Structure of MMF-GPR109A-G protein complex

EMDB-35257: 
Structure of EP54-C3aR-Go complex

EMDB-35259: 
Structure of Apo-C3aR-Go complex (Glacios)

EMDB-35263: 
Structure of EP54-C3aR-Gq complex

EMDB-35275: 
Structure of C3a-C3aR-Go complex (Composite map)

EMDB-35282: 
Structure of Apo-C3aR-Go complex (Titan)

EMDB-35292: 
Structure of C5a bound human C5aR1 in complex with Go (Composite map)

EMDB-35293: 
C3a-C3aR-Go (C3aR-Go complex only, Original Map)

EMDB-35294: 
C3a-C3aR-Go (C3a only, Original Map)

EMDB-35295: 
C5a-hC5aR1-Go (hC5aR1-Go complex only, Original map)
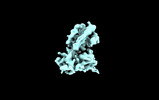
EMDB-35296: 
C5a-hC5aR1-Go complex (C5a only, Original map)

EMDB-36001: 
Structure of EP141-C3aR-Go complex

EMDB-36755: 
Structure of human C5a-desArg bound human C5aR1 in complex with Go

EMDB-40646: 
CryoEM structure of a therapeutic antibody (favezelimab) bound to human LAG3

EMDB-40716: 
CryoEM structure of a therapeutic antibody (favezelimab) bound to human LAG3 local refined

EMDB-23641: 
The structure of Bacillus subtilis BmrCD in the inward-facing conformation bound to Hoechst-33342 and ATP

PDB-7m33: 
The structure of Bacillus subtilis BmrCD in the inward-facing conformation bound to Hoechst-33342 and ATP

EMDB-24610: 
Structure of RNA-dependent RNA polymerase 2 (RDR2) from Arabidopsis thaliana

EMDB-24635: 
Arabidopsis RNA-dependent RNA polymerase 2

PDB-7roz: 
Structure of RNA-dependent RNA polymerase 2 (RDR2) from Arabidopsis thaliana

PDB-7rqs: 
Arabidopsis RNA-dependent RNA polymerase 2

EMDB-31972: 
Cryo-EM 3D reconstruction of Vibrio cholerae Cytolysin embedded in lipid bilayer- State 3

EMDB-31973: 
Cryo-EM 3D reconstruction of Vibrio cholerae Cytolysin adhered to liposome membrane surface

EMDB-31974: 
Cryo-EM 3D reconstruction of Vibrio cholerae Cytolysin partially embedded in lipid bilayer- State 2

EMDB-22788: 
Cryo-EM structure of the HCMV pentamer bound by Fabs 2-18 and 8I21

EMDB-23629: 
Cryo-EM structure of the HCMV pentamer bound by human neuropilin 2

EMDB-23640: 
Cryo-EM structure of the HCMV pentamer bound by antibodies 1-103, 1-32 and 2-25

PDB-7kbb: 
Cryo-EM structure of the HCMV pentamer bound by Fabs 2-18 and 8I21

PDB-7m22: 
Cryo-EM structure of the HCMV pentamer bound by human neuropilin 2

PDB-7m30: 
Cryo-EM structure of the HCMV pentamer bound by antibodies 1-103, 1-32 and 2-25

EMDB-31092: 
SARS-CoV2 Spike Protein structure at pH 6.5 with C1 Symmetry (Class 2)

EMDB-31093: 
SARS-CoV2 Spike Protein structure at pH 6.5 with C1 Symmetry (Class 3)

EMDB-31094: 
SARS-CoV2 Spike Protein structure at pH 6.5 with C1 Symmetry (Class 4)

EMDB-31095: 
SARS-CoV2 Spike Protein structure at pH 6.5 with C1 Symmetry (Class 5)

EMDB-31096: 
SARS-CoV2 Spike Protein structure at pH 7.4 with C1 Symmetry (Class 3)

EMDB-31097: 
SARS-CoV2 Spike Protein structure at pH 7.4 with C1 Symmetry (Class 5)

EMDB-31098: 
SARS-CoV2 Spike Protein structure at pH 7.4 with C3 Symmetry

EMDB-31099: 
SARS-CoV2 Spike Protein structure at pH 8.0 with C1 Symmetry (Class 1)

EMDB-31100: 
SARS-CoV2 Spike Protein structure at pH 7.4 with C1 Symmetry (Class 9)

EMDB-31101: 
SARS-CoV2 Spike Protein structure at pH 7.4 with C1 Symmetry (Class 8)

EMDB-31102: 
SARS-CoV2 Spike Protein structure at pH 8.0 with C1 Symmetry (Class 2)
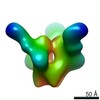
EMDB-6967: 
Anti-HIV-1 AIIMS-P01 Fab in complexed with BG505.SOSIP.664.C2 T332N gp140 trimer

EMDB-8369: 
Methicillin Resistant, Linezolid resistant Staphylococcus aureus 70S ribosome (delta S145 uL3)

PDB-5t7v: 
Methicillin Resistant, Linezolid resistant Staphylococcus aureus 70S ribosome (delta S145 uL3)

EMDB-8402: 
Methicillin sensitive Staphylococcus aureus 70S ribosome

PDB-5tcu: 
Methicillin sensitive Staphylococcus aureus 70S ribosome
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
