-検索条件
-検索結果
検索 (著者・登録者: lu & tw)の結果93件中、1から50件目までを表示しています

EMDB-39126: 
Structure of the FADD/Caspase-8/cFLIP death effector domain assembly

EMDB-39127: 
Structure of the FADD/Caspase-8/cFLIP death effector domain assembly
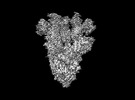
EMDB-28198: 
Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike in complex with LLNL-199

EMDB-28199: 
Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike in complex with 2130-1-0114-112

EMDB-38109: 
MCM in the Apo state.

EMDB-38112: 
local refinement of MCM_apo

EMDB-38113: 
local refinement of MCM_apo

EMDB-38114: 
local refinement of MCM_apo

EMDB-38115: 
local refinement of MCM_apo

EMDB-38116: 
local refinement of MCM_apo

EMDB-38117: 
local refinement of MCM_apo

EMDB-38118: 
local refinement of MCM_apo

EMDB-38120: 
local refinement of MCM_ATP_dsDNA

PDB-8x7t: 
MCM in the Apo state.
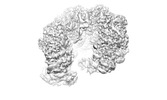
EMDB-38111: 
MCM in complex with dsDNA in presence of ATP.

EMDB-38119: 
MCM_ATP_dsDNA before local refinement

EMDB-38121: 
local refinement of MCM_ATP_dsDNA
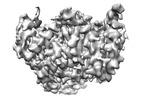
EMDB-38122: 
local refinement of MCM_ATP_dsDNA
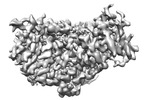
EMDB-38123: 
local refinement of MCM_ATP_dsDNA

EMDB-38124: 
local refinement of MCM_ATP_dsDNA

EMDB-38125: 
local refinement of MCM_ATP_dsDNA

EMDB-38126: 
local refinement of MCM_ATP_dsDNA

EMDB-38127: 
local refinement of MCM_ATP_dsDNA

PDB-8x7u: 
MCM in complex with dsDNA in presence of ATP.

EMDB-38110: 
Structure of MCM_apo before local refinement

EMDB-28776: 
Structure of VSD4-NaV1.7-NaVPas channel chimera bound to the hybrid inhibitor GNE-1305

EMDB-28777: 
Structure of VSD4-NaV1.7-NaVPas channel chimera bound to the acylsulfonamide inhibitor GDC-0310

EMDB-28778: 
Structure of VSD4-NaV1.7-NaVPas channel chimera bound to the arylsulfonamide inhibitor GNE-3565

EMDB-28779: 
Structure of VSD4-NaV1.7-NaVPas channel chimera bound to the hybrid inhibitor GNE-9296

PDB-8f0p: 
Structure of VSD4-NaV1.7-NaVPas channel chimera bound to the hybrid inhibitor GNE-1305

PDB-8f0q: 
Structure of VSD4-NaV1.7-NaVPas channel chimera bound to the acylsulfonamide inhibitor GDC-0310

PDB-8f0r: 
Structure of VSD4-NaV1.7-NaVPas channel chimera bound to the arylsulfonamide inhibitor GNE-3565

PDB-8f0s: 
Structure of VSD4-NaV1.7-NaVPas channel chimera bound to the hybrid inhibitor GNE-9296

EMDB-40094: 
17b10 fab in complex with full-length SARS-CoV-2 Spike G614 trimer

EMDB-40095: 
17B10 fab in complex with up-RBD of SARS-CoV-2 Spike G614 trimer

PDB-8gjm: 
17b10 fab in complex with full-length SARS-CoV-2 Spike G614 trimer

PDB-8gjn: 
17B10 fab in complex with up-RBD of SARS-CoV-2 Spike G614 trimer

EMDB-26005: 
Structure of the Inmazeb cocktail and resistance to escape against Ebola virus

PDB-7tn9: 
Structure of the Inmazeb cocktail and resistance to escape against Ebola virus

EMDB-25471: 
Structure of EBOV GP lacking the mucin-like domain with 1C11 scFv and 1C3 Fab bound

EMDB-13142: 
Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in inward-facing conformation without nucleotides

EMDB-13143: 
Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in inward-facing conformation with ADP/ATP

EMDB-13144: 
Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in inward-facing conformation with ADP/ATP and rhodamine 6G

EMDB-13145: 
Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in outward-facing conformation with ADP-orthovanadate/ATP

PDB-7p03: 
Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in inward-facing conformation without nucleotides

PDB-7p04: 
Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in inward-facing conformation with ADP/ATP

PDB-7p05: 
Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in inward-facing conformation with ADP/ATP and rhodamine 6G

PDB-7p06: 
Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in outward-facing conformation with ADP-orthovanadate/ATP

EMDB-12749: 
In situ cryo-electron tomogram of a pyrenoid inside a Chlamydomonas reinhardtii cell

EMDB-23156: 
SARS-CoV 2 Spike Protein bound to LY-CoV555
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
