-検索条件
-検索結果
検索 (著者・登録者: lou & zy)の結果51件中、1から50件目までを表示しています

EMDB-34314: 
SARS-CoV-2 RNA E-RTC complex with RMP-nsp9 and GMPPNP

EMDB-34316: 
SARS-CoV-2 E-RTC bound with MMP-nsp9 and GMPPNP

EMDB-34310: 
A mechanism for SARS-CoV-2 RNA capping and its inhibition by nucleotide analogue inhibitors

EMDB-34311: 
A mechanism for SARS-CoV-2 RNA capping and its inhibition by nucleotide analogue inhibitors

EMDB-34312: 
A mechanism for SARS-CoV-2 RNA capping and its inhibition by nucleotide analogue inhibitors

EMDB-34313: 
A mechanism for SARS-CoV-2 RNA capping and its inhibition by nucleotide analogue inhibitors
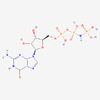
EMDB-34317: 
A mechanism for SARS-CoV-2 RNA capping and its inhibition by nucleotide analogue inhibitors

EMDB-34308: 
A mechanism for SARS-CoV-2 RNA capping and its inhibition by nucleotide analogue inhibitors
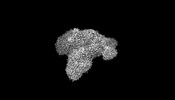
EMDB-34318: 
A mechanism for SARS-CoV-2 RNA capping and its inhibition by nucleotide analogue inhibitors

EMDB-33126: 
Cryo-EM structure of Dot1L and H2BK34ub-H3K79Nle nucleosome 1:1 complex

EMDB-33127: 
Cryo-EM structure of Dot1L and H2BK34ub-H3K79Nle nucleosome 2:1 complex
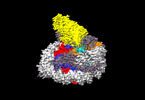
EMDB-33128: 
cryo-EM map of Dot1L and H2BK34ub-H3K79Nle nucleosome complex containing addition map (1:1)
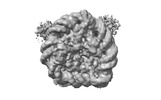
EMDB-33131: 
cryo-EM structure of H2BK34ub nucleosome

EMDB-33132: 
cryo-EM structure of unmodified nucleosome
EMDB-33139: 
cryo-EM map of Dot1L and H3K79Nle nucleosome complex (active state)
EMDB-33141: 
cryo-EM map of Dot1L and H3K79Nle nucleosome complex (inactive state)

EMDB-23312: 
BG505 SOSIP.v5.2 in complex with VRC40.01 and RM19R Fabs

PDB-7lg6: 
BG505 SOSIP.v5.2 in complex with VRC40.01 and RM19R Fabs

EMDB-23424: 
Cryo-EM map of Q23.17_DS-SOSIP in complex with Glycan276-Dependent Broadly Neutralizing Antibody 179NC75 Fab

PDB-7llk: 
Cryo-EM structure of Q23.17_DS-SOSIP in complex with Glycan276-Dependent Broadly Neutralizing Antibody 179NC75 Fab

PDB-7l6o: 
Cryo-EM structure of HIV-1 Env CH848.3.D0949.10.17chim.6R.DS.SOSIP.664

EMDB-30232: 
Structure of Dot1L-H2BK34ub Nucleosome Complex

EMDB-23518: 
Cryo-EM map of DH851.3 bound to HIV-1 CH505 Env

PDB-7lu9: 
Cryo-EM structure of DH851.3 bound to HIV-1 CH505 Env

EMDB-23411: 
Cryo-EM map of BG505 DS-SOSIP in complex with glycan276-dependent broadly neutralizing antibody VRC40.01 Fab

EMDB-23412: 
Cryo-EM map of BG505 DS-SOSIP in complex with Glycan276-Dependent Broadly Neutralizing Antibody VRC33.01 Fab

PDB-7ll1: 
Cryo-EM structure of BG505 DS-SOSIP in complex with glycan276-dependent broadly neutralizing antibody VRC40.01 Fab

PDB-7ll2: 
Cryo-EM structure of BG505 DS-SOSIP in complex with Glycan276-Dependent Broadly Neutralizing Antibody VRC33.01 Fab

EMDB-23519: 
Cryo-EM map of DH898.1 Fab-dimer bound near the CD4 binding site of HIV-1 Env CH848 SOSIP trimer

PDB-7lua: 
Cryo-EM structure of DH898.1 Fab-dimer bound near the CD4 binding site of HIV-1 Env CH848 SOSIP trimer

EMDB-23152: 
Cryo-electron microscopy reconstruction of antibody DH898.1 Fab-dimer bound to glycans 332, 392, and 396 of HIV Env CH848 10.17 SOSIP trimer

EMDB-23153: 
Cryo-electron microscopy local refinement of antibody DH898.1 Fab-dimer bound to glycans 332, 392, and 396 of HIV Env CH848 10.17 SOSIP trimer

EMDB-23124: 
Cryo-electron microcospy reconstruction of CH848.3.D0949.10.17chim.6R.DS.SOSIP.664 HIV Env

EMDB-23145: 
Cryo-electron microscopy reconstruction of locally refined antibody DH898.1 Fab-dimer

PDB-7l6m: 
Cryo-EM structure of DH898.1 Fab-dimer from local refinement of the Fab-dimer bound near the CD4 binding site of HIV-1 Env CH848 SOSIP trimer

EMDB-23094: 
Cryo-EM structure of SARS-CoV-2 2P S ectodomain bound to one copy of domain-swapped antibody 2G12

EMDB-23095: 
Cryo-EM structure of SARS-CoV-2 2P S ectodomain bound to two copies of domain-swapped antibody 2G12

EMDB-23097: 
Cryo-EM structure of SARS-CoV-2 2P S ectodomain bound domain-swapped antibody 2G12 from masked 3D refinement

PDB-7l02: 
Cryo-EM structure of SARS-CoV-2 2P S ectodomain bound to one copy of domain-swapped antibody 2G12

PDB-7l06: 
Cryo-EM structure of SARS-CoV-2 2P S ectodomain bound to two copies of domain-swapped antibody 2G12

PDB-7l09: 
Cryo-EM structure of SARS-CoV-2 2P S ectodomain bound domain-swapped antibody 2G12 from masked 3D refinement

EMDB-9607: 
Anthrax Toxin Receptor 1-bound spent particles of Seneca Valley Virus in acidic conditions

EMDB-9671: 
Adeno-Associated Virus 2 at 2.8 ang

EMDB-3982: 
Molecular Architecture of the Jumonji C histone demethylase KDM5B.

EMDB-9672: 
Adeno-Associated Virus 2 in complex with AAVR

EMDB-9608: 
Anthrax Toxin Receptor 1-bound full particles of Seneca Valley Virus in acidic conditions

EMDB-9611: 
Anthrax Toxin Receptor 1-bound the Seneca Valley Virus in neutral conditions

EMDB-9612: 
Structure of Seneca Valley Virus in acidic conditions

EMDB-9613: 
Structure of Seneca Valley Virus in neutral condition

EMDB-3649: 
Unraveling the self-assembly of Pseudomonas aeruginosa XcpQ secretin periplasmic domain provides new molecular insights into T2SS secreton architecture & dynamics
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
