-検索条件
-検索結果
検索 (著者・登録者: lin & sm)の結果1,084件中、1から50件目までを表示しています

EMDB-50296: 
70S Escherichia coli ribosome with P-site initiatior tRNA.

PDB-9fbv: 
70S Escherichia coli ribosome with P-site initiatior tRNA.

EMDB-40812: 
Structure of SARS-CoV-2 (HP-GSAS-Mut7) spike in complex with TXG-0078 Fab -Conformation 1

EMDB-40813: 
Structure of SARS-CoV-2 (HP-GSAS-Mut7) spike in complex with TXG-0078 Fab -Conformation 2
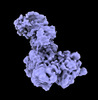
EMDB-40261: 
DDB1/CRBN in complex with ARV-471 and the ER ligand-binding domain
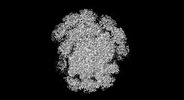
EMDB-37642: 
The structure of PSI-CAC(L-14)of R.salina at 2.7 angstroms resolution

EMDB-37654: 
structure of PSI-11CAC complex at Logrithmic growth phase

EMDB-37659: 
The structure of PSI-14CAC complex at stationary growth phase

EMDB-37660: 
The structure of PSI-11CAC at the stationary growth phase

EMDB-37674: 
the structure of PsaQ

EMDB-40814: 
Local refinement of SARS-CoV-2 (HP-GSAS-Mut7) spike NTD in complex with TXG-0078 Fab

PDB-8swh: 
Local refinement of SARS-CoV-2 (HP-GSAS-Mut7) spike NTD in complex with TXG-0078 Fab

EMDB-40825: 
10E8-GT10.2 immunogen in complex with human Fab 10E8 and mouse Fab W6-10

PDB-8sx3: 
10E8-GT10.2 immunogen in complex with human Fab 10E8 and mouse Fab W6-10

EMDB-41248: 
Structure of AT118-H Nanobody Antagonist in Complex with the Angiotensin II Type I Receptor

EMDB-41249: 
Structure of AT118-L Nanobody Antagonist in Complex with the Angiotensin II Type I Receptor and Losartan

EMDB-17296: 
Stabilised BA.1 SARS-CoV-2 spike with H6 nanobodies in '2 up 1 down' RBD conformation

PDB-8oyu: 
Stabilised BA.1 SARS-CoV-2 spike with H6 nanobodies in '2 up 1 down' RBD conformation

EMDB-16103: 
Human serotonin 5-HT3A receptor (apo, active/distorted conformation)

EMDB-16104: 
Human serotonin 5-HT3A receptor in complex with vortioxetine (detergent, ECD only, active/distorted conformation)
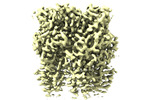
EMDB-16105: 
Human serotonin 5-HT3A receptor in complex with vortioxetine (nanodiscs, ECD, active/distorted conformation)

EMDB-17835: 
Consensus cryo-EM structure of Dynein-Dynactin-JIP3(1-185)-LIS1

EMDB-17836: 
Consensus cryo-EM structure of Dynein-dynactin-JIP3(1-560)-LIS1

EMDB-41277: 
Cryo-EM structure of a SUR1/Kir6.2-Q52R ATP-sensitive potassium channel in the presence of PIP2 in the open conformation

EMDB-41278: 
Cryo-EM structure of a SUR1/Kir6.2-Q52R ATP-sensitive potassium channel in the presence of PIP2 in the open conformation

EMDB-43766: 
Kir6.2-Q52R/SUR1 apo closed channel

PDB-8ti1: 
Cryo-EM structure of a SUR1/Kir6.2-Q52R ATP-sensitive potassium channel in the presence of PIP2 in the open conformation

PDB-8ti2: 
Cryo-EM structure of a SUR1/Kir6.2-Q52R ATP-sensitive potassium channel in the presence of PIP2 in the open conformation

EMDB-43629: 
Cryo-EM structure of phage DEV ejection proteins gp72:gp73

EMDB-17825: 
Cytoplasmic dynein-1 motor domain in post-powerstroke state

EMDB-17826: 
Cytoplasmic dynein-1 motor domain bound to dynactin-p150glued and LIS1

EMDB-17828: 
Cytoplasmic dynein-1 motor domain bound to LIS1

EMDB-17829: 
Cytoplasmic dynein-1 A1/A2 motor domains bound to LIS1

EMDB-17830: 
Cytoplasmic dynein-A heavy chain bound to dynactin-p150glued and IC-LC tower

EMDB-17831: 
Cytoplasmic dynein-B heavy chain bound to IC-LC tower

EMDB-17832: 
Cytoplasmic dynein-1 heavy chain bound to JIP3-LZI

EMDB-17833: 
Cytoplasmic dynein-1 heavy chain bound to JIP3-RH1

EMDB-17834: 
Dynactin pointed end bound to JIP3

EMDB-17873: 
Composite structure of Dynein-Dynactin-JIP3-LIS1

PDB-8pqv: 
Cytoplasmic dynein-1 motor domain in post-powerstroke state

PDB-8pqw: 
Cytoplasmic dynein-1 motor domain bound to dynactin-p150glued and LIS1

PDB-8pqy: 
Cytoplasmic dynein-1 motor domain bound to LIS1

PDB-8pqz: 
Cytoplasmic dynein-1 A1/A2 motor domains bound to LIS1

PDB-8pr0: 
Cytoplasmic dynein-A heavy chain bound to dynactin-p150glued and IC-LC tower

PDB-8pr1: 
Cytoplasmic dynein-B heavy chain bound to IC-LC tower

PDB-8pr2: 
Cytoplasmic dynein-1 heavy chain bound to JIP3-LZI

PDB-8pr3: 
Cytoplasmic dynein-1 heavy chain bound to JIP3-RH1

PDB-8pr4: 
Dynactin pointed end bound to JIP3

PDB-8pr5: 
Structure of the autoinhibited dynactin p150glued projection

PDB-8ptk: 
Composite structure of Dynein-Dynactin-JIP3-LIS1
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
