-検索条件
-検索結果
検索 (著者・登録者: leis & a)の結果68件中、1から50件目までを表示しています

EMDB-37260: 
Cryo-EM structure of Myosin VI in the autoinhibited state (without SAH-extension density)

EMDB-37261: 
Cryo-EM map of Myosin VI in the autoinhibited state (with SAH-extension density)

PDB-8w41: 
Cryo-EM structure of Myosin VI in the autoinhibited state

EMDB-41805: 
Cryo-EM structure of murine Thrombopoietin receptor ectodomain in complex with Tpo

PDB-8u18: 
Cryo-EM structure of murine Thrombopoietin receptor ectodomain in complex with Tpo

EMDB-42804: 
Structure of nucleotide-free Pediculus humanus (Ph) PINK1 dimer

EMDB-42806: 
Structure of AMP-PNP-bound Pediculus humanus (Ph) PINK1 dimer

EMDB-42807: 
Structure of ADP-bound and phosphorylated Pediculus humanus (Ph) PINK1 dimer

PDB-8uyf: 
Structure of nucleotide-free Pediculus humanus (Ph) PINK1 dimer

PDB-8uyh: 
Structure of AMP-PNP-bound Pediculus humanus (Ph) PINK1 dimer

PDB-8uyi: 
Structure of ADP-bound and phosphorylated Pediculus humanus (Ph) PINK1 dimer

EMDB-27641: 
The structure of the interleukin 11 signalling complex, truncated gp130

EMDB-27642: 
The structure of the IL-11 signalling complex, with full-length extracellular gp130

PDB-8dps: 
The structure of the interleukin 11 signalling complex, truncated gp130

PDB-8dpt: 
The structure of the IL-11 signalling complex, with full-length extracellular gp130

EMDB-40272: 
BtCoV-422 in complex with neutralizing antibody JC57-11

EMDB-40306: 
BtCoV-422 spike in complex with JC57-11 Fab, global map

EMDB-40310: 
BtCoV-422 spike in complex with JC57-11 Fab, local map

PDB-8sak: 
BtCoV-422 in complex with neutralizing antibody JC57-11

EMDB-18301: 
In-tissue cryo electron tomograms of App^NL-G-F amyloid plaques

EMDB-16018: 
Sarkosyl-extracted AppNL-G-F Abeta42 fibril structure

EMDB-16019: 
Sarkosyl-extracted AppNL-G-F Abeta42 fibril structure (Methoxy-X04-labelled mice)

PDB-8bfa: 
Sarkosyl-extracted AppNL-G-F Abeta42 fibril structure

PDB-8bfb: 
Sarkosyl-extracted AppNL-G-F Abeta42 fibril structure (Methoxy-X04-labelled mice)

EMDB-17118: 
20S proteasome & CBR3 complex

EMDB-27070: 
apo form Cryo-EM structure of Campylobacter jejune ketol-acid reductoisommerase crosslinked by Glutaraldehyde

EMDB-28189: 
SARS-CoV-2 Spike in complex with biparatopic nanobody BP10
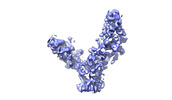
EMDB-28190: 
SARS-CoV-2 RBD in complex with biparatopic nanobody BP10 local refinement

EMDB-14582: 
Human heparan sulfate polymerase complex EXT1-EXT2

PDB-7zay: 
Human heparan sulfate polymerase complex EXT1-EXT2
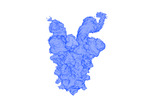
EMDB-25618: 
SARS-CoV-2 VFLIP spike boung to 2 Ab12 Fab fragments
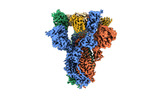
EMDB-25663: 
SARS-CoV-2 S (Spike Glycoprotein) D614G with Three (3) RBDs Up, Bound to Antibody 2-7 scFv, composite map

EMDB-25689: 
SARS-CoV-2 S (Spike Glycoprotein) D614G with Three (3) RBDs Up, Bound to Antibody 2-7 scFv, global map with poorly-resolved RBDs and scFvs

EMDB-25690: 
SARS-CoV-2 S (Spike Glycoprotein) D614G with Three (3) RBDs Up, Bound to Antibody 2-7 scFv, local refinement map

EMDB-25711: 
SARS-CoV-2 S (Spike Glycoprotein) D614G with One(1) RBD Up

PDB-7t3m: 
SARS-CoV-2 S (Spike Glycoprotein) D614G with Three (3) RBDs Up, Bound to Antibody 2-7 scFv, composite map

PDB-7t67: 
SARS-CoV-2 S (Spike Glycoprotein) D614G with One(1) RBD Up

EMDB-25677: 
Structure of dimeric phosphorylated Pediculus humanus (Ph) PINK1

EMDB-25678: 
Structure of dimeric phosphorylated Pediculus humanus (Ph) PINK1 with kinked alpha-C helix in chain B

EMDB-25679: 
Structure of dimeric phosphorylated Pediculus humanus (Ph) PINK1 with extended alpha-C helix in chain B

EMDB-25680: 
Structure of dodecameric unphosphorylated Pediculus humanus (Ph) PINK1 D357A mutant

EMDB-25681: 
Structure of dimeric unphosphorylated Pediculus humanus (Ph) PINK1 D357A mutant

PDB-7t4k: 
Structure of dimeric phosphorylated Pediculus humanus (Ph) PINK1 with kinked alpha-C helix in chain B

PDB-7t4l: 
Structure of dimeric phosphorylated Pediculus humanus (Ph) PINK1 with extended alpha-C helix in chain B

PDB-7t4m: 
Structure of dodecameric unphosphorylated Pediculus humanus (Ph) PINK1 D357A mutant

PDB-7t4n: 
Structure of dimeric unphosphorylated Pediculus humanus (Ph) PINK1 D357A mutant

EMDB-13662: 
Structure of the membrane soluble spike complex from the Lassa virus in a C3-symmetric map

EMDB-13667: 
Structure of the membrane soluble spike complex from the Lassa virus in a C1-symmetric map focused on the ectodomain

PDB-7puy: 
Structure of the membrane soluble spike complex from the Lassa virus in a C3-symmetric map

PDB-7pvd: 
Structure of the membrane soluble spike complex from the Lassa virus in a C1-symmetric map focused on the ectodomain
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
