-検索条件
-検索結果
検索 (著者・登録者: klem & r)の結果105件中、1から50件目までを表示しています
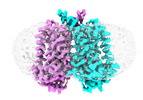
EMDB-43712: 
Human EBP complexed with compound 1
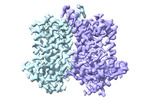
EMDB-43713: 
Human EBP complexed with compound 3a

PDB-8w0r: 
Human EBP complexed with compound 1

PDB-8w0s: 
Human EBP complexed with compound 3a
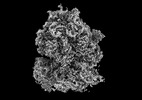
EMDB-17002: 
Human Methionine Aminopeptidase 2 at the 80S ribosome

EMDB-17003: 
Chaetomium thermophilum Methionine Aminopeptidase 2 at the 80S ribosome
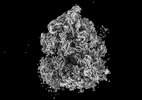
EMDB-17004: 
Chaetomium thermophilum Methionine Aminopeptidase 2 autoproteolysis product at the 80S ribosome

PDB-8ony: 
Human Methionine Aminopeptidase 2 at the 80S ribosome

PDB-8onz: 
Chaetomium thermophilum Methionine Aminopeptidase 2 at the 80S ribosome

PDB-8oo0: 
Chaetomium thermophilum Methionine Aminopeptidase 2 autoproteolysis product at the 80S ribosome

EMDB-16801: 
Homo sapiens Get1/Get2 heterotetramer in complex with a Get3 dimer

EMDB-16802: 
Homo sapiens Get1/Get2 heterotetramer (a3' deletion variant) in complex with a Get3 dimer

EMDB-16817: 
Chaetomium thermophilum Get1/Get2 heterotetramer in complex with a Get3 dimer (amphipol)

EMDB-16819: 
Chaetomium thermophilum Get1/Get2 heterotetramer in complex with a Get3 dimer (nanodisc)

PDB-8cr1: 
Homo sapiens Get1/Get2 heterotetramer in complex with a Get3 dimer

PDB-8cr2: 
Homo sapiens Get1/Get2 heterotetramer (a3' deletion variant) in complex with a Get3 dimer

PDB-8odu: 
Chaetomium thermophilum Get1/Get2 heterotetramer in complex with a Get3 dimer (amphipol)

PDB-8odv: 
Chaetomium thermophilum Get1/Get2 heterotetramer in complex with a Get3 dimer (nanodisc)

EMDB-14479: 
Cryo-EM structure of the ribosome-associated RAC complex on the 80S ribosome - RAC-1 conformation

EMDB-14480: 
Cryo-EM structure of the ribosome-associated RAC complex on the 80S ribosome - RAC-2 conformation

PDB-7z3n: 
Cryo-EM structure of the ribosome-associated RAC complex on the 80S ribosome - RAC-1 conformation

PDB-7z3o: 
Cryo-EM structure of the ribosome-associated RAC complex on the 80S ribosome - RAC-2 conformation

EMDB-29019: 
Alpha1/BetaB Heteromeric Glycine Receptor in 1 mM Glycine 20 uM Ivermectin State

PDB-8fe1: 
Alpha1/BetaB Heteromeric Glycine Receptor in 1 mM Glycine 20 uM Ivermectin State

EMDB-26130: 
Alpha1/BetaB Heteromeric Glycine Receptor in Strychnine-Bound State
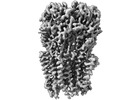
EMDB-26141: 
Alpha1/BetaB Heteromeric Glycine Receptor in Glycine-Bound State

EMDB-15136: 
In-cell structure of the actin filament Arp2/3 complex branch junction in lamellipodia of WT B16-F1 mouse melanoma cells

EMDB-15137: 
Tomogram of a lamellipodium of a WT B16-F1 mouse melanoma cell

EMDB-15138: 
In-cell structure of the actin filament Arp2/3 complex branch junction in lamellipodia of ArpC5 knockout B16-F1 mouse melanoma cells

EMDB-15139: 
Tomogram of a lamellipodium of an ArpC5 knockout B16-F1 mouse melanoma cell

EMDB-15140: 
In-cell structure of the actin filament Arp2/3 complex branch junction in lamellipodia of ArpC5L knockout B16-F1 mouse melanoma cells

EMDB-15141: 
Tomogram of a lamellipodium of an ArpC5L knockout B16-F1 mouse melanoma cell
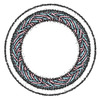
EMDB-14630: 
Membrane-bound CHMP2A-CHMP3 filament (430 Angstrom diameter)
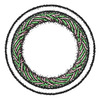
EMDB-14631: 
Membrane-bound CHMP2A-CHMP3 filament (410 Angstrom diameter)

PDB-7zcg: 
CHMP2A-CHMP3 heterodimer (430 Angstrom diameter)

PDB-7zch: 
CHMP2A-CHMP3 heterodimer (410 Angstrom diameter)

EMDB-12144: 
Brevibacterium linens encapsulin structure

PDB-7bcv: 
Brevibacterium linens encapsulin structure

EMDB-26732: 
Cryo-EM structure of WAVE Regulatory Complex

EMDB-26733: 
Cryo-EM structure of D-site Rac1-bound WAVE Regulatory Complex
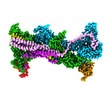
EMDB-26734: 
Cryo-EM structure of WAVE regulatory complex with Rac1 bound on both A and D site

PDB-7usc: 
Cryo-EM structure of WAVE Regulatory Complex

PDB-7usd: 
Cryo-EM structure of D-site Rac1-bound WAVE Regulatory Complex

PDB-7use: 
Cryo-EM structure of WAVE regulatory complex with Rac1 bound on both A and D site

EMDB-26360: 
Structure of E. coli dGTPase bound to T7 bacteriophage protein Gp1.2

EMDB-26361: 
Structure of E. coli dGTPase bound to T7 bacteriophage protein Gp1.2 and dGTP

EMDB-26362: 
Structure of E. coli dGTPase bound to T7 bacteriophage protein Gp1.2 and GTP

PDB-7u65: 
Structure of E. coli dGTPase bound to T7 bacteriophage protein Gp1.2

PDB-7u66: 
Structure of E. coli dGTPase bound to T7 bacteriophage protein Gp1.2 and dGTP

PDB-7u67: 
Structure of E. coli dGTPase bound to T7 bacteriophage protein Gp1.2 and GTP
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
