-検索条件
-検索結果
検索 (著者・登録者: kim & ds)の結果247件中、1から50件目までを表示しています

EMDB-42116: 
HCN1 complex with propofol

EMDB-42117: 
HCN1 nanodisc

EMDB-44425: 
HCN1 M305L with propofol

EMDB-44426: 
HCN1 M305L holo

EMDB-40825: 
10E8-GT10.2 immunogen in complex with human Fab 10E8 and mouse Fab W6-10

PDB-8sx3: 
10E8-GT10.2 immunogen in complex with human Fab 10E8 and mouse Fab W6-10

EMDB-35234: 
Cryo-EM structure of Acipimox bound human hydroxy-carboxylic acid receptor 2 in complex with Gi heterotrimer

EMDB-35235: 
Cryo-EM structure of GSK256073 bound human hydroxy-carboxylic acid receptor 2 in complex with Gi heterotrimer

EMDB-36900: 
Cryo-EM structure of niacin bound human hydroxy-carboxylic acid receptor 2 (Local refinement)

EMDB-36901: 
Cryo-EM structure of Acipimox bound human hydroxy-carboxylic acid receptor 2 (Local refinement)

EMDB-36902: 
Cryo-EM structure of GSK256073 bound human hydroxy-carboxylic acid receptor 2 (Local refinement)

EMDB-41075: 
SARS-CoV-2 spike in complex with Fab 71281-33

EMDB-41076: 
SARS-CoV-2 spike in complex with Fab 71281-33 (2)

EMDB-34437: 
Cryo-EM structure of niacin bound human hydroxy-carboxylic acid receptor 2 in complex with Gi heterotrimer

EMDB-17756: 
Structure of the murine trace amine-associated receptor TAAR7f bound to N,N-dimethylcyclohexylamine (DMCH) in complex with mini-Gs trimeric G protein

PDB-8pm2: 
Structure of the murine trace amine-associated receptor TAAR7f bound to N,N-dimethylcyclohexylamine (DMCH) in complex with mini-Gs trimeric G protein

EMDB-28756: 
Structure of SARS-CoV-2 Omicron BA.1 spike in complex with antibody Fab 1C3

EMDB-28757: 
Structure of SARS-CoV-2 spike with antibody Fabs 2A10 and 1H2 (Local refinement of the RBD and Fabs 1H2 and 2A10)

EMDB-28763: 
Negative stain EM map of SARS-CoV-2 D614G Spike in complex with 2A10 IgG

EMDB-28764: 
Negative stain EM map of SARS-CoV-2 D614G Spike in complex with 4H4 IgG

EMDB-28765: 
Negative stain EM map of SARS-CoV-2 D614G Spike in complex with 1C3 IgG

EMDB-28769: 
Negative stain EM map of SARS-CoV-2 Omicron BA.1 Spike in complex with 1C3 IgG

EMDB-28770: 
Negative stain EM map of SARS-CoV-2 D614G Spike in complex with 2G3 IgG

EMDB-28771: 
Negative stain EM map of SARS-CoV-2 D614G Spike in complex with 2E6 IgG

EMDB-28772: 
Negative stain EM map of SARS-CoV-2 D614G Spike in complex with 1H2 Fab

EMDB-28773: 
Negative stain EM map of SARS-CoV-2 D614G Spike in complex with 1G8 IgG

EMDB-31722: 
Cryo-EM structure of the mouse ABCB9 (ADP.BeF3-bound)

EMDB-31723: 
Cryo-EM structure of the mouse ABCB9 (PG-bound)
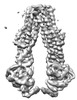
EMDB-31955: 
Cryo-EM structure of the mouse TAPL (9mer-peptide bound)

EMDB-25621: 
The Envelope Glycoprotein SIVmac239.K180S SOSIP trimer in complex with 3 copies of the neutralizing antibody K11
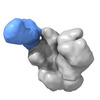
EMDB-25623: 
SIVmac239.K180S SOSIP in complex with FZ019.2 Fab

EMDB-25624: 
SIVmac239.K180S SOSIP in complex with FZ012.7 Fab

EMDB-25625: 
SIVmac239.K180S SOSIP in complex with an unknown Fab from the polyclonal serum of the SIV-infected Rhesus Macaque Rh33519

EMDB-25626: 
SIVmac239.K180S SOSIP in complex with an unknown Fab from the polyclonal serum of the SIV-infected Rhesus Macaque Rh31186

EMDB-25627: 
SIVmac239.K180S SOSIP in complex with an unknown Fab from the polyclonal serum of the SIV-infected Rhesus Macaque Rh34620

EMDB-25628: 
SIVmac239.K180S SOSIP in complex with an unknown Fab from the polyclonal serum of the SIV-infected Rhesus Macaque r11008

EMDB-25629: 
SIVmac239.K180S SOSIP in complex with an unknown Fab from the polyclonal serum of the SIV-infected Rhesus Macaque r11008

EMDB-25630: 
SIVmac239.K180S SOSIP in complex with an unknown Fab from the polyclonal serum of the SIV-infected Rhesus Macaque r11002

EMDB-25631: 
SIVmac239.K180S SOSIP in complex with an unknown Fab from the polyclonal serum of the SIV-infected Rhesus Macaque r11004

EMDB-25632: 
SIVmac239.K180S SOSIP in complex with an unknown Fab from the polyclonal serum of the SIV-infected Rhesus Macaque r11004
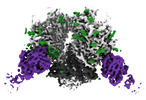
EMDB-25676: 
The Envelope Glycoprotein SIVmac239.K180S SOSIP trimer in complex with 3 copies of the neutralizing antibody K11

PDB-7t2p: 
The Envelope Glycoprotein SIVmac239.K180S SOSIP trimer in complex with 3 copies of the neutralizing antibody K11

PDB-7t4g: 
The Envelope Glycoprotein SIVmac239.K180S SOSIP trimer in complex with 3 copies of the neutralizing antibody K11

EMDB-27502: 
Cryo-EM structure of SARS-CoV-2 Alpha (B.1.1.7) spike protein

EMDB-27503: 
Cryo-EM structure of SARS-CoV-2 Alpha (B.1.1.7) spike protein in complex with human ACE2

EMDB-27504: 
Cryo-EM structure of SARS-CoV-2 Alpha (B.1.1.7) spike protein in complex with human ACE2 (focused refinement of RBD and ACE2)

EMDB-27505: 
Cryo-EM structure of SARS-CoV-2 Beta (B.1.351) spike protein

EMDB-27506: 
Cryo-EM structure of SARS-CoV-2 Beta (B.1.351) spike protein in complex with human ACE2

EMDB-27507: 
Cryo-EM structure of SARS-CoV-2 Beta (B.1.351) spike protein in complex with human ACE2 (focused refinement of RBD and ACE2)

EMDB-27508: 
Cryo-EM structure of SARS-CoV-2 Gamma (P.1) spike protein
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
