-検索条件
-検索結果
検索 (著者・登録者: hanke & a)の結果51件中、1から50件目までを表示しています

EMDB-16375: 
SARS-CoV2 Omicron BA.1 RBD in complex with CAB-A17 antibody

PDB-8c0y: 
SARS-CoV2 Omicron BA.1 RBD in complex with CAB-A17 antibody

EMDB-42970: 
Model and map from local refinement of a CAB-A17 - Omicron Ba.1 spike complex

PDB-8v4f: 
Model and map from local refinement of a CAB-A17 - Omicron Ba.1 spike complex
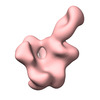
EMDB-42464: 
chEnv TTT protein in complex with 43A2 Fab

EMDB-42468: 
chEnv TTT protein in complex with CM01A Fab
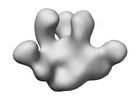
EMDB-43664: 
Triple tandem trimer immunogens for HIV-1 and influenza nucleic acid-based vaccines
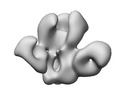
EMDB-43665: 
Triple tandem trimer immunogens for HIV-1 and influenza nucleic acid-based vaccines (cH125 TTT)

EMDB-43666: 
Triple tandem trimer immunogens for HIV-1 and influenza nucleic acid-based vaccines (H2/1 GCN4)

EMDB-43668: 
Triple tandem trimer immunogens for HIV-1 and influenza nucleic acid-based vaccines (H5/1 GCN4)

EMDB-43669: 
Triple tandem trimer immunogens for HIV-1 and influenza nucleic acid-based vaccines. H5 GCN4

EMDB-15270: 
SARS Cov2 Spike RBD in complex with Fab47

PDB-8a95: 
SARS Cov2 Spike RBD in complex with Fab47

EMDB-34551: 
Cryo-EM structure of HACE1 monomer

EMDB-34586: 
Cryo-EM structure of HACE1 dimer

PDB-8h8x: 
Cryo-EM structure of HACE1 monomer

PDB-8hae: 
Cryo-EM structure of HACE1 dimer

EMDB-15273: 
SARS Cov2 Spike in 1-up conformation complex with Fab47

PDB-8a99: 
SARS Cov2 Spike in 1-up conformation complex with Fab47

EMDB-15269: 
SARS CoV2 Spike in the 2-up state in complex with Fab47

EMDB-15271: 
SARS Cov2 Spike RBD in complex with Fab47

PDB-8a94: 
SARS CoV2 Spike in the 2-up state in complex with Fab47.

PDB-8a96: 
SARS Cov2 Spike RBD in complex with Fab47
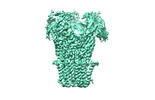
EMDB-13791: 
Pentameric ligand-gated ion channel, DeCLIC at pH 7 with 10 mM Ca2+
EMDB-13792: 
Pentameric ligand-gated ion channel, DeCLIC at pH 7 with 10 mM EDTA

PDB-7q3g: 
Pentameric ligand-gated ion channel, DeCLIC at pH 7 with 10 mM Ca2+

PDB-7q3h: 
Pentameric ligand-gated ion channel, DeCLIC at pH 7 with 10 mM EDTA

EMDB-12465: 
SARS-CoV-2 Spike RBD (dimer) in complex with two Fu2 nanobodies

EMDB-12561: 
SARS-CoV-2 Spike (dimers) in complex with six Fu2 nanobodies

PDB-7nll: 
SARS-CoV-2 Spike RBD (dimer) in complex with two Fu2 nanobodies

PDB-7ns6: 
SARS-CoV-2 Spike (dimers) in complex with six Fu2 nanobodies

EMDB-11978: 
Nanobody E bound to Spike-RBD in a localized reconstruction

EMDB-11981: 
SARS-CoV-spike bound to two neutralising nanobodies

PDB-7b14: 
Nanobody E bound to Spike-RBD in a localized reconstruction

PDB-7b18: 
SARS-CoV-spike bound to two neutralising nanobodies

EMDB-23522: 
TRiC-substrate complex in ATP/AlFx closed state

EMDB-23526: 
Human TRiC/CCT complex with reovirus outer capsid protein sigma-3

PDB-7lum: 
Human TRiC in ATP/AlFx closed state

PDB-7lup: 
Human TRiC/CCT complex with reovirus outer capsid protein sigma-3

EMDB-11980: 
SARS-CoV-spike RBD bound to two neutralising nanobodies.

PDB-7b17: 
SARS-CoV-spike RBD bound to two neutralising nanobodies.

EMDB-23018: 
SARS-CoV-2 spike in complex with nanobodies E

PDB-7ksg: 
SARS-CoV-2 spike in complex with nanobodies E

EMDB-11616: 
Cryo-EM structure of the SARS-CoV-2 spike protein bound to neutralizing sybodies (Sb23)

PDB-7a25: 
Cryo-EM structure of the SARS-CoV-2 spike protein bound to neutralizing sybodies (Sb23)

EMDB-11617: 
Cryo-EM structure of the SARS-CoV-2 spike protein bound to neutralizing sybodies (Sb23) 2-up conformation

PDB-7a29: 
Cryo-EM structure of the SARS-CoV-2 spike protein bound to neutralizing sybodies (Sb23) 2-up conformation

EMDB-11526: 
Cryo-EM structure of the SARS-CoV-2 spike protein bound to neutralizing nanobodies (Ty1)

EMDB-11728: 
SARS-CoV-2 spike glycoprotein bound to nanobodies (Ty1)

PDB-6zxn: 
Cryo-EM structure of the SARS-CoV-2 spike protein bound to neutralizing nanobodies (Ty1)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
