-検索条件
-検索結果
検索 (著者・登録者: faust & b)の結果79件中、1から50件目までを表示しています

EMDB-18892: 
Lipid droplet-Vacuole contacts in Ldo16 overexpression yeast strain.

EMDB-18893: 
Lipid droplet-vacuole and Nucleus-vacuole contacts in WT yeast cell starved for 4 hours

EMDB-18894: 
Lipid droplet lipophagy in 4-hour starved WT yeast cell.

EMDB-18895: 
Multiple vacuole-lipid droplet-nucleus contacts in 4-hour starved WT yeast cell.

EMDB-18896: 
Lipophagy in 5-day starved WT yeast cell.

EMDB-18897: 
Lipid droplets in proximity to a vacuole in dLdo strain cell after 5-day starvation.

EMDB-18898: 
Vacuolar contents of WT cell after 5-day starvation.

EMDB-18899: 
Lipid droplet-nucleus contacts in dLdo yeast strain after 5-day starvation.

EMDB-29493: 
Cryo-EM structure of human Needle/NAIP/NLRC4 (R288A)

EMDB-29496: 
Cryo-EM structure of full-length human NLRC4 inflammasome with C11 symmetry

EMDB-29498: 
Cryo-EM structure of full-length human NLRC4 inflammasome with C12 symmetry

PDB-8fvu: 
Cryo-EM structure of human Needle/NAIP/NLRC4 (R288A)

PDB-8fw2: 
Cryo-EM structure of full-length human NLRC4 inflammasome with C11 symmetry

PDB-8fw9: 
Cryo-EM structure of full-length human NLRC4 inflammasome with C12 symmetry

EMDB-27943: 
9H2 Fab-poliovirus 1 complex
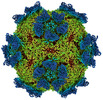
EMDB-27947: 
9H2 Fab-Sabin poliovirus 3 complex

EMDB-27948: 
9H2 Fab-poliovirus 2 complex
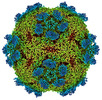
EMDB-27949: 
9H2 Fab-Sabin poliovirus 3 complex
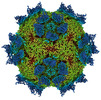
EMDB-27950: 
9H2 Fab-Sabin poliovirus 2 complex
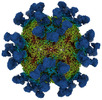
EMDB-27951: 
9H2 Fab-Sabin poliovirus 1 complex

PDB-8e8l: 
9H2 Fab-poliovirus 1 complex

PDB-8e8r: 
9H2 Fab-Sabin poliovirus 3 complex

PDB-8e8s: 
9H2 Fab-poliovirus 2 complex

PDB-8e8x: 
9H2 Fab-Sabin poliovirus 3 complex

PDB-8e8y: 
9H2 Fab-Sabin poliovirus 2 complex

PDB-8e8z: 
9H2 Fab-Sabin poliovirus 1 complex

EMDB-28900: 
Propionate bound to human olfactory receptor OR51E2 in complex with miniGs399 (transmembrane domain)

EMDB-28896: 
Human olfactory receptor OR51E2 bound to propionate in complex with miniGs399

PDB-8f76: 
Human olfactory receptor OR51E2 bound to propionate in complex with miniGs399

EMDB-27177: 
sd1.040 Fab in complex with SARS-CoV-2 Spike 2P glycoprotein

PDB-8d48: 
sd1.040 Fab in complex with SARS-CoV-2 Spike 2P glycoprotein
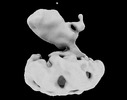
EMDB-27640: 
Org 2274179-0-bound Thyrotropin Receptor

EMDB-27647: 
Human thyrotropin analog TR1402 bound to human Thyrotropin receptor (extracellular domain map only)

EMDB-27648: 
Human thyrotropin analog TR1402 bound to human Thyrotropin receptor (transmembrane domain and G protein map only)
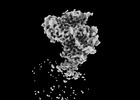
EMDB-27649: 
Native human TSH bound to human Thyrotropin receptor (extracellular domain map only)

EMDB-27650: 
Native human TSH bound to human Thyrotropin receptor (transmembrane domain and G protein map only)
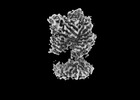
EMDB-27651: 
M22 Fab bound to human Thyrotropin receptor (extracellular domain map only)

EMDB-27652: 
M22 Fab bound to human Thyrotropin receptor (transmembrane domain and G protein map only)

EMDB-25762: 
Human Thyrotropin receptor bound by CS-17 Inverse Agonist Fab/Org 274179-0 Antagonist

PDB-7t9m: 
Human Thyrotropin receptor bound by CS-17 Inverse Agonist Fab/Org 274179-0 Antagonist

EMDB-25758: 
Native human TSH bound to human Thyrotropin receptor in complex with miniGs399 (composite structure)

EMDB-25763: 
M22 Agonist Autoantibody bound to Human Thyrotropin receptor in complex with miniGs399 (composite structure)

EMDB-26795: 
Human thyrotropin analog TR1402 bound to human Thyrotropin receptor in complex with miniGs399 (composite structure)
PDB-7t9i: 
Native human TSH bound to human Thyrotropin receptor in complex with miniGs399 (composite structure)

PDB-7t9n: 
M22 Agonist Autoantibody bound to Human Thyrotropin receptor in complex with miniGs399 (composite structure)

PDB-7utz: 
Human thyrotropin analog TR1402 bound to human Thyrotropin receptor in complex with miniGs399 (composite structure)

EMDB-25479: 
2.3 A structure of the ATP-dependent chromatin remodeler Chd1 bound to the nucleosome in a nucleotide-free state

EMDB-25480: 
2.7 A structure of the ATP-dependent chromatin remodeler Chd1 bound to the nucleosome in a nucleotide-free state. This entry contains a better resolved DNA-binding domain

EMDB-25481: 
2.6 A structure of the nucleosome from a class without Chd1

EMDB-25483: 
2.9 A structure of the nucleosome-bound ChEx, part of the chromatin remodeler Chd1
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
