-検索条件
-検索結果
検索 (著者・登録者: armache & j)の結果141件中、1から50件目までを表示しています

EMDB-42977: 
Cryo-EM structure of singly-bound SNF2h-nucleosome complex with SNF2h at inactive SHL2 (conformation 1)

EMDB-43000: 
Cryo-EM structure of SNF2h-nucleosome complex (consensus structure)

EMDB-43001: 
Cryo-EM structure of SNF2h-nucleosome complex (single-bound structure)

EMDB-43002: 
Cryo-EM structure of doubly-bound SNF2h-nucleosome complex

EMDB-43003: 
Cryo-EM structure of singly-bound SNF2h-nucleosome complex with SNF2h at inactive SHL2 (conformation 2)

EMDB-43004: 
Cryo-EM structure of doubly-bound SNF2h-nucleosome complex (conformation 1)

EMDB-43005: 
Cryo-EM structure of doubly-bound SNF2h-nucleosome complex (conformation 2)

PDB-8v4y: 
Cryo-EM structure of singly-bound SNF2h-nucleosome complex with SNF2h at inactive SHL2 (conformation 1)

PDB-8v6v: 
Cryo-EM structure of doubly-bound SNF2h-nucleosome complex

PDB-8v7l: 
Cryo-EM structure of singly-bound SNF2h-nucleosome complex with SNF2h at inactive SHL2 (conformation 2)

EMDB-41111: 
Catalytic and non-catalytic mechanisms of histone H4 lysine 20 methyltransferase SUV420H1

PDB-8t9h: 
Catalytic and non-catalytic mechanisms of histone H4 lysine 20 methyltransferase SUV420H1

EMDB-41109: 
Catalytic and non-catalytic mechanisms of histone H4 lysine 20 methyltransferase SUV420H1

EMDB-41113: 
Catalytic and non-catalytic mechanisms of histone H4 lysine 20 methyltransferase SUV420H1

EMDB-41259: 
Catalytic and non-catalytic mechanisms of histone H4 lysine 20 methyltransferase SUV420H1
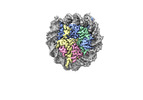
EMDB-41272: 
Catalytic and non-catalytic mechanisms of histone H4 lysine 20 methyltransferase SUV420H1

PDB-8t9f: 
Catalytic and non-catalytic mechanisms of histone H4 lysine 20 methyltransferase SUV420H1

PDB-8thu: 
Catalytic and non-catalytic mechanisms of histone H4 lysine 20 methyltransferase SUV420H1

EMDB-40789: 
BAP1/ASXL1 bound to the H2AK119Ub Nucleosome

EMDB-40790: 
Map focused on acidic patch BAP1/ASXL1 bound to the H2AK119Ub Nucleosome

EMDB-40791: 
Overall map of BAP1/ASXL1 bound to the H2AK119Ub Nucleosome

PDB-8svf: 
BAP1/ASXL1 bound to the H2AK119Ub Nucleosome

EMDB-29735: 
Structure of nucleosome-bound Sirtuin 6 deacetylase

PDB-8g57: 
Structure of nucleosome-bound Sirtuin 6 deacetylase

EMDB-28162: 
SARS-CoV-2 polyprotein substrate regulates the stepwise Mpro cleavage reaction

EMDB-28200: 
Cryo-EM structure of SARS CoV-2 Mpro WT protease

PDB-8eir: 
SARS-CoV-2 polyprotein substrate regulates the stepwise Mpro cleavage reaction

PDB-8eke: 
Cryo-EM structure of SARS CoV-2 Mpro WT protease

EMDB-25479: 
2.3 A structure of the ATP-dependent chromatin remodeler Chd1 bound to the nucleosome in a nucleotide-free state

EMDB-25480: 
2.7 A structure of the ATP-dependent chromatin remodeler Chd1 bound to the nucleosome in a nucleotide-free state. This entry contains a better resolved DNA-binding domain

EMDB-25481: 
2.6 A structure of the nucleosome from a class without Chd1

EMDB-25483: 
2.9 A structure of the nucleosome-bound ChEx, part of the chromatin remodeler Chd1

PDB-7swy: 
2.6 A structure of a 40-601[TA-rich+1]-40 nucleosome

PDB-7tn2: 
Composite model of a Chd1-nucleosome complex in the nucleotide-free state derived from 2.3A and 2.7A Cryo-EM maps

EMDB-23529: 
Structure of the Marseillevirus nucleosome

EMDB-23530: 
Marseillevirus heterotrimeric (hexameric) nucleosome
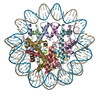
PDB-7lv8: 
Structure of the Marseillevirus nucleosome

PDB-7lv9: 
Marseillevirus heterotrimeric (hexameric) nucleosome

EMDB-22691: 
Active state Dot1 bound to the unacetylated H4 nucleosome

EMDB-22692: 
Active state Dot1 bound to the H4K16ac nucleosome

EMDB-22693: 
Noncatalytic conformation Dot1 bound to the unacetylated H4 nucleosome

EMDB-22694: 
Active state Dot1 bound to the H4K16ac nucleosome, Ubiquitin focused map

EMDB-22695: 
Active state Dot1 bound to the unacetylated H4 nucleosome, Ubiquitin focused map

PDB-7k6p: 
Active state Dot1 bound to the unacetylated H4 nucleosome

PDB-7k6q: 
Active state Dot1 bound to the H4K16ac nucleosome

EMDB-23021: 
Cryo-EM structure of PRC2:EZH1-AEBP2-JARID2

EMDB-23022: 
Cryo-EM map of PRC2:EZH1-AEBP2

EMDB-23024: 
PRC2:EZH1_A from a dimeric PRC2 bound to a nucleosome

EMDB-23025: 
PRC2:EZH1_B from a dimeric PRC2 bound to a nucleosome

EMDB-23026: 
Nucleosome from a dimeric PRC2 bound to a nucleosome
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
