2023年日本バイオインフォマティクス学会年会・第12回生命医薬情報学連合大会(IIBMP2023)にてライフサイエンス統合データベースセンター(DBCLS)と合同でランチョンセミナーを開催します。詳細は以下の通りです。皆さまのお越しをお待ちしております。
- 日時
- 2023年9月8日(金)12:30~13:30
- 場所
- 柏の葉カンファレンスセンター(千葉県柏市若柴178番地4: GoogleMap)第3会場
- 開催方式
- 対面のみ
- 定員
- 130名
- 参加費
- 無料(別途学会参加費は必要)
- 参加方法
- 当日8時30分より受付にて配布する整理券を事前に受け取り、セミナー開始前に会場受付にて整理券をお渡しください。
- セミナータイトル
- 生命科学におけるデータサイエンス促進のための基盤技術開発と最新動向
- 講演言語
- 日本語
- 講演者
-
- 片山 俊明(ライフサイエンス統合データベースセンター・大阪大学蛋白質研究所)
- 横地 政志(蛋白質研究奨励会)
- 講演要旨
-
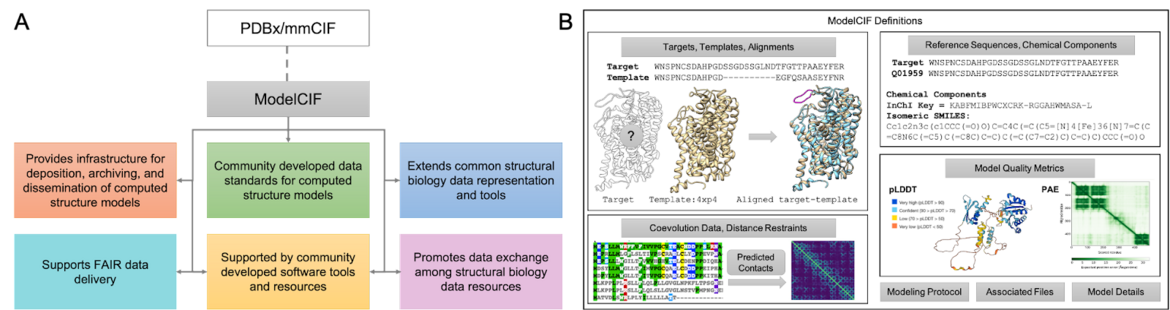
柏の葉キャンパスにあるライフサイエンス統合データベースセンター(DBCLS)は、大学共同利用 機関法人の情報・システム研究機構においてデータ共有支援と基盤技術研究開発を担う研究センターです。DBCLS では、多様な生命医科学のデータベースの知識グラフによるデータ統合とソフトウェアおよびサービスの開発を行っています。大阪大学の蛋白質研究所は蛋白質研究奨励会とも連携し、日本蛋白質構造データバンク(PDBj)の開発と運営を行っています。PDBj は、今年 20周年を迎えた国際蛋白質構造データバンク(wwPDB)の一員であり、その知識グラフ表現の開発、維持に関しては、PDBj が主導しています。
生命科学の分野では生命の階層性を反映して多様なデータベースが作成されていますが、生命システムの理解のためには、これのデータベースを統合的に繋いで活用することが求められるようになっています。例えば、疾患情報とゲノム情報を組み合わせて疾患関連遺伝子を推定できるかもしれませんが、疾患の仕組みを深く理解し、効果的な創薬研究を進めるためには、蛋白質の立体構造情報が欠かせません。また、遺伝子のバリアント、発現調節、修飾、分子間の相互作用、およびパスウェイなど、最新の情報が集約された基盤も重要な役割を果たします。DBCLS とPDBj は、JST/NBDC の基盤技術開発プログラム・統合化推進プログラムを受けて研究開発を進めており、本ランチョンセミナーではこれまでの基盤整備の現状と展望および最新情報をご紹介します。
関連項目




 OneDepのORCiDログインボタン(赤枠で囲まれた部分)
OneDepのORCiDログインボタン(赤枠で囲まれた部分)
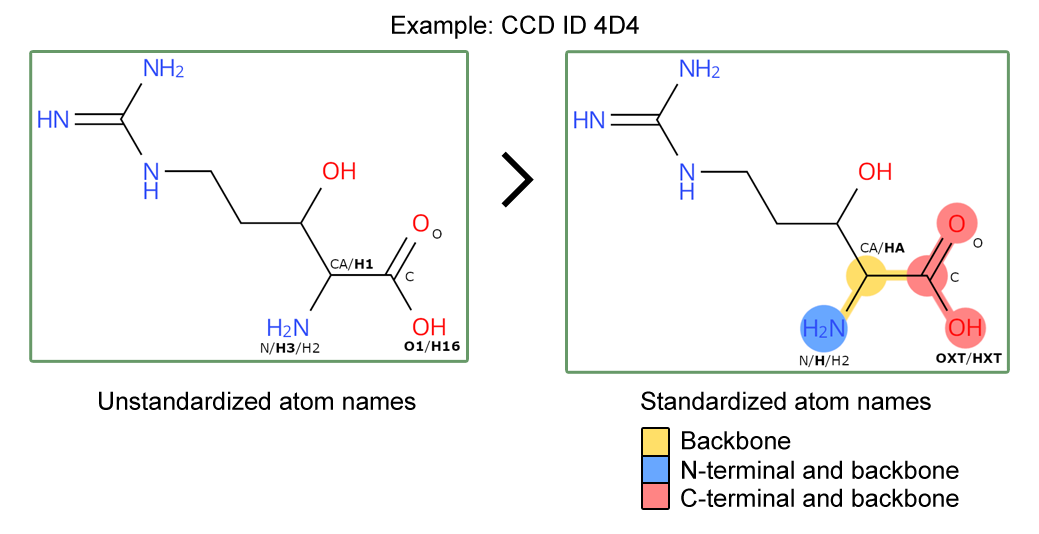 更新されたペプチドCCDには、標準化された原子名と主鎖/N末端およびC末端の注釈が加えられています
更新されたペプチドCCDには、標準化された原子名と主鎖/N末端およびC末端の注釈が加えられています



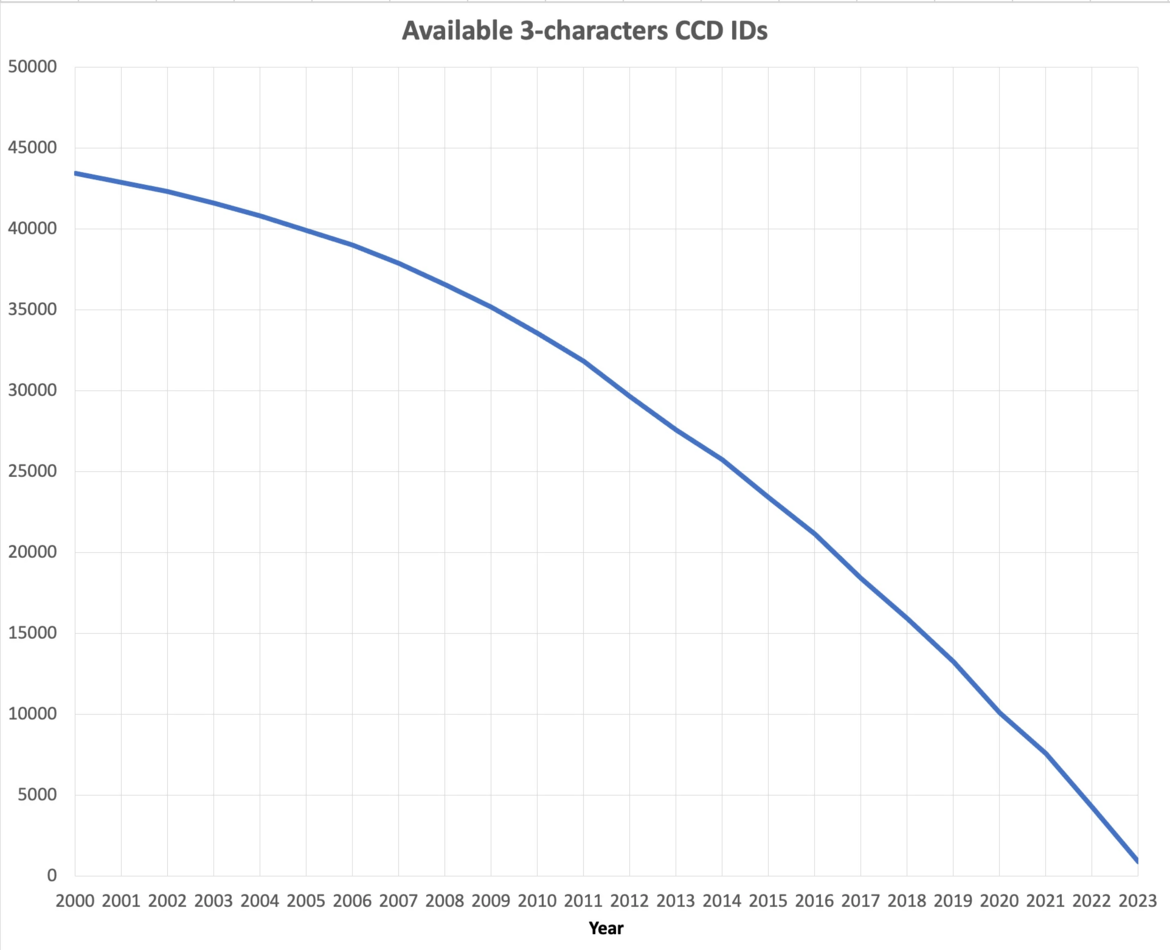 2023年9月時点で利用可能な3文字CCD ID数
2023年9月時点で利用可能な3文字CCD ID数


 wwPDBは2023年に設立20周年を迎えます
wwPDBは2023年に設立20周年を迎えます





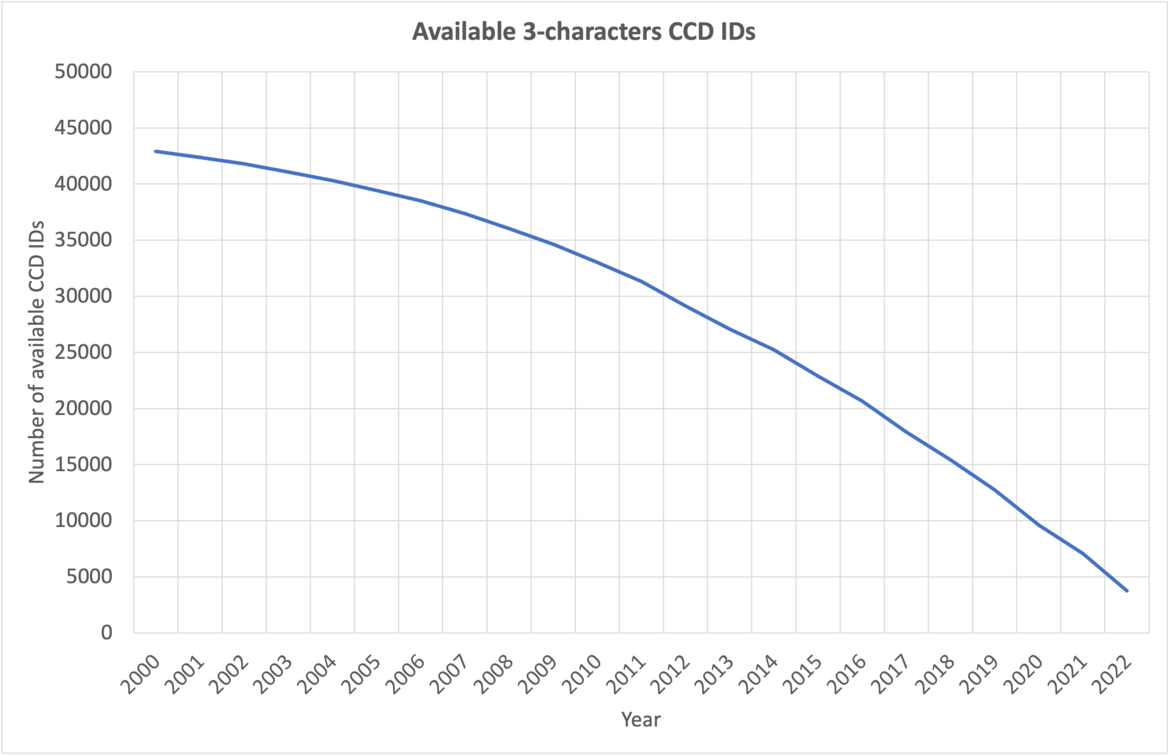 利用可能な3文字のCCD ID数の年推移
利用可能な3文字のCCD ID数の年推移