+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8uqe | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
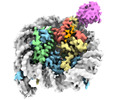
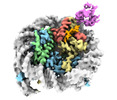
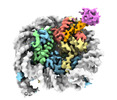
| タイトル | Crystal structure of RNF168 (RING)-UbcH5c fused to H2A-H2B via a 26-residue linker (RING not modeled in density) | |||||||||
 要素 要素 | E3 ubiquitin-protein ligase RNF168,Ubiquitin-conjugating enzyme E2 D3,Histone H2B type 2-E,Histone H2A type 1-B/E | |||||||||
 キーワード キーワード |  TRANSFERASE (転移酵素) / RNF168 / UbcH5c / TRANSFERASE (転移酵素) / RNF168 / UbcH5c /  Histone H2A (ヒストンH2A) / Histone H2A (ヒストンH2A) /  Histone H2B (ヒストンH2B) / Histone H2B (ヒストンH2B) /  Chromatin (クロマチン) / Chromatin (クロマチン) /  Ubiquitin ligase (ユビキチンリガーゼ) / Ubiquitin ligase (ユビキチンリガーゼ) /  Ubiquitin-conjugating enzyme (ユビキチン結合酵素) / DNA damage response / DNA double-strand break repair / Ubiquitin-conjugating enzyme (ユビキチン結合酵素) / DNA damage response / DNA double-strand break repair /  PROTEIN BINDING (タンパク質) / PROTEIN BINDING-Transferase complex PROTEIN BINDING (タンパク質) / PROTEIN BINDING-Transferase complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報histone H2AK15 ubiquitin ligase activity / histone ubiquitin ligase activity / (E3-independent) E2 ubiquitin-conjugating enzyme / protein K6-linked ubiquitination / Signaling by BMP /  クラススイッチ / double-strand break repair via classical nonhomologous end joining / protein K11-linked ubiquitination / DNA repair-dependent chromatin remodeling / K63-linked polyubiquitin modification-dependent protein binding ...histone H2AK15 ubiquitin ligase activity / histone ubiquitin ligase activity / (E3-independent) E2 ubiquitin-conjugating enzyme / protein K6-linked ubiquitination / Signaling by BMP / クラススイッチ / double-strand break repair via classical nonhomologous end joining / protein K11-linked ubiquitination / DNA repair-dependent chromatin remodeling / K63-linked polyubiquitin modification-dependent protein binding ...histone H2AK15 ubiquitin ligase activity / histone ubiquitin ligase activity / (E3-independent) E2 ubiquitin-conjugating enzyme / protein K6-linked ubiquitination / Signaling by BMP /  クラススイッチ / double-strand break repair via classical nonhomologous end joining / protein K11-linked ubiquitination / DNA repair-dependent chromatin remodeling / K63-linked polyubiquitin modification-dependent protein binding / positive regulation of protein targeting to mitochondrion / クラススイッチ / double-strand break repair via classical nonhomologous end joining / protein K11-linked ubiquitination / DNA repair-dependent chromatin remodeling / K63-linked polyubiquitin modification-dependent protein binding / positive regulation of protein targeting to mitochondrion /  ユビキチン結合酵素 / response to ionizing radiation / negative regulation of transcription elongation by RNA polymerase II / protein monoubiquitination / ubiquitin conjugating enzyme activity / protein K63-linked ubiquitination / negative regulation of BMP signaling pathway / ユビキチン結合酵素 / response to ionizing radiation / negative regulation of transcription elongation by RNA polymerase II / protein monoubiquitination / ubiquitin conjugating enzyme activity / protein K63-linked ubiquitination / negative regulation of BMP signaling pathway /  nucleosome binding / protein K48-linked ubiquitination / protein autoubiquitination / protein localization to CENP-A containing chromatin / interstrand cross-link repair / Replacement of protamines by nucleosomes in the male pronucleus / CENP-A containing nucleosome / nucleosome binding / protein K48-linked ubiquitination / protein autoubiquitination / protein localization to CENP-A containing chromatin / interstrand cross-link repair / Replacement of protamines by nucleosomes in the male pronucleus / CENP-A containing nucleosome /  ubiquitin ligase complex / SUMOylation of DNA damage response and repair proteins / epigenetic regulation of gene expression / Packaging Of Telomere Ends / Recognition and association of DNA glycosylase with site containing an affected purine / Cleavage of the damaged purine / Deposition of new CENPA-containing nucleosomes at the centromere / Recognition and association of DNA glycosylase with site containing an affected pyrimidine / Cleavage of the damaged pyrimidine / Inhibition of DNA recombination at telomere / positive regulation of DNA repair / Meiotic synapsis / RNA Polymerase I Promoter Opening / Assembly of the ORC complex at the origin of replication / TICAM1, RIP1-mediated IKK complex recruitment / ubiquitin ligase complex / SUMOylation of DNA damage response and repair proteins / epigenetic regulation of gene expression / Packaging Of Telomere Ends / Recognition and association of DNA glycosylase with site containing an affected purine / Cleavage of the damaged purine / Deposition of new CENPA-containing nucleosomes at the centromere / Recognition and association of DNA glycosylase with site containing an affected pyrimidine / Cleavage of the damaged pyrimidine / Inhibition of DNA recombination at telomere / positive regulation of DNA repair / Meiotic synapsis / RNA Polymerase I Promoter Opening / Assembly of the ORC complex at the origin of replication / TICAM1, RIP1-mediated IKK complex recruitment /  DNAメチル化 / IKK complex recruitment mediated by RIP1 / Condensation of Prophase Chromosomes / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) / SIRT1 negatively regulates rRNA expression / HCMV Late Events / DNAメチル化 / IKK complex recruitment mediated by RIP1 / Condensation of Prophase Chromosomes / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) / SIRT1 negatively regulates rRNA expression / HCMV Late Events /  innate immune response in mucosa / PRC2 methylates histones and DNA / innate immune response in mucosa / PRC2 methylates histones and DNA /  ubiquitin binding / Defective pyroptosis / Negative regulators of DDX58/IFIH1 signaling / HDACs deacetylate histones / ubiquitin binding / Defective pyroptosis / Negative regulators of DDX58/IFIH1 signaling / HDACs deacetylate histones /  Peroxisomal protein import / RNA Polymerase I Promoter Escape / Downregulation of SMAD2/3:SMAD4 transcriptional activity / Regulation of TNFR1 signaling / Nonhomologous End-Joining (NHEJ) / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / protein modification process / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / RING-type E3 ubiquitin transferase / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / NoRC negatively regulates rRNA expression / Oxygen-dependent proline hydroxylation of Hypoxia-inducible Factor Alpha / B-WICH complex positively regulates rRNA expression / G2/M DNA damage checkpoint / Inactivation of CSF3 (G-CSF) signaling / DNA Damage/Telomere Stress Induced Senescence / Metalloprotease DUBs / RMTs methylate histone arginines / Peroxisomal protein import / RNA Polymerase I Promoter Escape / Downregulation of SMAD2/3:SMAD4 transcriptional activity / Regulation of TNFR1 signaling / Nonhomologous End-Joining (NHEJ) / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / protein modification process / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / RING-type E3 ubiquitin transferase / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / NoRC negatively regulates rRNA expression / Oxygen-dependent proline hydroxylation of Hypoxia-inducible Factor Alpha / B-WICH complex positively regulates rRNA expression / G2/M DNA damage checkpoint / Inactivation of CSF3 (G-CSF) signaling / DNA Damage/Telomere Stress Induced Senescence / Metalloprotease DUBs / RMTs methylate histone arginines /  遺伝的組換え / Pre-NOTCH Transcription and Translation / 遺伝的組換え / Pre-NOTCH Transcription and Translation /  nucleosome assembly / double-strand break repair via nonhomologous end joining / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / protein polyubiquitination / Transcriptional regulation of granulopoiesis / ubiquitin-protein transferase activity / structural constituent of chromatin / UCH proteinases / nucleosome assembly / double-strand break repair via nonhomologous end joining / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / protein polyubiquitination / Transcriptional regulation of granulopoiesis / ubiquitin-protein transferase activity / structural constituent of chromatin / UCH proteinases /  ubiquitin protein ligase activity / double-strand break repair / ubiquitin protein ligase activity / double-strand break repair /  ヌクレオソーム / antimicrobial humoral immune response mediated by antimicrobial peptide / Antigen processing: Ubiquitination & Proteasome degradation / E3 ubiquitin ligases ubiquitinate target proteins / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks / site of double-strand break / ヌクレオソーム / antimicrobial humoral immune response mediated by antimicrobial peptide / Antigen processing: Ubiquitination & Proteasome degradation / E3 ubiquitin ligases ubiquitinate target proteins / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks / site of double-strand break /  Neddylation / RUNX1 regulates transcription of genes involved in differentiation of HSCs / chromatin organization / Processing of DNA double-strand break ends / HATs acetylate histones / antibacterial humoral response / Neddylation / RUNX1 regulates transcription of genes involved in differentiation of HSCs / chromatin organization / Processing of DNA double-strand break ends / HATs acetylate histones / antibacterial humoral response /  histone binding / ubiquitin-dependent protein catabolic process / Senescence-Associated Secretory Phenotype (SASP) histone binding / ubiquitin-dependent protein catabolic process / Senescence-Associated Secretory Phenotype (SASP)類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.562 Å 分子置換 / 解像度: 3.562 Å | |||||||||
 データ登録者 データ登録者 | Hu, Q. / Botuyan, M.V. / Mer, G. | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2024 ジャーナル: Mol Cell / 年: 2024タイトル: Mechanisms of RNF168 nucleosome recognition and ubiquitylation. 著者: Qi Hu / Debiao Zhao / Gaofeng Cui / Janarjan Bhandari / James R Thompson / Maria Victoria Botuyan / Georges Mer /  要旨: RNF168 plays a central role in the DNA damage response (DDR) by ubiquitylating histone H2A at K13 and K15. These modifications direct BRCA1-BARD1 and 53BP1 foci formation in chromatin, essential for ...RNF168 plays a central role in the DNA damage response (DDR) by ubiquitylating histone H2A at K13 and K15. These modifications direct BRCA1-BARD1 and 53BP1 foci formation in chromatin, essential for cell-cycle-dependent DNA double-strand break (DSB) repair pathway selection. The mechanism by which RNF168 catalyzes the targeted accumulation of H2A ubiquitin conjugates to form repair foci around DSBs remains unclear. Here, using cryoelectron microscopy (cryo-EM), nuclear magnetic resonance (NMR) spectroscopy, and functional assays, we provide a molecular description of the reaction cycle and dynamics of RNF168 as it modifies the nucleosome and recognizes its ubiquitylation products. We demonstrate an interaction of a canonical ubiquitin-binding domain within full-length RNF168, which not only engages ubiquitin but also the nucleosome surface, clarifying how such site-specific ubiquitin recognition propels a signal amplification loop. Beyond offering mechanistic insights into a key DDR protein, our study aids in understanding site specificity in both generating and interpreting chromatin ubiquitylation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8uqe.cif.gz 8uqe.cif.gz | 80.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8uqe.ent.gz pdb8uqe.ent.gz | 56.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8uqe.json.gz 8uqe.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/uq/8uqe https://data.pdbj.org/pub/pdb/validation_reports/uq/8uqe ftp://data.pdbj.org/pub/pdb/validation_reports/uq/8uqe ftp://data.pdbj.org/pub/pdb/validation_reports/uq/8uqe | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  8smwC  8smxC  8smyC  8smzC  8sn0C  8sn1C  8sn2C  8sn3C  8sn4C  8sn5C  8sn6C  8sn7C  8sn8C  8sn9C  8snaC  8txvC  8txwC  8txxC  8u13C  8u14C  8upfC  8uq8C  8uq9C  8uqaC  8uqbC  8uqcC  8uqdC  5eggS S: 精密化の開始モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 50542.828 Da / 分子数: 1 / 変異: S22R,C85K in UbcH5c / 由来タイプ: 組換発現 詳細: residues 1-94 of RNF168, followed by residues 2-147 of UbcH5c, followed by residues 33-123 of H2B, followed by residues 12-105 of H2A 由来: (組換発現)   Homo sapiens (ヒト) Homo sapiens (ヒト)遺伝子: RNF168, UBE2D3, UBC5C, UBCH5C, H2BC21, H2BFQ, HIST2H2BE, H2AC4, H2AFM, HIST1H2AB, H2AC8, H2AFA, HIST1H2AE 発現宿主:   Escherichia coli (大腸菌) Escherichia coli (大腸菌)参照: UniProt: Q8IYW5, UniProt: P61077, UniProt: Q16778, UniProt: P04908, RING-type E3 ubiquitin transferase,  ユビキチン結合酵素, (E3-independent) E2 ubiquitin-conjugating enzyme ユビキチン結合酵素, (E3-independent) E2 ubiquitin-conjugating enzyme |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.78 Å3/Da / 溶媒含有率: 67.44 % |
|---|---|
結晶化 | 温度: 288 K / 手法: 蒸気拡散法, ハンギングドロップ法 詳細: Crystals were obtained by mixing 2 microliters of the protein fusion (10 mg/mL) in 10 mM HEPES, pH 7.5, 600 mM NaCl, 1 mM TCEP and 2 microliters of the reservoir solution containing 0.1 M ...詳細: Crystals were obtained by mixing 2 microliters of the protein fusion (10 mg/mL) in 10 mM HEPES, pH 7.5, 600 mM NaCl, 1 mM TCEP and 2 microliters of the reservoir solution containing 0.1 M imidazole, pH 6.5, 1.0 M sodium acetate PH範囲: 6.5 |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 19-BM / 波長: 0.9792 Å / ビームライン: 19-BM / 波長: 0.9792 Å |
| 検出器 | タイプ: ADSC QUANTUM 315r / 検出器: CCD / 日付: 2018年11月28日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.9792 Å / 相対比: 1 : 0.9792 Å / 相対比: 1 |
| 反射 | 解像度: 3.562→50 Å / Num. obs: 7433 / % possible obs: 99.32 % / 冗長度: 6.8 % / CC1/2: 0.999 / CC star: 1 / Rmerge(I) obs: 0.1181 / Rpim(I) all: 0.0475 / Rrim(I) all: 0.1277 / Net I/σ(I): 13 |
| 反射 シェル | 解像度: 3.562→3.62 Å / 冗長度: 6.7 % / Rmerge(I) obs: 0.7045 / Mean I/σ(I) obs: 2.46 / Num. unique obs: 720 / CC1/2: 0.901 / CC star: 0.974 / Rpim(I) all: 0.2807 / Rrim(I) all: 0.7607 / % possible all: 98.61 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 5EGG 解像度: 3.562→41.54 Å / SU ML: 0.38 / 交差検証法: FREE R-VALUE / σ(F): 1.35 / 位相誤差: 33.42 / 立体化学のターゲット値: ML
| ||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 3.562→41.54 Å
| ||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー





















 PDBj
PDBj













