+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6y5e | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of human cGAS (K394E) bound to the nucleosome (focused refinement of cGAS-NCP subcomplex) | ||||||||||||||||||
 要素 要素 |
| ||||||||||||||||||
 キーワード キーワード |  IMMUNE SYSTEM (免疫系) / IMMUNE SYSTEM (免疫系) /  cGAS / STING / cGAS / STING /  Nucleosome (ヌクレオソーム) / Nucleosome (ヌクレオソーム) /  Innate Immunity (自然免疫系) / cGMP-AMP Innate Immunity (自然免疫系) / cGMP-AMP | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 cyclic GMP-AMP synthase / 2',3'-cyclic GMP-AMP synthase activity / STING mediated induction of host immune responses / cyclic GMP-AMP synthase / 2',3'-cyclic GMP-AMP synthase activity / STING mediated induction of host immune responses /  傍分泌 / poly-ADP-D-ribose modification-dependent protein binding / cGAS/STING signaling pathway / regulation of immunoglobulin production / 傍分泌 / poly-ADP-D-ribose modification-dependent protein binding / cGAS/STING signaling pathway / regulation of immunoglobulin production /  regulation of T cell activation / pattern recognition receptor signaling pathway / negative regulation of double-strand break repair via homologous recombination ... regulation of T cell activation / pattern recognition receptor signaling pathway / negative regulation of double-strand break repair via homologous recombination ... cyclic GMP-AMP synthase / 2',3'-cyclic GMP-AMP synthase activity / STING mediated induction of host immune responses / cyclic GMP-AMP synthase / 2',3'-cyclic GMP-AMP synthase activity / STING mediated induction of host immune responses /  傍分泌 / poly-ADP-D-ribose modification-dependent protein binding / cGAS/STING signaling pathway / regulation of immunoglobulin production / 傍分泌 / poly-ADP-D-ribose modification-dependent protein binding / cGAS/STING signaling pathway / regulation of immunoglobulin production /  regulation of T cell activation / pattern recognition receptor signaling pathway / negative regulation of double-strand break repair via homologous recombination / negative regulation of cGAS/STING signaling pathway / cellular response to exogenous dsRNA / cytoplasmic pattern recognition receptor signaling pathway / cGMP-mediated signaling / cAMP-mediated signaling / regulation of T cell activation / pattern recognition receptor signaling pathway / negative regulation of double-strand break repair via homologous recombination / negative regulation of cGAS/STING signaling pathway / cellular response to exogenous dsRNA / cytoplasmic pattern recognition receptor signaling pathway / cGMP-mediated signaling / cAMP-mediated signaling /  nucleosome binding / positive regulation of type I interferon production / negative regulation of megakaryocyte differentiation / protein localization to CENP-A containing chromatin / Chromatin modifying enzymes / Replacement of protamines by nucleosomes in the male pronucleus / CENP-A containing nucleosome / Packaging Of Telomere Ends / Recognition and association of DNA glycosylase with site containing an affected purine / Cleavage of the damaged purine / Deposition of new CENPA-containing nucleosomes at the centromere / positive regulation of defense response to virus by host / Recognition and association of DNA glycosylase with site containing an affected pyrimidine / Cleavage of the damaged pyrimidine / nucleosome binding / positive regulation of type I interferon production / negative regulation of megakaryocyte differentiation / protein localization to CENP-A containing chromatin / Chromatin modifying enzymes / Replacement of protamines by nucleosomes in the male pronucleus / CENP-A containing nucleosome / Packaging Of Telomere Ends / Recognition and association of DNA glycosylase with site containing an affected purine / Cleavage of the damaged purine / Deposition of new CENPA-containing nucleosomes at the centromere / positive regulation of defense response to virus by host / Recognition and association of DNA glycosylase with site containing an affected pyrimidine / Cleavage of the damaged pyrimidine /  phosphatidylinositol-4,5-bisphosphate binding / Inhibition of DNA recombination at telomere / Meiotic synapsis / telomere organization / activation of innate immune response / molecular condensate scaffold activity / RNA Polymerase I Promoter Opening / Interleukin-7 signaling / SUMOylation of chromatin organization proteins / Assembly of the ORC complex at the origin of replication / phosphatidylinositol-4,5-bisphosphate binding / Inhibition of DNA recombination at telomere / Meiotic synapsis / telomere organization / activation of innate immune response / molecular condensate scaffold activity / RNA Polymerase I Promoter Opening / Interleukin-7 signaling / SUMOylation of chromatin organization proteins / Assembly of the ORC complex at the origin of replication /  DNAメチル化 / Condensation of Prophase Chromosomes / HCMV Late Events / Chromatin modifications during the maternal to zygotic transition (MZT) / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / SIRT1 negatively regulates rRNA expression / DNAメチル化 / Condensation of Prophase Chromosomes / HCMV Late Events / Chromatin modifications during the maternal to zygotic transition (MZT) / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / SIRT1 negatively regulates rRNA expression /  innate immune response in mucosa / PRC2 methylates histones and DNA / Defective pyroptosis / HDACs deacetylate histones / determination of adult lifespan / RNA Polymerase I Promoter Escape / Nonhomologous End-Joining (NHEJ) / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / NoRC negatively regulates rRNA expression / G2/M DNA damage checkpoint / B-WICH complex positively regulates rRNA expression / HDMs demethylate histones / DNA Damage/Telomere Stress Induced Senescence / Metalloprotease DUBs / PKMTs methylate histone lysines / RMTs methylate histone arginines / innate immune response in mucosa / PRC2 methylates histones and DNA / Defective pyroptosis / HDACs deacetylate histones / determination of adult lifespan / RNA Polymerase I Promoter Escape / Nonhomologous End-Joining (NHEJ) / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / NoRC negatively regulates rRNA expression / G2/M DNA damage checkpoint / B-WICH complex positively regulates rRNA expression / HDMs demethylate histones / DNA Damage/Telomere Stress Induced Senescence / Metalloprotease DUBs / PKMTs methylate histone lysines / RMTs methylate histone arginines /  遺伝的組換え / Pre-NOTCH Transcription and Translation / 遺伝的組換え / Pre-NOTCH Transcription and Translation /  nucleosome assembly / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / Transcriptional regulation of granulopoiesis / structural constituent of chromatin / UCH proteinases / positive regulation of cellular senescence / nucleosome assembly / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / Transcriptional regulation of granulopoiesis / structural constituent of chromatin / UCH proteinases / positive regulation of cellular senescence /  ヌクレオソーム / antimicrobial humoral immune response mediated by antimicrobial peptide / E3 ubiquitin ligases ubiquitinate target proteins / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks / site of double-strand break / RUNX1 regulates transcription of genes involved in differentiation of HSCs / chromatin organization / Factors involved in megakaryocyte development and platelet production / Processing of DNA double-strand break ends / HATs acetylate histones / antibacterial humoral response / Senescence-Associated Secretory Phenotype (SASP) / ヌクレオソーム / antimicrobial humoral immune response mediated by antimicrobial peptide / E3 ubiquitin ligases ubiquitinate target proteins / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks / site of double-strand break / RUNX1 regulates transcription of genes involved in differentiation of HSCs / chromatin organization / Factors involved in megakaryocyte development and platelet production / Processing of DNA double-strand break ends / HATs acetylate histones / antibacterial humoral response / Senescence-Associated Secretory Phenotype (SASP) /  double-stranded DNA binding / defense response to virus / Oxidative Stress Induced Senescence / killing of cells of another organism / Estrogen-dependent gene expression / defense response to Gram-negative bacterium / double-stranded DNA binding / defense response to virus / Oxidative Stress Induced Senescence / killing of cells of another organism / Estrogen-dependent gene expression / defense response to Gram-negative bacterium /  chromosome, telomeric region / chromosome, telomeric region /  nuclear body / Ub-specific processing proteases / defense response to Gram-positive bacterium / Amyloid fiber formation / protein heterodimerization activity / nuclear body / Ub-specific processing proteases / defense response to Gram-positive bacterium / Amyloid fiber formation / protein heterodimerization activity /  DNA修復 / DNA修復 /  自然免疫系 / DNA damage response 自然免疫系 / DNA damage response類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.15 Å クライオ電子顕微鏡法 / 解像度: 3.15 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Pathare, G.R. / Cavadini, S. / Kempf, G. / Thoma, N.H. | ||||||||||||||||||
| 資金援助 |  スイス, 5件 スイス, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2020 ジャーナル: Nature / 年: 2020タイトル: Structural mechanism of cGAS inhibition by the nucleosome. 著者: Ganesh R Pathare / Alexiane Decout / Selene Glück / Simone Cavadini / Kristina Makasheva / Ruud Hovius / Georg Kempf / Joscha Weiss / Zuzanna Kozicka / Baptiste Guey / Pauline Melenec / Beat ...著者: Ganesh R Pathare / Alexiane Decout / Selene Glück / Simone Cavadini / Kristina Makasheva / Ruud Hovius / Georg Kempf / Joscha Weiss / Zuzanna Kozicka / Baptiste Guey / Pauline Melenec / Beat Fierz / Nicolas H Thomä / Andrea Ablasser /  要旨: The DNA sensor cyclic GMP-AMP synthase (cGAS) initiates innate immune responses following microbial infection, cellular stress and cancer. Upon activation by double-stranded DNA, cytosolic cGAS ...The DNA sensor cyclic GMP-AMP synthase (cGAS) initiates innate immune responses following microbial infection, cellular stress and cancer. Upon activation by double-stranded DNA, cytosolic cGAS produces 2'3' cGMP-AMP, which triggers the induction of inflammatory cytokines and type I interferons . cGAS is also present inside the cell nucleus, which is replete with genomic DNA, where chromatin has been implicated in restricting its enzymatic activity. However, the structural basis for inhibition of cGAS by chromatin remains unknown. Here we present the cryo-electron microscopy structure of human cGAS bound to nucleosomes. cGAS makes extensive contacts with both the acidic patch of the histone H2A-H2B heterodimer and nucleosomal DNA. The structural and complementary biochemical analysis also find cGAS engaged to a second nucleosome in trans. Mechanistically, binding of the nucleosome locks cGAS into a monomeric state, in which steric hindrance suppresses spurious activation by genomic DNA. We find that mutations to the cGAS-acidic patch interface are sufficient to abolish the inhibitory effect of nucleosomes in vitro and to unleash the activity of cGAS on genomic DNA in living cells. Our work uncovers the structural basis of the interaction between cGAS and chromatin and details a mechanism that permits self-non-self discrimination of genomic DNA by cGAS. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6y5e.cif.gz 6y5e.cif.gz | 390.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6y5e.ent.gz pdb6y5e.ent.gz | 307.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6y5e.json.gz 6y5e.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/y5/6y5e https://data.pdbj.org/pub/pdb/validation_reports/y5/6y5e ftp://data.pdbj.org/pub/pdb/validation_reports/y5/6y5e ftp://data.pdbj.org/pub/pdb/validation_reports/y5/6y5e | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 4種, 5分子 AEBFK
| #1: タンパク質 | 分子量: 11228.073 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: HIST2H3A, HIST2H3C, H3F2, H3FM, HIST2H3D / 発現宿主: Homo sapiens (ヒト) / 遺伝子: HIST2H3A, HIST2H3C, H3F2, H3FM, HIST2H3D / 発現宿主:   Escherichia coli K-12 (大腸菌) / 参照: UniProt: Q71DI3 Escherichia coli K-12 (大腸菌) / 参照: UniProt: Q71DI3#2: タンパク質 | |  ヒストンH4 ヒストンH4分子量: 9180.745 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) Homo sapiens (ヒト)遺伝子: H4C1, H4/A, H4FA, HIST1H4A, H4C2, H4/I, H4FI, HIST1H4B, H4C3, H4/G, H4FG, HIST1H4C, H4C4, H4/B, H4FB, HIST1H4D, H4C5, H4/J, H4FJ, HIST1H4E, H4C6, H4/C, H4FC, HIST1H4F, H4C8, H4/H, H4FH, ...遺伝子: H4C1, H4/A, H4FA, HIST1H4A, H4C2, H4/I, H4FI, HIST1H4B, H4C3, H4/G, H4FG, HIST1H4C, H4C4, H4/B, H4FB, HIST1H4D, H4C5, H4/J, H4FJ, HIST1H4E, H4C6, H4/C, H4FC, HIST1H4F, H4C8, H4/H, H4FH, HIST1H4H, H4C9, H4/M, H4FM, HIST1H4I, H4C11, H4/E, H4FE, HIST1H4J, H4C12, H4/D, H4FD, HIST1H4K, H4C13, H4/K, H4FK, HIST1H4L, H4C14, H4/N, H4F2, H4FN, HIST2H4, HIST2H4A, H4C15, H4/O, H4FO, HIST2H4B, H4-16, HIST4H4 発現宿主:   Escherichia coli K-12 (大腸菌) / 参照: UniProt: P62805 Escherichia coli K-12 (大腸菌) / 参照: UniProt: P62805#5: タンパク質 | |  ヒストンH4 ヒストンH4分子量: 9409.056 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) Homo sapiens (ヒト)遺伝子: H4C1, H4/A, H4FA, HIST1H4A, H4C2, H4/I, H4FI, HIST1H4B, H4C3, H4/G, H4FG, HIST1H4C, H4C4, H4/B, H4FB, HIST1H4D, H4C5, H4/J, H4FJ, HIST1H4E, H4C6, H4/C, H4FC, HIST1H4F, H4C8, H4/H, H4FH, ...遺伝子: H4C1, H4/A, H4FA, HIST1H4A, H4C2, H4/I, H4FI, HIST1H4B, H4C3, H4/G, H4FG, HIST1H4C, H4C4, H4/B, H4FB, HIST1H4D, H4C5, H4/J, H4FJ, HIST1H4E, H4C6, H4/C, H4FC, HIST1H4F, H4C8, H4/H, H4FH, HIST1H4H, H4C9, H4/M, H4FM, HIST1H4I, H4C11, H4/E, H4FE, HIST1H4J, H4C12, H4/D, H4FD, HIST1H4K, H4C13, H4/K, H4FK, HIST1H4L, H4C14, H4/N, H4F2, H4FN, HIST2H4, HIST2H4A, H4C15, H4/O, H4FO, HIST2H4B, H4-16, HIST4H4 発現宿主:   Escherichia coli K-12 (大腸菌) / 参照: UniProt: P62805 Escherichia coli K-12 (大腸菌) / 参照: UniProt: P62805#10: タンパク質 | |  / h-cGAS / 2'3'-cGAMP synthase / Mab-21 domain-containing protein 1 / h-cGAS / 2'3'-cGAMP synthase / Mab-21 domain-containing protein 1分子量: 42404.840 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: CGAS, C6orf150, MB21D1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: CGAS, C6orf150, MB21D1 / 発現宿主:   Escherichia coli K-12 (大腸菌) / 参照: UniProt: Q8N884, Escherichia coli K-12 (大腸菌) / 参照: UniProt: Q8N884,  cyclic GMP-AMP synthase cyclic GMP-AMP synthase |
|---|
-Histone H2A type 2- ... , 2種, 2分子 CG
| #3: タンパク質 | 分子量: 11696.688 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: HIST2H2AC, H2AFQ / 発現宿主: Homo sapiens (ヒト) / 遺伝子: HIST2H2AC, H2AFQ / 発現宿主:   Escherichia coli K-12 (大腸菌) / 参照: UniProt: Q16777 Escherichia coli K-12 (大腸菌) / 参照: UniProt: Q16777 |
|---|---|
| #6: タンパク質 | 分子量: 11825.869 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: HIST2H2AC, H2AFQ / 発現宿主: Homo sapiens (ヒト) / 遺伝子: HIST2H2AC, H2AFQ / 発現宿主:   Escherichia coli K-12 (大腸菌) / 参照: UniProt: Q16777 Escherichia coli K-12 (大腸菌) / 参照: UniProt: Q16777 |
-Histone H2B type 1- ... , 2種, 2分子 DH
| #4: タンパク質 | 分子量: 10320.800 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: H2BC12, H2BFT, HIRIP1, HIST1H2BK / 発現宿主: Homo sapiens (ヒト) / 遺伝子: H2BC12, H2BFT, HIRIP1, HIST1H2BK / 発現宿主:   Escherichia coli K-12 (大腸菌) / 参照: UniProt: O60814 Escherichia coli K-12 (大腸菌) / 参照: UniProt: O60814 |
|---|---|
| #7: タンパク質 | 分子量: 10477.994 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: H2BC12, H2BFT, HIRIP1, HIST1H2BK / 発現宿主: Homo sapiens (ヒト) / 遺伝子: H2BC12, H2BFT, HIRIP1, HIST1H2BK / 発現宿主:   Escherichia coli K-12 (大腸菌) / 参照: UniProt: O60814 Escherichia coli K-12 (大腸菌) / 参照: UniProt: O60814 |
-DNA鎖 , 2種, 2分子 IJ
| #8: DNA鎖 | 分子量: 46998.945 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)   Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|---|
| #9: DNA鎖 | 分子量: 47457.234 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-非ポリマー , 2種, 9分子 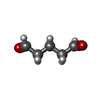


| #11: 化合物 | ChemComp-PTD /  グルタルアルデヒド グルタルアルデヒド#12: 化合物 | ChemComp-ZN / | |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.24 MDa / 実験値: YES | ||||||||||||||||||||||||||||||
| 由来(天然) |
| ||||||||||||||||||||||||||||||
| 由来(組換発現) |
| ||||||||||||||||||||||||||||||
| 緩衝液 | pH: 7.4 | ||||||||||||||||||||||||||||||
| 試料 | 濃度: 1 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES | ||||||||||||||||||||||||||||||
| 試料支持 | グリッドの材料: COPPER / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 | ||||||||||||||||||||||||||||||
急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: SPOT SCAN FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: SPOT SCAN |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / Cs Bright-field microscopy / Cs : 0.01 mm / C2レンズ絞り径: 100 µm : 0.01 mm / C2レンズ絞り径: 100 µm |
| 撮影 | 電子線照射量: 45 e/Å2 / 検出モード: COUNTING フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) |
- 解析
解析
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION |
|---|---|
| 対称性 | 点対称性 : C1 (非対称) : C1 (非対称) |
3次元再構成 | 解像度: 3.15 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 62036 / 対称性のタイプ: POINT |
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj












































