+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6aky | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | The Crystal structure of Human Chemokine Receptor CCR5 in complex with compound 34 | ||||||
 要素 要素 | C-C chemokine receptor type 5,Rubredoxin,C-C chemokine receptor type 5 | ||||||
 キーワード キーワード |  SIGNALING PROTEIN / G Protein-Coupled Receptor Chemokine Receptor CCR5 Antagonist Complex structure SIGNALING PROTEIN / G Protein-Coupled Receptor Chemokine Receptor CCR5 Antagonist Complex structure | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報chemokine (C-C motif) ligand 5 binding / negative regulation of macrophage apoptotic process /  signaling / signaling /  chemokine receptor activity / C-C chemokine receptor activity / C-C chemokine binding / phosphatidylinositol phospholipase C activity / response to cholesterol / Chemokine receptors bind chemokines / release of sequestered calcium ion into cytosol by sarcoplasmic reticulum ...chemokine (C-C motif) ligand 5 binding / negative regulation of macrophage apoptotic process / chemokine receptor activity / C-C chemokine receptor activity / C-C chemokine binding / phosphatidylinositol phospholipase C activity / response to cholesterol / Chemokine receptors bind chemokines / release of sequestered calcium ion into cytosol by sarcoplasmic reticulum ...chemokine (C-C motif) ligand 5 binding / negative regulation of macrophage apoptotic process /  signaling / signaling /  chemokine receptor activity / C-C chemokine receptor activity / C-C chemokine binding / phosphatidylinositol phospholipase C activity / response to cholesterol / Chemokine receptors bind chemokines / release of sequestered calcium ion into cytosol by sarcoplasmic reticulum / dendritic cell chemotaxis / Interleukin-10 signaling / Binding and entry of HIV virion / cellular defense response / chemokine receptor activity / C-C chemokine receptor activity / C-C chemokine binding / phosphatidylinositol phospholipase C activity / response to cholesterol / Chemokine receptors bind chemokines / release of sequestered calcium ion into cytosol by sarcoplasmic reticulum / dendritic cell chemotaxis / Interleukin-10 signaling / Binding and entry of HIV virion / cellular defense response /  coreceptor activity / cell chemotaxis / calcium-mediated signaling / calcium ion transport / coreceptor activity / cell chemotaxis / calcium-mediated signaling / calcium ion transport /  走化性 / 走化性 /  分裂促進因子活性化タンパク質キナーゼ / virus receptor activity / cell-cell signaling / 分裂促進因子活性化タンパク質キナーゼ / virus receptor activity / cell-cell signaling /  actin binding / positive regulation of cytosolic calcium ion concentration / G alpha (i) signalling events / cellular response to lipopolysaccharide / actin binding / positive regulation of cytosolic calcium ion concentration / G alpha (i) signalling events / cellular response to lipopolysaccharide /  electron transfer activity / cell surface receptor signaling pathway / electron transfer activity / cell surface receptor signaling pathway /  エンドソーム / エンドソーム /  免疫応答 / 免疫応答 /  炎症 / iron ion binding / G protein-coupled receptor signaling pathway / external side of plasma membrane / 炎症 / iron ion binding / G protein-coupled receptor signaling pathway / external side of plasma membrane /  細胞膜 / identical protein binding / 細胞膜 / identical protein binding /  細胞膜 / 細胞膜 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト)  Clostridium pasteurianum (バクテリア) Clostridium pasteurianum (バクテリア) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.8 Å 分子置換 / 解像度: 2.8 Å | ||||||
 データ登録者 データ登録者 | Zhu, Y. / Zhao, Q. / Wu, B. | ||||||
 引用 引用 |  ジャーナル: J. Med. Chem. / 年: 2018 ジャーナル: J. Med. Chem. / 年: 2018タイトル: Structure-Based Design of 1-Heteroaryl-1,3-propanediamine Derivatives as a Novel Series of CC-Chemokine Receptor 5 Antagonists. 著者: Peng, P. / Chen, H. / Zhu, Y. / Wang, Z. / Li, J. / Luo, R.H. / Wang, J. / Chen, L. / Yang, L.M. / Jiang, H. / Xie, X. / Wu, B. / Zheng, Y.T. / Liu, H. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6aky.cif.gz 6aky.cif.gz | 86.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6aky.ent.gz pdb6aky.ent.gz | 60.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6aky.json.gz 6aky.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ak/6aky https://data.pdbj.org/pub/pdb/validation_reports/ak/6aky ftp://data.pdbj.org/pub/pdb/validation_reports/ak/6aky ftp://data.pdbj.org/pub/pdb/validation_reports/ak/6aky | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 43876.227 Da / 分子数: 1 / 変異: C58Y, G163N, A233D, K303E / 由来タイプ: 組換発現 詳細: Chimera protein of C-C chemokine receptor type 5 and Rubredoxin. Rubredoxin was inserted into CCR5 between residue 223 and 227. 由来: (組換発現)   Homo sapiens (ヒト), (組換発現) Homo sapiens (ヒト), (組換発現)   Clostridium pasteurianum (バクテリア) Clostridium pasteurianum (バクテリア)遺伝子: CCR5, CMKBR5 発現宿主:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)参照: UniProt: P51681, UniProt: P00268 |
|---|---|
| #2: 化合物 | ChemComp-ZN / |
| #3: 化合物 | ChemComp-OLC / ( |
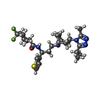
| #4: 化合物 | ChemComp-A4X / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.59 Å3/Da / 溶媒含有率: 52.6 % |
|---|---|
結晶化 | 温度: 293 K / 手法: 脂質キュービック相法 / 詳細: PEG400, HEPES, ammonium acetate |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SPring-8 SPring-8  / ビームライン: BL41XU / 波長: 1 Å / ビームライン: BL41XU / 波長: 1 Å |
| 検出器 | タイプ: DECTRIS PILATUS3 6M / 検出器: PIXEL / 日付: 2015年9月13日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1 Å / 相対比: 1 : 1 Å / 相対比: 1 |
| 反射 | 解像度: 2.8→50 Å / Num. obs: 12438 / % possible obs: 96.7 % / 冗長度: 4 % / Biso Wilson estimate: 82.94 Å2 / Rpim(I) all: 0.069 / Net I/σ(I): 19 |
| 反射 シェル | 解像度: 2.8→2.9 Å / Num. unique obs: 2071 / Rpim(I) all: 0.576 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 4MBS 解像度: 2.8→31.56 Å / Cor.coef. Fo:Fc: 0.905 / Cor.coef. Fo:Fc free: 0.912 / SU R Cruickshank DPI: 1.016 / 交差検証法: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 1.059 / SU Rfree Blow DPI: 0.37 / SU Rfree Cruickshank DPI: 0.373
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 82.89 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.54 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 解像度: 2.8→31.56 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.8→3.07 Å / Total num. of bins used: 6
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj