| 登録情報 | データベース: PDB / ID: 4nxq
|
|---|
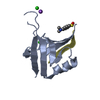
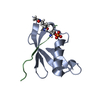
| タイトル | Crystal Structure of T-cell Lymphoma Invasion and Metastasis-1 PDZ Domain Quadruple Mutant (QM) in Complex With Caspr4 Peptide |
|---|
 要素 要素 | - Contactin-associated protein-like 4 peptide
- T-lymphoma invasion and metastasis-inducing protein 1
|
|---|
 キーワード キーワード |  signaling Protein/Peptide / Beta barrel fold protein / signaling Protein/Peptide / Beta barrel fold protein /  PDZ domain (PDZドメイン) / PDZ domain (PDZドメイン) /  peptide binding (ペプチド) / specificity mutant / scaffold signaling protein for cell adhesion and cell junction / peptide binding (ペプチド) / specificity mutant / scaffold signaling protein for cell adhesion and cell junction /  signaling domain / protein-peptide complex / signaling domain / protein-peptide complex /  signaling Protein-Peptide complex signaling Protein-Peptide complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
positive regulation of Schwann cell chemotaxis / regulation of non-canonical Wnt signaling pathway / regulation of dopaminergic neuron differentiation /  regulation of synaptic transmission, dopaminergic / regulation of synaptic transmission, dopaminergic /  extrinsic component of postsynaptic density membrane / Activated NTRK2 signals through CDK5 / regulation of grooming behavior / extrinsic component of postsynaptic density membrane / Activated NTRK2 signals through CDK5 / regulation of grooming behavior /  regulation of epithelial to mesenchymal transition / positive regulation of dendritic spine morphogenesis / regulation of insulin secretion involved in cellular response to glucose stimulus ...positive regulation of Schwann cell chemotaxis / regulation of non-canonical Wnt signaling pathway / regulation of dopaminergic neuron differentiation / regulation of epithelial to mesenchymal transition / positive regulation of dendritic spine morphogenesis / regulation of insulin secretion involved in cellular response to glucose stimulus ...positive regulation of Schwann cell chemotaxis / regulation of non-canonical Wnt signaling pathway / regulation of dopaminergic neuron differentiation /  regulation of synaptic transmission, dopaminergic / regulation of synaptic transmission, dopaminergic /  extrinsic component of postsynaptic density membrane / Activated NTRK2 signals through CDK5 / regulation of grooming behavior / extrinsic component of postsynaptic density membrane / Activated NTRK2 signals through CDK5 / regulation of grooming behavior /  regulation of epithelial to mesenchymal transition / positive regulation of dendritic spine morphogenesis / regulation of insulin secretion involved in cellular response to glucose stimulus / regulation of modification of postsynaptic actin cytoskeleton / cell-cell contact zone / protein localization to membrane / cardiac muscle hypertrophy / activation of GTPase activity / regulation of epithelial to mesenchymal transition / positive regulation of dendritic spine morphogenesis / regulation of insulin secretion involved in cellular response to glucose stimulus / regulation of modification of postsynaptic actin cytoskeleton / cell-cell contact zone / protein localization to membrane / cardiac muscle hypertrophy / activation of GTPase activity /  Wnt signaling pathway, planar cell polarity pathway / regulation of small GTPase mediated signal transduction / main axon / Wnt signaling pathway, planar cell polarity pathway / regulation of small GTPase mediated signal transduction / main axon /  regulation of synaptic transmission, GABAergic / neuron projection extension / positive regulation of axonogenesis / small GTPase-mediated signal transduction / NRAGE signals death through JNK / Rac protein signal transduction / CDC42 GTPase cycle / EPH-ephrin mediated repulsion of cells / RHOA GTPase cycle / ephrin receptor signaling pathway / RAC2 GTPase cycle / RAC3 GTPase cycle / positive regulation of epithelial to mesenchymal transition / axonal growth cone / EPHB-mediated forward signaling / RAC1 GTPase cycle / cell-matrix adhesion / regulation of ERK1 and ERK2 cascade / guanyl-nucleotide exchange factor activity / response to cocaine / cell projection / regulation of synaptic transmission, GABAergic / neuron projection extension / positive regulation of axonogenesis / small GTPase-mediated signal transduction / NRAGE signals death through JNK / Rac protein signal transduction / CDC42 GTPase cycle / EPH-ephrin mediated repulsion of cells / RHOA GTPase cycle / ephrin receptor signaling pathway / RAC2 GTPase cycle / RAC3 GTPase cycle / positive regulation of epithelial to mesenchymal transition / axonal growth cone / EPHB-mediated forward signaling / RAC1 GTPase cycle / cell-matrix adhesion / regulation of ERK1 and ERK2 cascade / guanyl-nucleotide exchange factor activity / response to cocaine / cell projection /  receptor tyrosine kinase binding / ruffle membrane / receptor tyrosine kinase binding / ruffle membrane /  kinase binding / G alpha (12/13) signalling events / kinase binding / G alpha (12/13) signalling events /  遊走 / cell-cell junction / 遊走 / cell-cell junction /  presynaptic membrane / positive regulation of protein binding / presynaptic membrane / positive regulation of protein binding /  microtubule binding / protein-containing complex assembly / microtubule binding / protein-containing complex assembly /  微小管 / 微小管 /  樹状突起スパイン / membrane => GO:0016020 / 樹状突起スパイン / membrane => GO:0016020 /  細胞接着 / positive regulation of cell migration / neuronal cell body / 細胞接着 / positive regulation of cell migration / neuronal cell body /  シナプス / glutamatergic synapse / シナプス / glutamatergic synapse /  lipid binding / positive regulation of cell population proliferation / lipid binding / positive regulation of cell population proliferation /  細胞膜 / 細胞膜 /  細胞質基質類似検索 - 分子機能 細胞質基質類似検索 - 分子機能 Contactin-associated protein-like 4 /  TIAM1, CC-Ex domain / Tiam1/Tiam2/Protein still life / T-lymphoma invasion and metastasis CC-Ex domain / TIAM1, CC-Ex domain / Tiam1/Tiam2/Protein still life / T-lymphoma invasion and metastasis CC-Ex domain /  ラミニン / Guanine-nucleotide dissociation stimulator, CDC24, conserved site / Dbl homology (DH) domain signature. / Laminin G domain profile. / ラミニン / Guanine-nucleotide dissociation stimulator, CDC24, conserved site / Dbl homology (DH) domain signature. / Laminin G domain profile. /  ラミニン / ラミニン /  ラミニン ...Contactin-associated protein-like 4 / ラミニン ...Contactin-associated protein-like 4 /  TIAM1, CC-Ex domain / Tiam1/Tiam2/Protein still life / T-lymphoma invasion and metastasis CC-Ex domain / TIAM1, CC-Ex domain / Tiam1/Tiam2/Protein still life / T-lymphoma invasion and metastasis CC-Ex domain /  ラミニン / Guanine-nucleotide dissociation stimulator, CDC24, conserved site / Dbl homology (DH) domain signature. / Laminin G domain profile. / ラミニン / Guanine-nucleotide dissociation stimulator, CDC24, conserved site / Dbl homology (DH) domain signature. / Laminin G domain profile. /  ラミニン / ラミニン /  ラミニン / Coagulation factors 5/8 type C domain (FA58C) signature 2. / ラミニン / Coagulation factors 5/8 type C domain (FA58C) signature 2. /  Fibrinogen, alpha/beta/gamma chain, C-terminal globular domain / Fibrinogen-like, C-terminal / Fibrinogen C-terminal domain profile. / Coagulation factors 5/8 type C domain (FA58C) signature 1. / Fibrinogen, alpha/beta/gamma chain, C-terminal globular domain / Fibrinogen-like, C-terminal / Fibrinogen C-terminal domain profile. / Coagulation factors 5/8 type C domain (FA58C) signature 1. /  Raf-like Ras-binding domain / Raf-like Ras-binding / Ras-binding domain (RBD) profile. / Raf-like Ras-binding domain / Raf-like Ras-binding / Ras-binding domain (RBD) profile. /  Raf-like Ras-binding domain / Coagulation factor 5/8 C-terminal domain, discoidin domain / Coagulation factors 5/8 type C domain (FA58C) profile. / Raf-like Ras-binding domain / Coagulation factor 5/8 C-terminal domain, discoidin domain / Coagulation factors 5/8 type C domain (FA58C) profile. /  F5/8 type C domain / Coagulation factor 5/8 C-terminal domain / Dbl homology (DH) domain superfamily / F5/8 type C domain / Coagulation factor 5/8 C-terminal domain / Dbl homology (DH) domain superfamily /  RhoGEF domain / Guanine nucleotide exchange factor for Rho/Rac/Cdc42-like GTPases / Dbl homology (DH) domain / Dbl homology (DH) domain profile. / RhoGEF domain / Guanine nucleotide exchange factor for Rho/Rac/Cdc42-like GTPases / Dbl homology (DH) domain / Dbl homology (DH) domain profile. /  PDZドメイン / Pdz3 Domain / PDZドメイン / Pdz3 Domain /  Epidermal growth factor-like domain. / Epidermal growth factor-like domain. /  PH domain / EGF-like domain profile. / PH domain profile. / PH domain / EGF-like domain profile. / PH domain profile. /  Pleckstrin homology domain. / Pleckstrin homology domain. /  PDZドメイン / PDZドメイン /  Pleckstrin homology domain / Pleckstrin homology domain /  EGF様ドメイン / PDZ domain profile. / Domain present in PSD-95, Dlg, and ZO-1/2. / EGF様ドメイン / PDZ domain profile. / Domain present in PSD-95, Dlg, and ZO-1/2. /  PDZドメイン / PDZ superfamily / Galactose-binding-like domain superfamily / Concanavalin A-like lectin/glucanase domain superfamily / PH-like domain superfamily / Roll / Mainly Beta類似検索 - ドメイン・相同性 PDZドメイン / PDZ superfamily / Galactose-binding-like domain superfamily / Concanavalin A-like lectin/glucanase domain superfamily / PH-like domain superfamily / Roll / Mainly Beta類似検索 - ドメイン・相同性 Rho guanine nucleotide exchange factor TIAM1 / Contactin-associated protein-like 4類似検索 - 構成要素 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.1 Å 分子置換 / 解像度: 2.1 Å |
|---|
 データ登録者 データ登録者 | Liu, X. / Speckhard, D.C. / Shepherd, T.R. / Hengel, S.R. / Fuentes, E.J. |
|---|
 引用 引用 |  ジャーナル: Structure / 年: 2016 ジャーナル: Structure / 年: 2016
タイトル: Distinct Roles for Conformational Dynamics in Protein-Ligand Interactions.
著者: Liu, X. / Speckhard, D.C. / Shepherd, T.R. / Sun, Y.J. / Hengel, S.R. / Yu, L. / Fowler, C.A. / Gakhar, L. / Fuentes, E.J. |
|---|
| 履歴 | | 登録 | 2013年12月9日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2015年5月13日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2017年2月15日 | Group: Database references |
|---|
| 改定 1.2 | 2022年12月21日 | Group: Database references / カテゴリ: database_2 / struct_ref_seq_dif
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details |
|---|
| 改定 1.3 | 2023年9月20日 | Group: Data collection / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond / pdbx_initial_refinement_model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード signaling Protein/Peptide / Beta barrel fold protein /
signaling Protein/Peptide / Beta barrel fold protein /  PDZ domain (PDZドメイン) /
PDZ domain (PDZドメイン) /  peptide binding (ペプチド) / specificity mutant / scaffold signaling protein for cell adhesion and cell junction /
peptide binding (ペプチド) / specificity mutant / scaffold signaling protein for cell adhesion and cell junction /  signaling domain / protein-peptide complex /
signaling domain / protein-peptide complex /  signaling Protein-Peptide complex
signaling Protein-Peptide complex 機能・相同性情報
機能・相同性情報 regulation of synaptic transmission, dopaminergic /
regulation of synaptic transmission, dopaminergic /  extrinsic component of postsynaptic density membrane / Activated NTRK2 signals through CDK5 / regulation of grooming behavior /
extrinsic component of postsynaptic density membrane / Activated NTRK2 signals through CDK5 / regulation of grooming behavior /  regulation of epithelial to mesenchymal transition / positive regulation of dendritic spine morphogenesis / regulation of insulin secretion involved in cellular response to glucose stimulus ...positive regulation of Schwann cell chemotaxis / regulation of non-canonical Wnt signaling pathway / regulation of dopaminergic neuron differentiation /
regulation of epithelial to mesenchymal transition / positive regulation of dendritic spine morphogenesis / regulation of insulin secretion involved in cellular response to glucose stimulus ...positive regulation of Schwann cell chemotaxis / regulation of non-canonical Wnt signaling pathway / regulation of dopaminergic neuron differentiation /  regulation of synaptic transmission, dopaminergic /
regulation of synaptic transmission, dopaminergic /  extrinsic component of postsynaptic density membrane / Activated NTRK2 signals through CDK5 / regulation of grooming behavior /
extrinsic component of postsynaptic density membrane / Activated NTRK2 signals through CDK5 / regulation of grooming behavior /  regulation of epithelial to mesenchymal transition / positive regulation of dendritic spine morphogenesis / regulation of insulin secretion involved in cellular response to glucose stimulus / regulation of modification of postsynaptic actin cytoskeleton / cell-cell contact zone / protein localization to membrane / cardiac muscle hypertrophy / activation of GTPase activity /
regulation of epithelial to mesenchymal transition / positive regulation of dendritic spine morphogenesis / regulation of insulin secretion involved in cellular response to glucose stimulus / regulation of modification of postsynaptic actin cytoskeleton / cell-cell contact zone / protein localization to membrane / cardiac muscle hypertrophy / activation of GTPase activity /  Wnt signaling pathway, planar cell polarity pathway / regulation of small GTPase mediated signal transduction / main axon /
Wnt signaling pathway, planar cell polarity pathway / regulation of small GTPase mediated signal transduction / main axon /  regulation of synaptic transmission, GABAergic / neuron projection extension / positive regulation of axonogenesis / small GTPase-mediated signal transduction / NRAGE signals death through JNK / Rac protein signal transduction / CDC42 GTPase cycle / EPH-ephrin mediated repulsion of cells / RHOA GTPase cycle / ephrin receptor signaling pathway / RAC2 GTPase cycle / RAC3 GTPase cycle / positive regulation of epithelial to mesenchymal transition / axonal growth cone / EPHB-mediated forward signaling / RAC1 GTPase cycle / cell-matrix adhesion / regulation of ERK1 and ERK2 cascade / guanyl-nucleotide exchange factor activity / response to cocaine / cell projection /
regulation of synaptic transmission, GABAergic / neuron projection extension / positive regulation of axonogenesis / small GTPase-mediated signal transduction / NRAGE signals death through JNK / Rac protein signal transduction / CDC42 GTPase cycle / EPH-ephrin mediated repulsion of cells / RHOA GTPase cycle / ephrin receptor signaling pathway / RAC2 GTPase cycle / RAC3 GTPase cycle / positive regulation of epithelial to mesenchymal transition / axonal growth cone / EPHB-mediated forward signaling / RAC1 GTPase cycle / cell-matrix adhesion / regulation of ERK1 and ERK2 cascade / guanyl-nucleotide exchange factor activity / response to cocaine / cell projection /  receptor tyrosine kinase binding / ruffle membrane /
receptor tyrosine kinase binding / ruffle membrane /  kinase binding / G alpha (12/13) signalling events /
kinase binding / G alpha (12/13) signalling events /  遊走 / cell-cell junction /
遊走 / cell-cell junction /  presynaptic membrane / positive regulation of protein binding /
presynaptic membrane / positive regulation of protein binding /  microtubule binding / protein-containing complex assembly /
microtubule binding / protein-containing complex assembly /  微小管 /
微小管 /  樹状突起スパイン / membrane => GO:0016020 /
樹状突起スパイン / membrane => GO:0016020 /  細胞接着 / positive regulation of cell migration / neuronal cell body /
細胞接着 / positive regulation of cell migration / neuronal cell body /  シナプス / glutamatergic synapse /
シナプス / glutamatergic synapse /  lipid binding / positive regulation of cell population proliferation /
lipid binding / positive regulation of cell population proliferation /  細胞膜 /
細胞膜 /  細胞質基質
細胞質基質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.1 Å
分子置換 / 解像度: 2.1 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Structure / 年: 2016
ジャーナル: Structure / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4nxq.cif.gz
4nxq.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4nxq.ent.gz
pdb4nxq.ent.gz PDB形式
PDB形式 4nxq.json.gz
4nxq.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/nx/4nxq
https://data.pdbj.org/pub/pdb/validation_reports/nx/4nxq ftp://data.pdbj.org/pub/pdb/validation_reports/nx/4nxq
ftp://data.pdbj.org/pub/pdb/validation_reports/nx/4nxq リンク
リンク 集合体
集合体



 要素
要素
 Homo sapiens (ヒト) / 株: human
Homo sapiens (ヒト) / 株: human ヒト / 遺伝子: TIAM1 / プラスミド: PET21A-6HIS-rTEV / 発現宿主:
ヒト / 遺伝子: TIAM1 / プラスミド: PET21A-6HIS-rTEV / 発現宿主: 
 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: Q13009
Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: Q13009
 Homo sapiens (ヒト) / 参照: UniProt: Q9C0A0
Homo sapiens (ヒト) / 参照: UniProt: Q9C0A0 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 4.2.2 / 波長: 1 Å
/ ビームライン: 4.2.2 / 波長: 1 Å : 1 Å / 相対比: 1
: 1 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj

















