+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7891 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative Stain EM map of polyclonal serum in complex with BG505 SOSIP.664 from rabbit 3417 at post boost 2. | |||||||||
 マップデータ マップデータ | Negative stain EM map of polyclonal serum in complex with BG505 SOSIP.664 from rabbit 3417 at post boost 2 | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 20.0 Å | |||||||||
 データ登録者 データ登録者 | Ward AB / Turner HL / Nogal B | |||||||||
 引用 引用 |  ジャーナル: Immunity / 年: 2018 ジャーナル: Immunity / 年: 2018タイトル: Electron-Microscopy-Based Epitope Mapping Defines Specificities of Polyclonal Antibodies Elicited during HIV-1 BG505 Envelope Trimer Immunization. 著者: Matteo Bianchi / Hannah L Turner / Bartek Nogal / Christopher A Cottrell / David Oyen / Matthias Pauthner / Raiza Bastidas / Rebecca Nedellec / Laura E McCoy / Ian A Wilson / Dennis R Burton ...著者: Matteo Bianchi / Hannah L Turner / Bartek Nogal / Christopher A Cottrell / David Oyen / Matthias Pauthner / Raiza Bastidas / Rebecca Nedellec / Laura E McCoy / Ian A Wilson / Dennis R Burton / Andrew B Ward / Lars Hangartner /   要旨: Characterizing polyclonal antibody responses via currently available methods is inherently complex and difficult. Mapping epitopes in an immune response is typically incomplete, which creates a ...Characterizing polyclonal antibody responses via currently available methods is inherently complex and difficult. Mapping epitopes in an immune response is typically incomplete, which creates a barrier to fully understanding the humoral response to antigens and hinders rational vaccine design efforts. Here, we describe a method of characterizing polyclonal responses by using electron microscopy, and we applied this method to the immunization of rabbits with an HIV-1 envelope glycoprotein vaccine candidate, BG505 SOSIP.664. We detected known epitopes within the polyclonal sera and revealed how antibody responses evolved during the prime-boosting strategy to ultimately result in a neutralizing antibody response. We uncovered previously unidentified epitopes, including an epitope proximal to one recognized by human broadly neutralizing antibodies as well as potentially distracting non-neutralizing epitopes. Our method provides an efficient and semiquantitative map of epitopes that are targeted in a polyclonal antibody response and should be of widespread utility in vaccine and infection studies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7891.map.gz emd_7891.map.gz | 6.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7891-v30.xml emd-7891-v30.xml emd-7891.xml emd-7891.xml | 11.1 KB 11.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7891.png emd_7891.png | 78.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7891 http://ftp.pdbj.org/pub/emdb/structures/EMD-7891 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7891 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7891 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7552C  7553C  7554C  7555C  7556C  7557C  7570C  7887C 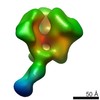 7888C  7889C  7890C  7892C  7893C  7894C  7895C  7896C  7903C  7904C  7906C  6cjkC  6didC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7891.map.gz / 形式: CCP4 / 大きさ: 11.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7891.map.gz / 形式: CCP4 / 大きさ: 11.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain EM map of polyclonal serum in complex with BG505 SOSIP.664 from rabbit 3417 at post boost 2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Negative stain EM map of polyclonal serum in complex with BG505 S...
| 全体 | 名称: Negative stain EM map of polyclonal serum in complex with BG505 SOSIP.664 from rabbit 3417 at post boost 2. Serum digested and Fab purified before adding trimer. |
|---|---|
| 要素 |
|
-超分子 #1: Negative stain EM map of polyclonal serum in complex with BG505 S...
| 超分子 | 名称: Negative stain EM map of polyclonal serum in complex with BG505 SOSIP.664 from rabbit 3417 at post boost 2. Serum digested and Fab purified before adding trimer. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.14 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 構成要素 - 式: TBS |
| 染色 | タイプ: NEGATIVE / 材質: Uranyl formate |
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CELLULOSE ACETATE / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI SPIRIT |
|---|---|
| 撮影 | フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 391 / 平均電子線量: 25.0 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 実験機器 |  モデル: Tecnai Spirit / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 20.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: RELION (ver. 2.1) / 使用した粒子像数: 40492 |
|---|---|
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: RELION (ver. 2.1) |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: RELION (ver. 2.1) |
| 最終 3次元分類 | ソフトウェア - 名称: RELION (ver. 2.1) |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















