+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3307 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the human TAF-less PIC in the closed state | |||||||||
 マップデータ マップデータ | Reconstruction of the human TAF-less PIC | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  TFIID (TFIID) / TFIID (TFIID) /  TFIIA / TFIIA /  transcription (転写 (生物学)) / transcription (転写 (生物学)) /  RNA polymerase II (RNAポリメラーゼII) / RNA polymerase II (RNAポリメラーゼII) /  general transcription factors (基本転写因子) / general transcription factors (基本転写因子) /  preinitiation complex / preinitiation complex /  core promoter (プロモーター) / core promoter (プロモーター) /  DNA binding (デオキシリボ核酸) DNA binding (デオキシリボ核酸) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 LRR domain binding / LRR domain binding /  microfibril binding / RNA Polymerase III Chain Elongation / MMXD complex / core TFIIH complex portion of holo TFIIH complex / RNA Polymerase III Transcription Termination / positive regulation of DNA helicase activity / negative regulation of DNA helicase activity / Cytosolic iron-sulfur cluster assembly / nucleotide-excision repair, DNA duplex unwinding ... microfibril binding / RNA Polymerase III Chain Elongation / MMXD complex / core TFIIH complex portion of holo TFIIH complex / RNA Polymerase III Transcription Termination / positive regulation of DNA helicase activity / negative regulation of DNA helicase activity / Cytosolic iron-sulfur cluster assembly / nucleotide-excision repair, DNA duplex unwinding ... LRR domain binding / LRR domain binding /  microfibril binding / RNA Polymerase III Chain Elongation / MMXD complex / core TFIIH complex portion of holo TFIIH complex / RNA Polymerase III Transcription Termination / positive regulation of DNA helicase activity / negative regulation of DNA helicase activity / Cytosolic iron-sulfur cluster assembly / nucleotide-excision repair, DNA duplex unwinding / microfibril binding / RNA Polymerase III Chain Elongation / MMXD complex / core TFIIH complex portion of holo TFIIH complex / RNA Polymerase III Transcription Termination / positive regulation of DNA helicase activity / negative regulation of DNA helicase activity / Cytosolic iron-sulfur cluster assembly / nucleotide-excision repair, DNA duplex unwinding /  central nervous system myelin formation / positive regulation of mitotic recombination / regulation of transcription by RNA polymerase I / RPAP3/R2TP/prefoldin-like complex / positive regulation of core promoter binding / meiotic sister chromatid cohesion / RNA polymerase II core complex assembly / hair cell differentiation / central nervous system myelin formation / positive regulation of mitotic recombination / regulation of transcription by RNA polymerase I / RPAP3/R2TP/prefoldin-like complex / positive regulation of core promoter binding / meiotic sister chromatid cohesion / RNA polymerase II core complex assembly / hair cell differentiation /  ventricular system development / hair follicle maturation / cyclin-dependent protein kinase activating kinase holoenzyme complex / RNA polymerase transcription factor SL1 complex / snRNA transcription by RNA polymerase II / nucleotide-excision repair factor 3 complex / transcription factor TFIIE complex / nucleotide-excision repair, preincision complex assembly / phosphatase activator activity / ventricular system development / hair follicle maturation / cyclin-dependent protein kinase activating kinase holoenzyme complex / RNA polymerase transcription factor SL1 complex / snRNA transcription by RNA polymerase II / nucleotide-excision repair factor 3 complex / transcription factor TFIIE complex / nucleotide-excision repair, preincision complex assembly / phosphatase activator activity /  紫外線 / CAK-ERCC2 complex / transcription factor TFIIK complex / RNA polymerase III general transcription initiation factor activity / embryonic cleavage / transcription open complex formation at RNA polymerase II promoter / TFIIF-class transcription factor complex binding / transcriptional start site selection at RNA polymerase II promoter / Cytosolic sensors of pathogen-associated DNA / RNA Polymerase III Transcription Initiation From Type 1 Promoter / RNA Polymerase III Transcription Initiation From Type 2 Promoter / RNA Polymerase III Transcription Initiation From Type 3 Promoter / transcription factor TFIIF complex / 5'-3' DNA helicase activity / adult heart development / RNA Polymerase III Abortive And Retractive Initiation / transcription factor TFIIA complex / G protein-coupled receptor internalization / : / female germ cell nucleus / transcription factor TFIIH holo complex / transcription factor TFIIH core complex / 紫外線 / CAK-ERCC2 complex / transcription factor TFIIK complex / RNA polymerase III general transcription initiation factor activity / embryonic cleavage / transcription open complex formation at RNA polymerase II promoter / TFIIF-class transcription factor complex binding / transcriptional start site selection at RNA polymerase II promoter / Cytosolic sensors of pathogen-associated DNA / RNA Polymerase III Transcription Initiation From Type 1 Promoter / RNA Polymerase III Transcription Initiation From Type 2 Promoter / RNA Polymerase III Transcription Initiation From Type 3 Promoter / transcription factor TFIIF complex / 5'-3' DNA helicase activity / adult heart development / RNA Polymerase III Abortive And Retractive Initiation / transcription factor TFIIA complex / G protein-coupled receptor internalization / : / female germ cell nucleus / transcription factor TFIIH holo complex / transcription factor TFIIH core complex /  male pronucleus / male pronucleus /  female pronucleus / cyclin-dependent protein serine/threonine kinase activator activity / Abortive elongation of HIV-1 transcript in the absence of Tat / FGFR2 alternative splicing / female pronucleus / cyclin-dependent protein serine/threonine kinase activator activity / Abortive elongation of HIV-1 transcript in the absence of Tat / FGFR2 alternative splicing /  卵母細胞 / [RNA-polymerase]-subunit kinase / RNA polymerase II general transcription initiation factor binding / 3'-5' DNA helicase activity / MicroRNA (miRNA) biogenesis / nuclear thyroid hormone receptor binding / 卵母細胞 / [RNA-polymerase]-subunit kinase / RNA polymerase II general transcription initiation factor binding / 3'-5' DNA helicase activity / MicroRNA (miRNA) biogenesis / nuclear thyroid hormone receptor binding /  転写開始前複合体 / RNA Polymerase I Transcription Termination / regulation of mitotic cell cycle phase transition / cyclin-dependent protein serine/threonine kinase regulator activity / : / positive regulation of nuclear-transcribed mRNA poly(A) tail shortening / Viral Messenger RNA Synthesis / hematopoietic stem cell proliferation / Signaling by FGFR2 IIIa TM / cell division site / RNA Pol II CTD phosphorylation and interaction with CE during HIV infection / RNA Pol II CTD phosphorylation and interaction with CE / spinal cord development / RNA polymerase II general transcription initiation factor activity / transcription factor TFIID complex / Formation of the Early Elongation Complex / Formation of the HIV-1 Early Elongation Complex / mRNA Capping / maintenance of transcriptional fidelity during transcription elongation by RNA polymerase II / PIWI-interacting RNA (piRNA) biogenesis / RNA polymerase II complex binding / mRNA Splicing - Minor Pathway / 転写開始前複合体 / RNA Polymerase I Transcription Termination / regulation of mitotic cell cycle phase transition / cyclin-dependent protein serine/threonine kinase regulator activity / : / positive regulation of nuclear-transcribed mRNA poly(A) tail shortening / Viral Messenger RNA Synthesis / hematopoietic stem cell proliferation / Signaling by FGFR2 IIIa TM / cell division site / RNA Pol II CTD phosphorylation and interaction with CE during HIV infection / RNA Pol II CTD phosphorylation and interaction with CE / spinal cord development / RNA polymerase II general transcription initiation factor activity / transcription factor TFIID complex / Formation of the Early Elongation Complex / Formation of the HIV-1 Early Elongation Complex / mRNA Capping / maintenance of transcriptional fidelity during transcription elongation by RNA polymerase II / PIWI-interacting RNA (piRNA) biogenesis / RNA polymerase II complex binding / mRNA Splicing - Minor Pathway /  protein acetylation / protein acetylation /  bone mineralization / HIV Transcription Initiation / RNA Polymerase II HIV Promoter Escape / Transcription of the HIV genome / RNA Polymerase II Promoter Escape / RNA Polymerase II Transcription Pre-Initiation And Promoter Opening / RNA Polymerase II Transcription Initiation / RNA Polymerase II Transcription Initiation And Promoter Clearance / erythrocyte maturation / regulation of cyclin-dependent protein serine/threonine kinase activity / ATPase activator activity / viral transcription / regulation of G1/S transition of mitotic cell cycle / tRNA transcription by RNA polymerase III / bone mineralization / HIV Transcription Initiation / RNA Polymerase II HIV Promoter Escape / Transcription of the HIV genome / RNA Polymerase II Promoter Escape / RNA Polymerase II Transcription Pre-Initiation And Promoter Opening / RNA Polymerase II Transcription Initiation / RNA Polymerase II Transcription Initiation And Promoter Clearance / erythrocyte maturation / regulation of cyclin-dependent protein serine/threonine kinase activity / ATPase activator activity / viral transcription / regulation of G1/S transition of mitotic cell cycle / tRNA transcription by RNA polymerase III /  acetyltransferase activity / RNA Polymerase I Transcription Initiation / Processing of Capped Intron-Containing Pre-mRNA acetyltransferase activity / RNA Polymerase I Transcription Initiation / Processing of Capped Intron-Containing Pre-mRNA類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
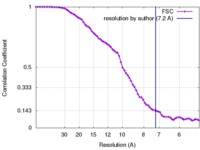
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 7.2 Å クライオ電子顕微鏡法 / 解像度: 7.2 Å | |||||||||
 データ登録者 データ登録者 | Louder RK / He Y / Lopez-Blanco JR / Fang J / Chacon P / Nogales E | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2016 ジャーナル: Nature / 年: 2016タイトル: Structure of promoter-bound TFIID and model of human pre-initiation complex assembly. 著者: Robert K Louder / Yuan He / José Ramón López-Blanco / Jie Fang / Pablo Chacón / Eva Nogales /   要旨: The general transcription factor IID (TFIID) plays a central role in the initiation of RNA polymerase II (Pol II)-dependent transcription by nucleating pre-initiation complex (PIC) assembly at the ...The general transcription factor IID (TFIID) plays a central role in the initiation of RNA polymerase II (Pol II)-dependent transcription by nucleating pre-initiation complex (PIC) assembly at the core promoter. TFIID comprises the TATA-binding protein (TBP) and 13 TBP-associated factors (TAF1-13), which specifically interact with a variety of core promoter DNA sequences. Here we present the structure of human TFIID in complex with TFIIA and core promoter DNA, determined by single-particle cryo-electron microscopy at sub-nanometre resolution. All core promoter elements are contacted by subunits of TFIID, with TAF1 and TAF2 mediating major interactions with the downstream promoter. TFIIA bridges the TBP-TATA complex with lobe B of TFIID. We also present the cryo-electron microscopy reconstruction of a fully assembled human TAF-less PIC. Superposition of common elements between the two structures provides novel insights into the general role of TFIID in promoter recognition, PIC assembly, and transcription initiation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3307.map.gz emd_3307.map.gz | 1.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3307-v30.xml emd-3307-v30.xml emd-3307.xml emd-3307.xml | 20 KB 20 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3307_fsc.xml emd_3307_fsc.xml | 8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3307.png emd_3307.png | 64.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3307 http://ftp.pdbj.org/pub/emdb/structures/EMD-3307 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3307 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3307 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3307.map.gz / 形式: CCP4 / 大きさ: 26.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3307.map.gz / 形式: CCP4 / 大きさ: 26.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of the human TAF-less PIC | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.64 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 ムービー
ムービー コントローラー
コントローラー 試料の構成要素
試料の構成要素

























































 解析
解析 電子顕微鏡法
電子顕微鏡法