+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10390 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of a human nucleosome at 3.2 A resolution | ||||||||||||
 マップデータ マップデータ | Structure of a human nucleosome at 3.2A | ||||||||||||
 試料 試料 |
| ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of megakaryocyte differentiation / protein localization to CENP-A containing chromatin / Chromatin modifying enzymes / Replacement of protamines by nucleosomes in the male pronucleus / CENP-A containing nucleosome / Packaging Of Telomere Ends / Recognition and association of DNA glycosylase with site containing an affected purine / Cleavage of the damaged purine / Deposition of new CENPA-containing nucleosomes at the centromere / Recognition and association of DNA glycosylase with site containing an affected pyrimidine ...negative regulation of megakaryocyte differentiation / protein localization to CENP-A containing chromatin / Chromatin modifying enzymes / Replacement of protamines by nucleosomes in the male pronucleus / CENP-A containing nucleosome / Packaging Of Telomere Ends / Recognition and association of DNA glycosylase with site containing an affected purine / Cleavage of the damaged purine / Deposition of new CENPA-containing nucleosomes at the centromere / Recognition and association of DNA glycosylase with site containing an affected pyrimidine / Cleavage of the damaged pyrimidine / Inhibition of DNA recombination at telomere / Meiotic synapsis / telomere organization / RNA Polymerase I Promoter Opening / Interleukin-7 signaling / SUMOylation of chromatin organization proteins / Assembly of the ORC complex at the origin of replication /  DNAメチル化 / Condensation of Prophase Chromosomes / HCMV Late Events / Chromatin modifications during the maternal to zygotic transition (MZT) / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / SIRT1 negatively regulates rRNA expression / DNAメチル化 / Condensation of Prophase Chromosomes / HCMV Late Events / Chromatin modifications during the maternal to zygotic transition (MZT) / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / SIRT1 negatively regulates rRNA expression /  innate immune response in mucosa / PRC2 methylates histones and DNA / Defective pyroptosis / HDACs deacetylate histones / RNA Polymerase I Promoter Escape / Nonhomologous End-Joining (NHEJ) / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / NoRC negatively regulates rRNA expression / G2/M DNA damage checkpoint / B-WICH complex positively regulates rRNA expression / HDMs demethylate histones / DNA Damage/Telomere Stress Induced Senescence / Metalloprotease DUBs / PKMTs methylate histone lysines / RMTs methylate histone arginines / innate immune response in mucosa / PRC2 methylates histones and DNA / Defective pyroptosis / HDACs deacetylate histones / RNA Polymerase I Promoter Escape / Nonhomologous End-Joining (NHEJ) / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / NoRC negatively regulates rRNA expression / G2/M DNA damage checkpoint / B-WICH complex positively regulates rRNA expression / HDMs demethylate histones / DNA Damage/Telomere Stress Induced Senescence / Metalloprotease DUBs / PKMTs methylate histone lysines / RMTs methylate histone arginines /  遺伝的組換え / Pre-NOTCH Transcription and Translation / 遺伝的組換え / Pre-NOTCH Transcription and Translation /  nucleosome assembly / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / Transcriptional regulation of granulopoiesis / structural constituent of chromatin / UCH proteinases / nucleosome assembly / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / Transcriptional regulation of granulopoiesis / structural constituent of chromatin / UCH proteinases /  ヌクレオソーム / antimicrobial humoral immune response mediated by antimicrobial peptide / E3 ubiquitin ligases ubiquitinate target proteins / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks / RUNX1 regulates transcription of genes involved in differentiation of HSCs / chromatin organization / Factors involved in megakaryocyte development and platelet production / Processing of DNA double-strand break ends / HATs acetylate histones / antibacterial humoral response / Senescence-Associated Secretory Phenotype (SASP) / Oxidative Stress Induced Senescence / killing of cells of another organism / Estrogen-dependent gene expression / defense response to Gram-negative bacterium / ヌクレオソーム / antimicrobial humoral immune response mediated by antimicrobial peptide / E3 ubiquitin ligases ubiquitinate target proteins / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks / RUNX1 regulates transcription of genes involved in differentiation of HSCs / chromatin organization / Factors involved in megakaryocyte development and platelet production / Processing of DNA double-strand break ends / HATs acetylate histones / antibacterial humoral response / Senescence-Associated Secretory Phenotype (SASP) / Oxidative Stress Induced Senescence / killing of cells of another organism / Estrogen-dependent gene expression / defense response to Gram-negative bacterium /  chromosome, telomeric region / Ub-specific processing proteases / defense response to Gram-positive bacterium / Amyloid fiber formation / protein heterodimerization activity / negative regulation of cell population proliferation / protein-containing complex / chromosome, telomeric region / Ub-specific processing proteases / defense response to Gram-positive bacterium / Amyloid fiber formation / protein heterodimerization activity / negative regulation of cell population proliferation / protein-containing complex /  DNA binding / DNA binding /  extracellular space / extracellular space /  RNA binding / extracellular exosome / extracellular region / RNA binding / extracellular exosome / extracellular region /  核質 / 核質 /  生体膜 / 生体膜 /  細胞核 / 細胞核 /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | ||||||||||||
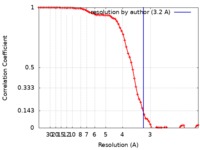
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.2 Å クライオ電子顕微鏡法 / 解像度: 3.2 Å | ||||||||||||
 データ登録者 データ登録者 | Dodonova SO / Zhu F / Dienemann C / Taipale J / Cramer P | ||||||||||||
| 資金援助 |  ドイツ, 3件 ドイツ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2020 ジャーナル: Nature / 年: 2020タイトル: Nucleosome-bound SOX2 and SOX11 structures elucidate pioneer factor function. 著者: Svetlana O Dodonova / Fangjie Zhu / Christian Dienemann / Jussi Taipale / Patrick Cramer /   要旨: 'Pioneer' transcription factors are required for stem-cell pluripotency, cell differentiation and cell reprogramming. Pioneer factors can bind nucleosomal DNA to enable gene expression from regions ...'Pioneer' transcription factors are required for stem-cell pluripotency, cell differentiation and cell reprogramming. Pioneer factors can bind nucleosomal DNA to enable gene expression from regions of the genome with closed chromatin. SOX2 is a prominent pioneer factor that is essential for pluripotency and self-renewal of embryonic stem cells. Here we report cryo-electron microscopy structures of the DNA-binding domains of SOX2 and its close homologue SOX11 bound to nucleosomes. The structures show that SOX factors can bind and locally distort DNA at superhelical location 2. The factors also facilitate detachment of terminal nucleosomal DNA from the histone octamer, which increases DNA accessibility. SOX-factor binding to the nucleosome can also lead to a repositioning of the N-terminal tail of histone H4 that includes residue lysine 16. We speculate that this repositioning is incompatible with higher-order nucleosome stacking, which involves contacts of the H4 tail with a neighbouring nucleosome. Our results indicate that pioneer transcription factors can use binding energy to initiate chromatin opening, and thereby facilitate nucleosome remodelling and subsequent transcription. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10390.map.gz emd_10390.map.gz | 9.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10390-v30.xml emd-10390-v30.xml emd-10390.xml emd-10390.xml | 26.2 KB 26.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10390_fsc.xml emd_10390_fsc.xml | 6.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10390.png emd_10390.png | 172.9 KB | ||
| その他 |  emd_10390_half_map_1.map.gz emd_10390_half_map_1.map.gz emd_10390_half_map_2.map.gz emd_10390_half_map_2.map.gz | 9.7 MB 9.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10390 http://ftp.pdbj.org/pub/emdb/structures/EMD-10390 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10390 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10390 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10390.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10390.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of a human nucleosome at 3.2A | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: half-map 2, nucleosome
| ファイル | emd_10390_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map 2, nucleosome | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
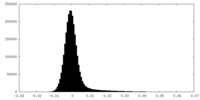
| 密度ヒストグラム |
-ハーフマップ: half-map 1, nucleosome
| ファイル | emd_10390_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map 1, nucleosome | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
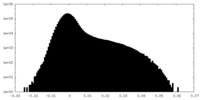
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Nucleosome
| 全体 | 名称: Nucleosome ヌクレオソーム ヌクレオソーム |
|---|---|
| 要素 |
|
-超分子 #1: Nucleosome
| 超分子 | 名称: Nucleosome / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Nucleosome |
|---|---|
| 分子量 | 理論値: 205 KDa |
-超分子 #2: Proteins
| 超分子 | 名称: Proteins / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#4 / 詳細: Proteins |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
-超分子 #3: DNA
| 超分子 | 名称: DNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #5-#6 / 詳細: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 組換発現 | 生物種: synthetic construct (人工物) |
-分子 #1: Histone H3.2
| 分子 | 名称: Histone H3.2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 15.389036 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MARTKQTARK STGGKAPRKQ LATKAARKSA PATGGVKKPH RYRPGTVALR EIRRYQKSTE LLIRKLPFQR LVREIAQDFK TDLRFQSSA VMALQEASEA YLVGLFEDTN LAAIHAKRVT IMPKDIQLAR RIRGERA |
-分子 #2: Histone H4
| 分子 | 名称: Histone H4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.394426 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MSGRGKGGKG LGKGGAKRHR KVLRDNIQGI TKPAIRRLAR RGGVKRISGL IYEETRGVLK VFLENVIRDA VTYTEHAKRK TVTAMDVVY ALKRQGRTLY GFGG |
-分子 #3: Histone H2A type 1-B/E
| 分子 | 名称: Histone H2A type 1-B/E / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 16.707277 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MGSSHHHHHH ENLYFQSNAP WMSGRGKQGG KARAKAKTRS SRAGLQFPVG RVHRLLRKGN YSERVGAGAP VYLAAVLEYL TAEILELAG NAARDNKKTR IIPRHLQLAI RNDEELNKLL GRVTIAQGGV LPNIQAVLLP KKTESHHKAK GK |
-分子 #4: Histone H2B type 1-K
| 分子 | 名称: Histone H2B type 1-K / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 13.921213 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MPEPAKSAPA PKKGSKKAVT KAQKKDGKKR KRSRKESYSV YVYKVLKQVH PDTGISSKAM GIMNSFVNDI FERIAGEASR LAHYNKRST ITSREIQTAV RLLLPGELAK HAVSEGTKAV TKYTSAK |
-分子 #5: DNA (147-MER)
| 分子 | 名称: DNA (147-MER) / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 45.240848 KDa |
| 配列 | 文字列: (DA)(DT)(DC)(DT)(DA)(DC)(DA)(DC)(DG)(DA) (DC)(DG)(DC)(DT)(DC)(DT)(DT)(DC)(DC)(DG) (DA)(DT)(DC)(DT)(DA)(DA)(DT)(DT)(DT) (DA)(DT)(DG)(DT)(DT)(DT)(DG)(DT)(DT)(DA) (DG) (DC)(DG)(DT)(DT)(DA) ...文字列: (DA)(DT)(DC)(DT)(DA)(DC)(DA)(DC)(DG)(DA) (DC)(DG)(DC)(DT)(DC)(DT)(DT)(DC)(DC)(DG) (DA)(DT)(DC)(DT)(DA)(DA)(DT)(DT)(DT) (DA)(DT)(DG)(DT)(DT)(DT)(DG)(DT)(DT)(DA) (DG) (DC)(DG)(DT)(DT)(DA)(DT)(DA)(DC) (DT)(DA)(DT)(DT)(DC)(DT)(DA)(DA)(DT)(DT) (DC)(DT) (DT)(DT)(DG)(DT)(DT)(DT)(DC) (DG)(DG)(DT)(DG)(DG)(DT)(DA)(DT)(DT)(DG) (DT)(DT)(DT) (DA)(DT)(DT)(DT)(DT)(DG) (DT)(DT)(DC)(DC)(DT)(DT)(DT)(DG)(DT)(DG) (DC)(DG)(DT)(DT) (DC)(DA)(DG)(DC)(DT) (DT)(DA)(DA)(DT)(DG)(DC)(DC)(DT)(DA)(DA) (DC)(DG)(DA)(DC)(DA) (DC)(DT)(DC)(DG) (DG)(DA)(DG)(DA)(DT)(DC)(DG)(DG)(DA)(DA) (DG)(DA)(DG)(DC)(DA)(DC) (DA)(DC)(DG) (DT)(DG)(DA)(DT) |
-分子 #6: DNA (147-MER)
| 分子 | 名称: DNA (147-MER) / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 45.484273 KDa |
| 配列 | 文字列: (DA)(DT)(DC)(DA)(DC)(DG)(DT)(DG)(DT)(DG) (DC)(DT)(DC)(DT)(DT)(DC)(DC)(DG)(DA)(DT) (DC)(DT)(DC)(DC)(DG)(DA)(DG)(DT)(DG) (DT)(DC)(DG)(DT)(DT)(DA)(DG)(DG)(DC)(DA) (DT) (DT)(DA)(DA)(DG)(DC) ...文字列: (DA)(DT)(DC)(DA)(DC)(DG)(DT)(DG)(DT)(DG) (DC)(DT)(DC)(DT)(DT)(DC)(DC)(DG)(DA)(DT) (DC)(DT)(DC)(DC)(DG)(DA)(DG)(DT)(DG) (DT)(DC)(DG)(DT)(DT)(DA)(DG)(DG)(DC)(DA) (DT) (DT)(DA)(DA)(DG)(DC)(DT)(DG)(DA) (DA)(DC)(DG)(DC)(DA)(DC)(DA)(DA)(DA)(DG) (DG)(DA) (DA)(DC)(DA)(DA)(DA)(DA)(DT) (DA)(DA)(DA)(DC)(DA)(DA)(DT)(DA)(DC)(DC) (DA)(DC)(DC) (DG)(DA)(DA)(DA)(DC)(DA) (DA)(DA)(DG)(DA)(DA)(DT)(DT)(DA)(DG)(DA) (DA)(DT)(DA)(DG) (DT)(DA)(DT)(DA)(DA) (DC)(DG)(DC)(DT)(DA)(DA)(DC)(DA)(DA)(DA) (DC)(DA)(DT)(DA)(DA) (DA)(DT)(DT)(DA) (DG)(DA)(DT)(DC)(DG)(DG)(DA)(DA)(DG)(DA) (DG)(DC)(DG)(DT)(DC)(DG) (DT)(DG)(DT) (DA)(DG)(DA)(DT) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.15 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa / 詳細: 0.39 mB, 25 mA | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 289 K / 装置: FEI VITROBOT MARK IV 詳細: The sample was applied onto glow-discharged Quantifoil holey carbon grids. The grids were blotted from both sides for 5-10 seconds at 16*C in a chamber at 100% humidity and plunge-frozen into ...詳細: The sample was applied onto glow-discharged Quantifoil holey carbon grids. The grids were blotted from both sides for 5-10 seconds at 16*C in a chamber at 100% humidity and plunge-frozen into liquid ethane using a manual plunger.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 30 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 詳細 | At least 50% of the data were collected at 25* stage tilt in order to partially compensate for preferred orientation of particles on the grid, and to improve angular distribution. |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 平均電子線量: 1.125 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



































 Z
Z Y
Y X
X