+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4706 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of T7 bacteriophage fiberless tail complex | ||||||||||||||||||||||||
 マップデータ マップデータ | Bacteriophage ejection complex | ||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報virus tail, tube / viral portal complex / symbiont genome ejection through host cell envelope, short tail mechanism / viral DNA genome packaging 類似検索 - 分子機能 | ||||||||||||||||||||||||
| 生物種 |    Enterobacteria phage T7 (ファージ) Enterobacteria phage T7 (ファージ) | ||||||||||||||||||||||||
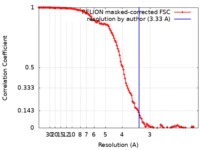
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.33 Å クライオ電子顕微鏡法 / 解像度: 3.33 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Cuervo A / Fabrega-Ferrer M / Machon C / Conesa JJ / Perez-Ruiz M / Coll M / Carrascosa JL | ||||||||||||||||||||||||
| 資金援助 |  スペイン, 7件 スペイン, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: Structures of T7 bacteriophage portal and tail suggest a viral DNA retention and ejection mechanism. 著者: Ana Cuervo / Montserrat Fàbrega-Ferrer / Cristina Machón / José Javier Conesa / Francisco J Fernández / Rosa Pérez-Luque / Mar Pérez-Ruiz / Joan Pous / M Cristina Vega / José L Carrascosa / Miquel Coll /  要旨: Double-stranded DNA bacteriophages package their genome at high pressure inside a procapsid through the portal, an oligomeric ring protein located at a unique capsid vertex. Once the DNA has been ...Double-stranded DNA bacteriophages package their genome at high pressure inside a procapsid through the portal, an oligomeric ring protein located at a unique capsid vertex. Once the DNA has been packaged, the tail components assemble on the portal to render the mature infective virion. The tail tightly seals the ejection conduit until infection, when its interaction with the host membrane triggers the opening of the channel and the viral genome is delivered to the host cell. Using high-resolution cryo-electron microscopy and X-ray crystallography, here we describe various structures of the T7 bacteriophage portal and fiber-less tail complex, which suggest a possible mechanism for DNA retention and ejection: a portal closed conformation temporarily retains the genome before the tail is assembled, whereas an open portal is found in the tail. Moreover, a fold including a seven-bladed β-propeller domain is described for the nozzle tail protein. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4706.map.gz emd_4706.map.gz | 53.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4706-v30.xml emd-4706-v30.xml emd-4706.xml emd-4706.xml | 20.9 KB 20.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4706_fsc.xml emd_4706_fsc.xml | 12.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4706.png emd_4706.png | 1.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4706 http://ftp.pdbj.org/pub/emdb/structures/EMD-4706 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4706 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4706 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4706.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4706.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Bacteriophage ejection complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.048 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Bacteriophage tail complex
| 全体 | 名称: Bacteriophage tail complex |
|---|---|
| 要素 |
|
-超分子 #1: Bacteriophage tail complex
| 超分子 | 名称: Bacteriophage tail complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:    Enterobacteria phage T7 (ファージ) Enterobacteria phage T7 (ファージ) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 534 KDa |
-超分子 #2: Portal protein
| 超分子 | 名称: Portal protein / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:    Enterobacteria phage T7 (ファージ) Enterobacteria phage T7 (ファージ) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-超分子 #3: Adaptor protein
| 超分子 | 名称: Adaptor protein / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:    Enterobacteria phage T7 (ファージ) Enterobacteria phage T7 (ファージ) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-超分子 #4: Nozzle protein
| 超分子 | 名称: Nozzle protein / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:    Enterobacteria phage T7 (ファージ) Enterobacteria phage T7 (ファージ) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-分子 #1: Portal protein
| 分子 | 名称: Portal protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Enterobacteria phage T7 (ファージ) Enterobacteria phage T7 (ファージ) |
| 分子量 | 理論値: 59.173984 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MAEKRTGLAE DGAKSVYERL KNDRAPYETR AQNCAQYTIP SLFPKDSDNA STDYQTPWQA VGARGLNNLA SKLMLALFPM QTWMRLTIS EYEAKQLLSD PDGLAKVDEG LSMVERIIMN YIESNSYRVT LFEALKQLVV AGNVLLYLPE PEGSNYNPMK L YRLSSYVV ...文字列: MAEKRTGLAE DGAKSVYERL KNDRAPYETR AQNCAQYTIP SLFPKDSDNA STDYQTPWQA VGARGLNNLA SKLMLALFPM QTWMRLTIS EYEAKQLLSD PDGLAKVDEG LSMVERIIMN YIESNSYRVT LFEALKQLVV AGNVLLYLPE PEGSNYNPMK L YRLSSYVV QRDAFGNVLQ MVTRDQIAFG ALPEDIRKAV EGQGGEKKAD ETIDVYTHIY LDEDSGEYLR YEEVEGMEVQ GS DGTYPKE ACPYIPIRMV RLDGESYGRS YIEEYLGDLR SLENLQEAIV KMSMISSKVI GLVNPAGITQ PRRLTKAQTG DFV TGRPED ISFLQLEKQA DFTVAKAVSD AIEARLSFAF MLNSAVQRTG ERVTAEEIRY VASELEDTLG GVYSILSQEL QLPL VRVLL KQLQATQQIP ELPKEAVEPT ISTGLEAIGR GQDLDKLERC VTAWAALAPM RDDPDINLAM IKLRIANAIG IDTSG ILLT EEQKQQKMAQ QSMQMGMDNG AAALAQGMAA QATASPEAMA AAADSVGLQP GI |
-分子 #2: Tail tubular protein gp11
| 分子 | 名称: Tail tubular protein gp11 / タイプ: protein_or_peptide / ID: 2 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Enterobacteria phage T7 (ファージ) Enterobacteria phage T7 (ファージ) |
| 分子量 | 理論値: 26.20282 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MRGSHHHHHH GMASMTGGNN MGRDLYDDDD KDPSSMRSYD MNVETAAELS AVNDILASIG EPPVSTLEGD ANADAANARR ILNKINRQI QSRGWTFNIE EGITLLPDVY SNLIVYSDDY LSLMSTSGQS IYVNRGGYVY DRTSQSDRFD SGITVNIIRL R DYDEMPEC ...文字列: MRGSHHHHHH GMASMTGGNN MGRDLYDDDD KDPSSMRSYD MNVETAAELS AVNDILASIG EPPVSTLEGD ANADAANARR ILNKINRQI QSRGWTFNIE EGITLLPDVY SNLIVYSDDY LSLMSTSGQS IYVNRGGYVY DRTSQSDRFD SGITVNIIRL R DYDEMPEC FRYWIVTKAS RQFNNRFFGA PEVEGVLQEE EDEARRLCME YEMDYGGYNM LDGDAFTSGL LTR |
-分子 #3: Tail tubular protein gp12
| 分子 | 名称: Tail tubular protein gp12 / タイプ: protein_or_peptide / ID: 3 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Enterobacteria phage T7 (ファージ) Enterobacteria phage T7 (ファージ) |
| 分子量 | 理論値: 89.480594 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MALISQSIKN LKGGISQQPD ILRYPDQGSR QVNGWSSETE GLQKRPPLVF LNTLGDNGAL GQAPYIHLIN RDEHEQYYAV FTGSGIRVF DLSGNEKQVR YPNGSNYIKT ANPRNDLRMV TVADYTFIVN RNVVAQKNTK SVNLPNYNPN QDGLINVRGG Q YGRELIVH ...文字列: MALISQSIKN LKGGISQQPD ILRYPDQGSR QVNGWSSETE GLQKRPPLVF LNTLGDNGAL GQAPYIHLIN RDEHEQYYAV FTGSGIRVF DLSGNEKQVR YPNGSNYIKT ANPRNDLRMV TVADYTFIVN RNVVAQKNTK SVNLPNYNPN QDGLINVRGG Q YGRELIVH INGKDVAKYK IPDGSQPEHV NNTDAQWLAE ELAKQMRTNL SDWTVNVGQG FIHVTAPSGQ QIDSFTTKDG YA DQLINPV THYAQSFSKL PPNAPNGYMV KIVGDASKSA DQYYVRYDAE RKVWTETLGW NTEDQVLWET MPHALVRAAD GNF DFKWLE WSPKSCGDVD TNPWPSFVGS SINDVFFFRN RLGFLSGENI ILSRTAKYFN FYPASIANLS DDDPIDVAVS TNRI AILKY AVPFSEELLI WSDEAQFVLT ASGTLTSKSV ELNLTTQFDV QDRARPFGIG RNVYFASPRS SFTSIHRYYA VQDVS SVKN AEDITSHVPN YIPNGVFSIC GSGTENFCSV LSHGDPSKIF MYKFLYLNEE LRQQSWSHWD FGENVQVLAC QSISSD MYV ILRNEFNTFL ARISFTKNAI DLQGEPYRAF MDMKIRYTIP SGTYNDDTFT TSIHIPTIYG ANFGRGKITV LEPDGKI TV FEQPTAGWNS DPWLRLSGNL EGRMVYIGFN INFVYEFSKF LIKQTADDGS TSTEDIGRLQ LRRAWVNYEN SGTFDIYV E NQSSNWKYTM AGARLGSNTL RAGRLNLGTG QYRFPVVGNA KFNTVYILSD ETTPLNIIGC GWEGNYLRRS SGI |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.8 構成要素:
詳細: 50 mM Tris-HCl pH 7.8, 100 mM NaCl, 10 mM MgCl2 | ||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER/RHODIUM / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY 詳細: acetone and treated with 0.1% w/v poly-L-Lysine (Sigma) for 1 min in water | ||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 295.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 0.84 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-6r21: |
-原子モデル構築 2
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-6r21: |
-原子モデル構築 3
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-6r21: |
 ムービー
ムービー コントローラー
コントローラー