+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7suc | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
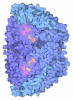
| タイトル | XFEL Serial Crystallography Reveals the Room Temperature Structure of Methyl-Coenzyme M Reductase | ||||||||||||||||||||||||||||||
 要素 要素 | (Methyl-coenzyme M reductase I subunit ...) x 3 | ||||||||||||||||||||||||||||||
 キーワード キーワード | TRANSFERASE / Methyl-Coenzyme M Reductase (MCR) / Methanogens / METAL BINDING PROTEIN | ||||||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報coenzyme-B sulfoethylthiotransferase / coenzyme-B sulfoethylthiotransferase activity / methanogenesis / metal ion binding / cytoplasm 類似検索 - 分子機能 | ||||||||||||||||||||||||||||||
| 生物種 |   Methanothermobacter marburgensis str. Marburg (古細菌) Methanothermobacter marburgensis str. Marburg (古細菌) | ||||||||||||||||||||||||||||||
| 手法 |  X線回折 / X線回折 /  自由電子レーザー / 自由電子レーザー /  分子置換 / 解像度: 1.9 Å 分子置換 / 解像度: 1.9 Å | ||||||||||||||||||||||||||||||
 データ登録者 データ登録者 | Ohmer, C.J. / Dasgupta, M. | ||||||||||||||||||||||||||||||
| 資金援助 |  米国, 9件 米国, 9件
| ||||||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: J.Inorg.Biochem. / 年: 2022 ジャーナル: J.Inorg.Biochem. / 年: 2022タイトル: XFEL serial crystallography reveals the room temperature structure of methyl-coenzyme M reductase. 著者: Ohmer, C.J. / Dasgupta, M. / Patwardhan, A. / Bogacz, I. / Kaminsky, C. / Doyle, M.D. / Chen, P.Y. / Keable, S.M. / Makita, H. / Simon, P.S. / Massad, R. / Fransson, T. / Chatterjee, R. / ...著者: Ohmer, C.J. / Dasgupta, M. / Patwardhan, A. / Bogacz, I. / Kaminsky, C. / Doyle, M.D. / Chen, P.Y. / Keable, S.M. / Makita, H. / Simon, P.S. / Massad, R. / Fransson, T. / Chatterjee, R. / Bhowmick, A. / Paley, D.W. / Moriarty, N.W. / Brewster, A.S. / Gee, L.B. / Alonso-Mori, R. / Moss, F. / Fuller, F.D. / Batyuk, A. / Sauter, N.K. / Bergmann, U. / Drennan, C.L. / Yachandra, V.K. / Yano, J. / Kern, J.F. / Ragsdale, S.W. | ||||||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7suc.cif.gz 7suc.cif.gz | 1.1 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7suc.ent.gz pdb7suc.ent.gz | 765.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7suc.json.gz 7suc.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7suc_validation.pdf.gz 7suc_validation.pdf.gz | 1.9 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7suc_full_validation.pdf.gz 7suc_full_validation.pdf.gz | 1.9 MB | 表示 | |
| XML形式データ |  7suc_validation.xml.gz 7suc_validation.xml.gz | 87.6 KB | 表示 | |
| CIF形式データ |  7suc_validation.cif.gz 7suc_validation.cif.gz | 124.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/su/7suc https://data.pdbj.org/pub/pdb/validation_reports/su/7suc ftp://data.pdbj.org/pub/pdb/validation_reports/su/7suc ftp://data.pdbj.org/pub/pdb/validation_reports/su/7suc | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||
| 単位格子 |
|
- 要素
要素
-Methyl-coenzyme M reductase I subunit ... , 3種, 6分子 AaBbCc
| #1: タンパク質 | 分子量: 60377.273 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   Methanothermobacter marburgensis str. Marburg (古細菌) Methanothermobacter marburgensis str. Marburg (古細菌)株: ATCC BAA-927 / DSM 2133 / JCM 14651 / NBRC 100331 / OCM 82 / Marburg 参照: UniProt: P11558, coenzyme-B sulfoethylthiotransferase #2: タンパク質 | 分子量: 47148.477 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   Methanothermobacter marburgensis str. Marburg (古細菌) Methanothermobacter marburgensis str. Marburg (古細菌)株: ATCC BAA-927 / DSM 2133 / JCM 14651 / NBRC 100331 / OCM 82 / Marburg 参照: UniProt: P11560, coenzyme-B sulfoethylthiotransferase #3: タンパク質 | 分子量: 28423.768 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   Methanothermobacter marburgensis str. Marburg (古細菌) Methanothermobacter marburgensis str. Marburg (古細菌)株: ATCC BAA-927 / DSM 2133 / JCM 14651 / NBRC 100331 / OCM 82 / Marburg 参照: UniProt: P11562, coenzyme-B sulfoethylthiotransferase |
|---|
-非ポリマー , 7種, 749分子 










| #4: 化合物 | ChemComp-MG / #5: 化合物 | #6: 化合物 | #7: 化合物 | #8: 化合物 | ChemComp-ACT / | #9: 化合物 | ChemComp-EDO / | #10: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.23 Å3/Da / 溶媒含有率: 44.81 % 解説: Pale yellow, Plate-like crystals ranging 20-60 microns in size |
|---|---|
| 結晶化 | 温度: 300 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7.55 / 詳細: 18% PEG 400, 150mM Mg Acetate, 100mM HEPES (pH 7.5) / PH範囲: 7.5-8.0 / Temp details: Isotemp |
-データ収集
| 回折 | 平均測定温度: 300 K / Ambient temp details: Isotemp / Serial crystal experiment: Y |
|---|---|
| 放射光源 | 由来:  自由電子レーザー / サイト: 自由電子レーザー / サイト:  SLAC LCLS SLAC LCLS  / ビームライン: MFX / 波長: 1.3007 Å / ビームライン: MFX / 波長: 1.3007 Å |
| 検出器 | タイプ: RAYONIX MX340-HS / 検出器: CCD / 日付: 2020年7月11日 / Frequency: 30 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.3007 Å / 相対比: 1 |
| 反射 | 解像度: 1.9→21.25 Å / Num. obs: 189345 / % possible obs: 99.91 % / 冗長度: 41.43 % / Biso Wilson estimate: 26.7 Å2 / CC1/2: 0.979 / R split: 0.515 / Net I/σ(I): 3.356 |
| 反射 シェル | 解像度: 1.9→1.93 Å / 冗長度: 9.89 % / Mean I/σ(I) obs: 0.696 / Num. unique obs: 9505 / CC1/2: 0.379 / R split: 0.831 / % possible all: 99.82 |
| Serial crystallography measurement | Collection time total: 0.68 hours / Focal spot size: 16 µm2 / Pulse duration: 30 fsec. / Pulse energy: 2200 µJ / Pulse photon energy: 9.5 keV / XFEL pulse repetition rate: 30 Hz |
| Serial crystallography sample delivery | 解説: Drop On Tape / 手法: injection |
| Serial crystallography sample delivery injection | Carrier solvent: mother liquor 解説: Crystal slurry is injected onto a kapton tape from an acoustic droplet injector (ADE). The drop travels on the tape towards the X-ray interaction point where it is hit by the XFEL beam. This ...解説: Crystal slurry is injected onto a kapton tape from an acoustic droplet injector (ADE). The drop travels on the tape towards the X-ray interaction point where it is hit by the XFEL beam. This is the Drop-On-Tape (DOT) sample delivery system used by our team previously. |
| Serial crystallography data reduction | Crystal hits: 24589 / Frames total: 73742 / Lattices indexed: 22706 / XFEL run numbers: 44-53 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 3M1V 解像度: 1.9→21.25 Å / SU ML: 0.2017 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 18.6978 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.1 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 30.52 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.9→21.25 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj