+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 1hbn | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
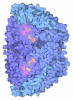
| Title | METHYL-COENZYME M REDUCTASE | |||||||||
 Components Components | (METHYL-COENZYME M REDUCTASE I ...) x 3 | |||||||||
 Keywords Keywords | METHANOGENESIS / BIOLOGICAL METHANOGENESIS / NI-ENZYME / OXIDOREDUCTASE | |||||||||
| Function / homology |  Function and homology information Function and homology informationcoenzyme-B sulfoethylthiotransferase / coenzyme-B sulfoethylthiotransferase activity / methanogenesis / metal ion binding / cytoplasm Similarity search - Function | |||||||||
| Biological species |   METHANOTHERMOBACTER THERMAUTOTROPHICUS (archaea) METHANOTHERMOBACTER THERMAUTOTROPHICUS (archaea) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MIR / Resolution: 1.16 Å MIR / Resolution: 1.16 Å | |||||||||
 Authors Authors | Ermler, U. / Grabarse, W. | |||||||||
 Citation Citation |  Journal: J.Mol.Biol. / Year: 2001 Journal: J.Mol.Biol. / Year: 2001Title: On the Mechanism of Biological Methane Formation: Structural Evidence for Conformational Changes in Methyl-Coenzyme M Reductase Upon Substrate Binding Authors: Grabarse, W. / Mahlert, F. / Duin, E.C. / Goubeaud, M. / Shima, S. / Thauer, R.K. / Lamzin, V. / Ermler, U. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  1hbn.cif.gz 1hbn.cif.gz | 1 MB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb1hbn.ent.gz pdb1hbn.ent.gz | 872.9 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  1hbn.json.gz 1hbn.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/hb/1hbn https://data.pdbj.org/pub/pdb/validation_reports/hb/1hbn ftp://data.pdbj.org/pub/pdb/validation_reports/hb/1hbn ftp://data.pdbj.org/pub/pdb/validation_reports/hb/1hbn | HTTPS FTP |
|---|
-Related structure data
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
| ||||||||
| Noncrystallographic symmetry (NCS) | NCS oper: (Code: given Matrix: (0.6268, -0.7779, 0.0458), Vector: |
- Components
Components
-METHYL-COENZYME M REDUCTASE I ... , 3 types, 6 molecules ADBECF
| #1: Protein | Mass: 60508.469 Da / Num. of mol.: 2 / Source method: isolated from a natural source Source: (natural)   METHANOTHERMOBACTER THERMAUTOTROPHICUS (archaea) METHANOTHERMOBACTER THERMAUTOTROPHICUS (archaea)Cellular location: CYTOPLASM / Strain: MARBURG / References: UniProt: P11558 #2: Protein | Mass: 47148.477 Da / Num. of mol.: 2 / Source method: isolated from a natural source Source: (natural)   METHANOTHERMOBACTER THERMAUTOTROPHICUS (archaea) METHANOTHERMOBACTER THERMAUTOTROPHICUS (archaea)Cellular location: CYTOPLASM / Strain: MARBURG / References: UniProt: P11560 #3: Protein | Mass: 28666.039 Da / Num. of mol.: 2 / Source method: isolated from a natural source / Details: MCR-OX-SILENT STATE, ENZYME-SUBSTRATE COMPLEX Source: (natural)   METHANOTHERMOBACTER THERMAUTOTROPHICUS (archaea) METHANOTHERMOBACTER THERMAUTOTROPHICUS (archaea)Cellular location: CYTOPLASM / Strain: MARBURG / References: UniProt: P11562 |
|---|
-Non-polymers , 9 types, 2441 molecules 















| #4: Chemical | | #5: Chemical | #6: Chemical | #7: Chemical | ChemComp-GOL / #8: Chemical | ChemComp-ZN / | #9: Chemical | ChemComp-MG / #10: Chemical | ChemComp-NA / #11: Chemical | #12: Water | ChemComp-HOH / | |
|---|
-Details
| Has protein modification | Y |
|---|---|
| Sequence details | REFERENCE: 1 RESIDUE IS LACKING IN THE C-TERMINUS AND THE N-TERMINUS, C AND N-TERMINUS ARE VISIBLE ...REFERENCE: 1 RESIDUE IS LACKING IN THE C-TERMINUS AND THE N-TERMINUS, C AND N-TERMINUS ARE VISIBLE IN THE CRYSTAL STRUCTURE. MET 1 WAS CLEARLY SHOWN NOT TO BE PRESENT. |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 3.9 Å3/Da / Density % sol: 31 % | ||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Crystal grow | pH: 7.5 Details: 25% PEG 400, 0.2 M NACL, 20 MG/ML FINAL PROTEIN CONC 0.2 M MGCL, 0.1 M HEPES PH 7.5 | ||||||||||||||||||||||||||||||||||||||||||||||||
| Crystal grow | *PLUS Temperature: 4 ℃ / Method: vapor diffusion, hanging drop | ||||||||||||||||||||||||||||||||||||||||||||||||
| Components of the solutions | *PLUS
|
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  EMBL/DESY, HAMBURG EMBL/DESY, HAMBURG  / Beamline: BW7B / Wavelength: 1.08 / Beamline: BW7B / Wavelength: 1.08 |
| Detector | Type: MAR scanner 345 mm plate / Detector: IMAGE PLATE / Date: Apr 15, 1997 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.08 Å / Relative weight: 1 |
| Reflection | Resolution: 1.16→30 Å / Num. obs: 735767 / % possible obs: 93 % / Redundancy: 3 % / Rmerge(I) obs: 0.066 / Rsym value: 0.07 / Net I/σ(I): 18 |
| Reflection shell | Resolution: 1.16→1.23 Å / Redundancy: 2.5 % / Rmerge(I) obs: 0.33 / Mean I/σ(I) obs: 3 / Rsym value: 0.34 / % possible all: 89 |
| Reflection | *PLUS Lowest resolution: 30 Å / % possible obs: 93.1 % / Redundancy: 3.9 % / Rmerge(I) obs: 0.067 |
| Reflection shell | *PLUS Lowest resolution: 1.17 Å / % possible obs: 89.5 % / Redundancy: 2.1 % / Rmerge(I) obs: 0.333 / Mean I/σ(I) obs: 3.4 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MIR / Resolution: 1.16→30 Å / Cross valid method: THROUGHOUT / σ(F): 0 MIR / Resolution: 1.16→30 Å / Cross valid method: THROUGHOUT / σ(F): 0
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.16→30 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Software | *PLUS Name: REFMAC / Classification: refinement | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement | *PLUS Rfactor obs: 0.124 / Rfactor Rfree: 0.149 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | *PLUS | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | *PLUS |
 Movie
Movie Controller
Controller
















 PDBj
PDBj