| 登録情報 | データベース: PDB / ID: 6v2s
|
|---|
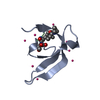
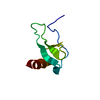
| タイトル | Crystal Structure of chromodomain of MPP8 in complex with inhibitor UNC3866 |
|---|
 要素 要素 | - M-phase phosphoprotein 8
- UNC3866
|
|---|
 キーワード キーワード | GENE REGULATION / structural genomics / Structural Genomics Consortium / SGC |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
constitutive heterochromatin formation / transposable element silencing by heterochromatin formation / histone H3K9me2/3 reader activity / negative regulation of gene expression via chromosomal CpG island methylation / negative regulation of gene expression, epigenetic / : / heterochromatin / Regulation of endogenous retroelements by the Human Silencing Hub (HUSH) complex / nucleosome / chromatin binding ...constitutive heterochromatin formation / transposable element silencing by heterochromatin formation / histone H3K9me2/3 reader activity / negative regulation of gene expression via chromosomal CpG island methylation / negative regulation of gene expression, epigenetic / : / heterochromatin / Regulation of endogenous retroelements by the Human Silencing Hub (HUSH) complex / nucleosome / chromatin binding / nucleolus / nucleoplasm / nucleus / plasma membrane / cytoplasm / cytosol類似検索 - 分子機能 : / Chromo domain, conserved site / Chromo domain signature. / Chromo domain / Chromo (CHRromatin Organisation MOdifier) domain / Chromo and chromo shadow domain profile. / OB fold (Dihydrolipoamide Acetyltransferase, E2P) - #40 / Chromo/chromo shadow domain / Chromatin organization modifier domain / Chromo-like domain superfamily ...: / Chromo domain, conserved site / Chromo domain signature. / Chromo domain / Chromo (CHRromatin Organisation MOdifier) domain / Chromo and chromo shadow domain profile. / OB fold (Dihydrolipoamide Acetyltransferase, E2P) - #40 / Chromo/chromo shadow domain / Chromatin organization modifier domain / Chromo-like domain superfamily / Ankyrin repeat / Ankyrin repeats (3 copies) / Ankyrin repeat profile. / Ankyrin repeat region circular profile. / ankyrin repeats / Ankyrin repeat / Ankyrin repeat-containing domain superfamily / OB fold (Dihydrolipoamide Acetyltransferase, E2P) / Beta Barrel / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト)
synthetic construct (人工物) |
|---|
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 1.6 Å 分子置換 / 解像度: 1.6 Å |
|---|
 データ登録者 データ登録者 | Liu, Y. / Tempel, W. / Walker, J.R. / Bountra, C. / Arrowsmith, C.H. / Edwards, A.M. / Min, J. / Structural Genomics Consortium (SGC) |
|---|
 引用 引用 |  ジャーナル: Cell Chem Biol / 年: 2020 ジャーナル: Cell Chem Biol / 年: 2020
タイトル: Structural Basis for the Binding Selectivity of Human CDY Chromodomains.
著者: Dong, C. / Liu, Y. / Lyu, T.J. / Beldar, S. / Lamb, K.N. / Tempel, W. / Li, Y. / Li, Z. / James, L.I. / Qin, S. / Wang, Y. / Min, J. |
|---|
| 履歴 | | 登録 | 2019年11月25日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2019年12月25日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2020年6月17日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year |
|---|
| 改定 1.2 | 2020年7月29日 | Group: Database references / カテゴリ: citation
Item: _citation.journal_volume / _citation.page_first / _citation.page_last |
|---|
| 改定 1.3 | 2023年10月11日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  分子置換 / 解像度: 1.6 Å
分子置換 / 解像度: 1.6 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Cell Chem Biol / 年: 2020
ジャーナル: Cell Chem Biol / 年: 2020 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6v2s.cif.gz
6v2s.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6v2s.ent.gz
pdb6v2s.ent.gz PDB形式
PDB形式 6v2s.json.gz
6v2s.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6v2s_validation.pdf.gz
6v2s_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6v2s_full_validation.pdf.gz
6v2s_full_validation.pdf.gz 6v2s_validation.xml.gz
6v2s_validation.xml.gz 6v2s_validation.cif.gz
6v2s_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/v2/6v2s
https://data.pdbj.org/pub/pdb/validation_reports/v2/6v2s ftp://data.pdbj.org/pub/pdb/validation_reports/v2/6v2s
ftp://data.pdbj.org/pub/pdb/validation_reports/v2/6v2s リンク
リンク 集合体
集合体


 要素
要素 Homo sapiens (ヒト) / 遺伝子: MPHOSPH8, MPP8 / プラスミド: pET28-MHL / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: MPHOSPH8, MPP8 / プラスミド: pET28-MHL / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU FR-E / 波長: 1.5418 Å
回転陽極 / タイプ: RIGAKU FR-E / 波長: 1.5418 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj






